Xromatin

Xromatin — Xromatin, eukariotik hüceyrələrdə olan bir DNT, protein və RNT kompleksidir. Əsas funksiyası uzun DNT molekullarını daha kompakt, daha sıx strukturlara qablaşdırmaqdır[1]. Bu, tellərin dolaşmasına mane olur və hüceyrə bölünməsi zamanı DNT-nin möhkəmləndirilməsində, DNT zədələnməsinin qarşısının alınmasında və gen köçrülməsinin və DNT replikasiyasının tənzimlənməsində mühüm rol oynayır. Mitoz və meyoz zamanı xromatin anafazda xromosomların düzgün quruluşunu asanlaşdırır; bu mərhələdə görünən xromosomların xarakterik formaları, DNT-nin yüksək qatılaşdırılmış xromatinə bükülməsinin nəticəsidir[2].
Xromatinin əsas protein komponentləri, DNT-yə bağlanan və iplərin sarıldığı "lövbər" kimi fəaliyyət göstərən histonlardır. Ümumiyyətlə, xromatin təşkilatının üç səviyyəsi vardır[3]:
- DNT histon zülallarını əhatə edir, nükleosomlar və meydana gətirir (xromatin).
- Birdən çox histon, ən kompakt formada (heteroxromatin) nükleosom massivlərindən ibarət olan 30 nanometrlik bir lifə bükülür.
- 30 nm lifin daha yüksək səviyyəli DNT-nin üst-üstə sarılması metafaz xromosomunu (mitoz və mayoz zamanı) əmələ gətirir.
Lakin bir çox orqanizm bu təşkilati qanunauyğunluqları izləmir. Məsələn, quşların sperma və eritrositlərində əksər eukarotik hüceyrələrə nisbətən daha sıx dolu xromatin var və tripanosomatoid protozoa xromatinlərini görünən xromosomlara ümumiyyətlə qatışdırmır. Prokarotik hüceyrələr DNT-lərini təşkil etmək üçün tamamilə fərqli strukturlara malikdirlər (prokarotik xromosomun ekvivalenti genofor adlanır və nükleoid bölgəsində yerləşir)[4].
Xromatin şəbəkəsinin ümumi quruluşu əlavə olaraq hüceyrə dövrünün mərhələsindən asılıdır. İnterfaza zamanı xromatin, DNT-ni köçürən və təkrarlayan RNT və DNT polimerazlarına daxil olmaq üçün struktur olaraq boşdur. Interfaza zamanı xromatinin lokal quruluşu DNT-də mövcud olan xüsusi genlərdən asılıdır. Aktiv olaraq köçürülən ("açılmış") DNT-lərin bölgələri daha az sıx və euxromatin olaraq bilinən bir quruluşda RNT polimerazları ilə yaxından əlaqələndirilir, hərəkətsiz genləri olan ("söndürülmüş") bölgələr isə ümumiyyətlə daha sıxlaşır və heteroxromatindəki struktur zülallarla əlaqələndirilir. Metilasyon və asetilasyon yolu ilə struktur xromatin zülallarının epigenetik modifikasiyası da yerli kromatin quruluşunu və dolayısı ilə gen ifadəsini dəyişdirir. Xromatin şəbəkələrinin quruluşu hal-hazırda yaxşı öyrənilməyib və molekulyar biologiyada aktiv tədqiqat sahəsi olaraq qalır.
Xromatinin dinamik quruluşu və iyerarxiyası
[redaktə | vikimətni redaktə et]
Xromatin hüceyrə dövrü ərzində müxtəlif struktur dəyişikliklərinə məruz qalır. Histon zülalları xromatinin əsas qablaşdırıcıları və təşkilatçılarıdır və xromatin qablaşdırmasını dəyişdirmək üçün müxtəlif post-translational modifikasiyalarla dəyişdirilə bilər (histon modifikasiyası). Dəyişikliklərin əksəriyyəti histonların quyruqlarında olur. Xromatinin mövcudluğu və sıxılma baxımından nəticələri həm dəyişdirilmiş amin turşusundan, həm də modifikasiya növündən asılıdır. Məsələn, histonların asetilasiyası boşalma və replikasiya və transkripsiya üçün xromatinin mövcudluğunun artmasına səbəb olur. Lisin trimetilasyonu ya artan transkripsiya aktivliyinə (histon H3 lizin 4 trimetilasyonu), ya da transkripsiyal repressiyaya və xromatinin sıxılmasına (histon H3 lizin 9 və ya 27-nin trimetilasyonu) səbəb ola bilər. Bir neçə tədqiqat fərqli dəyişikliklərin eyni vaxtda baş verə biləcəyini göstərdi. Məsələn, erkən məməlilərin inkişafında ikitərəfli bir quruluşun (histon H3-də lizin 4 və 27 trimetilasyonu ilə) iştirak etdiyi irəli sürülmüşdür.
Polikom qrup zülalları, kromatin quruluşunu modulyasiya edərək gen tənzimlənməsində rol oynayır[5].
DNT quruluşu
[redaktə | vikimətni redaktə et]
Təbiətdə DNT üç quruluş meydana gətirə bilər: A-, B- və Z-DNT. A- və B-DNT çox oxşardır, sağ əlli zəncirlar əmələ gətirir, Z-DNT isə ziqzaq fosfat onurğa sütununa sahib bir sol spiraldir. B-və Z-DNT arasındakı bağlama xüsusiyyətlərinə görə Z-DNT-nin xromatin quruluşunda və transkripsiyada xüsusi rol oynadığına inanılır[6].
B və Z-DNA qovşağında bir baza cütü normal bağlanmadan qopur. İkiqat rol oynayırlar: bir çox zülalın tanınma yeri və RNT polimeraz və ya nükleosomların bağlanmasından bükülmə stresini qəbul edən.
Bir ip üzərində nükleosomlar
[redaktə | vikimətni redaktə et]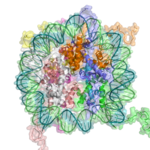
Xromatinin əsas təkrarlanan elementi, yeri həlldəki saf DNT-dən daha qısa olan birləşdirici DNT hissələri ilə bir-birinə bağlı olan nükleosomdur[7].
Nüvə histonlarına əlavə olaraq, nükleosomdakı DNT zolağının çıxışı girişi ilə əlaqə quran bir bağlayıcı histon H1 var. Nükleosomun nüvə hissəciyi, histon H1 ilə birlikdə xromosom olaraq bilinir. Bağlayıcı DNT-nin tərkibində təxminən 20 ilə 60 baza cütü olan nukleozomlar, fizioloji olmayan şərtlər altında hər tel üçün təxminən 10 nm muncuq əmələ gətirə bilər[8].
Nükleozomlar, ümumi DNT paketindəki funksiyalarının tələb etdiyi kimi DNT-ni spesifik olaraq birləşdirir. Bununla birlikdə, nükleosomların yerini təyin edən DNT ardıcıllığı üçün böyük üstünlüklər var. Bu, ilk növbədə fərqli DNT ardıcıllığının fərqli fiziki xüsusiyyətlərindən qaynaqlanır: məsələn, adenin (A) və timin (T) daxili kiçik yivlərdə daha müsbət şəkildə sıxılır[9]. Bu, nükleosomların əsasən hər 10 baza cütü (DNT zəncir təkrarı) ətrafında bir vəziyyətdə bağlana biləcəyi deməkdir.
30 nanometrlik kromatin lifi
[redaktə | vikimətni redaktə et]
H1 əlavə edildikdə, iplikdəki boncukların quruluşu, öz növbəsində, 30 nm lif və ya ip olaraq bilinən, 30 nm diametrli bir zəncir bir quruluşa çevrilir. Xromatin lifinin hüceyrədəki dəqiq quruluşu təfərrüatlı bilinmir[10].
Bu səviyyəli xromatin quruluşunun, əsasən transkripsiyasına görə səssiz genləri ehtiva edən bir heteroxromatinin bir forması olduğuna inanılır. Elektron mikroskopik tədqiqatlar göstərir ki, 30 nm lif çox dinamikdir, beləliklə transkripsiyada iştirak edən RNT polimerazından keçərkən 10 nm muncuq ipi lifli quruluşa çevrilir[11].

Mövcud modellər ümumiyyətlə nükleosomların lif oxuna dik olduğunu və bağlayıcı histonların içəridə yerləşdiyini fərz edirlər. Sabit 30 nm lif DNT boyunca nukleosomların nizamlı düzülüşünə əsaslanır. Bağlayıcı DNT əyilməyə və dönməyə nisbətən davamlıdır. Bu, bağlayıcı DNT-nin uzunluğunu lif sabitliyi üçün kritik hala gətirir, nükleosomların DNT-də lazımsız stres olmadan fırlanaraq istənilən istiqamətə qatlanmasına imkan verən uzunluqlara ayrılmasını tələb edir. Bu baxımdan bağlayıcı DNT-nin fərqli uzunluqları, xromatin lifinin fərqli qatlanan topologiyalarına səbəb olmalıdır[12]. Yenidən qurulmuş liflərin elektron mikroskopiya şəkillərinə əsaslanan son nəzəri işlər bu nöqteyi-nəzərdən dəstəkləyir .
Xromatinin hüceyrə nüvəsindəki məkan təşkili
[redaktə | vikimətni redaktə et]Xromatinin nüvənin içində yerləşməsi təsadüfi deyil — müəyyən ərazilərdə xromatinin müəyyən sahələrinə rast gəlmək olar. Ərazilər, məsələn, protein kompleksləri ilə bir-birinə bağlanan lamino ilə əlaqəli sahələr (LAD) və topoloji cəhətdən əlaqəli sahələrdir (TAD). Hal-hazırda Strings & Binders Switch (SBS) modeli və Dynamic Loop (DL) modeli kimi polimer modelləri nüvə içərisində kromatin qatlanmasını təsvir etmək üçün istifadə olunur[13] .
Hüceyrə dövrünə bağlı struktur təşkilatı
[redaktə | vikimətni redaktə et]

- İnterfaza: Mitotik interfaza zamanı xromatin quruluşu, DNT nüvəsində kondensasiya edilərkən, DNT transkripsiyasına və DNT təmir faktorlarına asanlıqla daxil olmaq üçün optimize edilmişdir. Quruluş, DNT-yə lazımi girişdən asılı olaraq dəyişir. RNT polimeraza müntəzəm giriş tələb edən genlər, euxromatin tərəfindən təmin edilən daha yüngül bir quruluşa ehtiyac duyurlar.
- Metafaza: Xromatinin metafaz quruluşu interfazdan çox fərqlidir. Kariotiplərdə görülən klassik xromosom quruluşunu meydana gətirən fiziki güc və idarəolunma qabiliyyəti üçün müəyyən edilmişdir. Kondensasiya olunmuş kromatinin quruluşunun zülalların mərkəzi bir aparılan 30 nm lifdən ibarət döngələrdən ibarət olduğuna inanılır. Lakin kifayət qədər xarakterizə olunmur[14]. Xromosom skletləri kompakt xromosomlarda xromatinin tutulmasında mühüm rol oynayır. 30 nm quruluşun ilmələri daha yüksək düzəlişlərdə çərçivə ilə daha sıxlaşdırılır. Xromatinin fiziki gücü, bölünmənin bu mərhələsində qızı xromosomların ayrılması zamanı DNT-də kəsilmə zədələnməsinin qarşısını almaq üçün vacibdir. Gücünü artırmaq üçün xromatin tərkibi sentromeraya yaxınlaşdıqda, ilk növbədə alternativ histon H1 analogları vasitəsi ilə dəyişir. Mitoz zamanı, xromatinin çox hissəsi sıx şəkildə sıxılmış olsa da, o qədər də sıx olmayan kiçik sahələr var. Bu bölgələr tez-tez xromatin əmələ gəlməsindən əvvəl bu hüceyrə tipində aktiv olan genlərin təbliğat bölgələrinə uyğundur. Bu sahələrin sıxılmamasına "yaddaş" ın mitoza girmədən əvvəl genlərin aktiv olduğu qızı hüceyrələrə köçürülməsi üçün vacib sayılan epigenetik bir mexanizm olan anlage deyilir. Bu işarələmə mexanizmi bu yaddaşın köçürülməsinə kömək etmək üçün lazımdır, çünki transkripsiya mitoz zamanı dayanır[15].
Xromatin və transkripsiya partlayışları
[redaktə | vikimətni redaktə et]Xromatin və fermentlər ilə qarşılıqlı əlaqəsi araşdırılmış və bunun gen ekspresyonunda vacib bir amil olduğu qənaətinə gəlinmişdir. Rockefeller Universitetinin professoru Vincent G. Alfrey, RNT sintezinin histon asetilasyonla əlaqəli olduğunu bildirdi. Histonların uclarına yapışmış amin turşusu lizin müsbət yüklənmişdir. Bu quyruqların asetilasiyası, kromatinin uclarını neytral hala gətirərək DNT-yə giriş imkanı verir[16].
Kromatin dekondensasiya edildikdə, DNT molekulyar mexanizmlər tərəfindən nüfuz etməyə açıqdır. Açıq və qapalı xromatin arasındakı dalğalanmalar transkripsiyanı kəsə və ya transkripsiyanı poza bilər. Transkripsiya faktorları komplekslərinin xromatinlə birləşməsi və ayrılması kimi digər amillərin də iştirakı ehtimal olunur[17] . Bu fenomen, transkripsiyanın sadə ehtimal modellərindən fərqli olaraq, izogen populyasiyaların hüceyrələri arasında gen ifadəsinin yüksək dəyişkənliyini izah edə bilər.
Alternativ xromatin təşkilatları
[redaktə | vikimətni redaktə et]Çoxhüceyrəli heyvanlarda spermiogenez zamanı spermatid xromatin daha aralı, genişlənmiş, demək olar ki, kristal quruluşa çevrilir. Bu proses transkripsiyanın ləğvi ilə əlaqələndirilir və nüvə zülallarının mübadiləsini əhatə edir. Histonlar daha çox yerdəyişir və yerinə protaminlər (argininlə zəngin kiçik zülallar) qoyulur. Histonları olmayan maya bölgələrində transkripsiyadan sonra çox kövrək olacağı güman edilir; HMG qutusu zülalı olan HMO1, nükleosomsuz kromatinin sabitləşməsinə kömək edir[18].
Xromatin və DNT bərpa
[redaktə | vikimətni redaktə et]Eukariotik DNT-nin xromatinə qablaşdırılması, fermentlərin təsir sahələrinə cəlb edilməsini tələb edən bütün DNT əsaslı proseslər üçün bir maneədir[19] . Hüceyrə DNT təmirinin kritik prosesini həll etmək üçün kromatinin yenidən qurulması lazımdır. Eukariotlarda ATP-yə bağlı xromatin yenidən qurma kompleksləri və histonu dəyişdirən fermentlər bu yenidənqurma prosesi üçün istifadə olunan iki əsas amildir.
DNT zədələnmə yerində xromatin rahatlaması sürətlə baş verir[20]. Bu proses, DNT-nin bir saniyədən az bir müddətdə zədələndiyi zaman görünməyə başlayan PARP1 zülalı tərəfindən başlanır, zədələnmədən sonra 1,6 saniyə ərzində maksimum yığılma yarısıdır. Yenidənqurma kromatin Alc1 daha sonra PARP1 məhsuluna sürətlə yapışır və zədələnmədən sonra 10 saniyə ərzində DNT zədələnməsinə çatır. Alc1 təsiri səbəbindən maksimum xromatin rahatlamasının təxminən yarısı 10 saniyədən sonra baş verir . Bu, MRE11 DNA təmir fermentinin 13 saniyə ərzində DNA təmirinə başlamaq üçün istifadə edilməsinə imkan verir[21].
2H2AX, fosforillənmiş H2AX forması, DNT zədələnməsindən sonra xromatinin dekondensasiyasına səbəb olan ilk mərhələlərdə də iştirak edir. Histon H2AX variantı insan kromatinindəki H2A histonlarının təxminən 10% -ni təşkil edir. γH2AX (serin 139 tərəfindən fosforillənmiş H2AX) hüceyrələrin şüalanmasından 20 saniyə sonra (ikiqat zəncirli DNT qırılmasının əmələ gəlməsi ilə) aşkar edilə bilər və maksimum γH2AX yığılmasının yarısı bir dəqiqədə baş verir. Fosforlaşdırılmış γH2AX olan xromatinin ölçüsü, ikiqat telli DNT qırılmasında təxminən iki milyon baz cütüdür. Öz-özlüyündə, γH2AX xromatin dekondensasiyasına səbəb olmur, lakin şüalandıqdan sonra 30 saniyə ərzində RNF8 zülalı γH2AX ilə əlaqəli olaraq aşkar edilə bilər[22]. RNF8, bir nukleosom yenidən qurma komponenti və NuRD deasetilaz kompleksi ilə sonrakı qarşılıqlı əlaqəsi sayəsində geniş kromatin dekondensasiyası təmin edir .
DNT zədələnməsindən sonra rahatlandıqdan sonra DNT-nin düzəldilməsindən sonra xromatin təxminən 20 dəqiqə ərzində zərərdən əvvəl səviyyəyə yaxın bir sıxılma vəziyyətinə gətirilir[23].
Xromatin tədqiqat metodları
[redaktə | vikimətni redaktə et]Genom boyu xromatin vəziyyətlərini təyin etmək üçün müxtəlif histon modifikasiyalarına qarşı yönəldilmiş ChIP-seq (xromatin immunoprecipitasiya ardıcıllığı) istifadə edilə bilər. Müxtəlif dəyişikliklər fərqli xromatin halları ilə əlaqələndirilmişdir.
- DNase-seq (DNase I hipersensitiv bölgələrin sıralanması) genomdakı açıq və ya əlçatan bölgələri xəritələşdirmək üçün genomdakı əlçatan bölgələrin DNase I fermentinə həssaslığından istifadə edir[24].
- FAIRE-seq (tənzimləyici elementlərin formaldehid dəstəyi ilə sıralanması), nükleosomları olmayan bölgələri genomdan çıxarmaq üçün iki fazlı ayırma texnikasında zülalla əlaqəli DNT-nin kimyəvi xüsusiyyətlərindən istifadə edir[25].
- ATAC-seq (Transpozisiya Mövcud Kromatin Sıralaması Analizi) (sintetik) transpozonları genomun əlçatan bölgələrinə inteqrasiya etmək üçün Tn5 transpozazından istifadə edir, beləliklə genomdakı nükleosomların və transkripsiya amillərinin vurğulanır[26][27].
- DNT izləri, zülallarla əlaqəli DNT-nin müəyyənləşdirilməsinə yönəlmiş bir texnikadır. Genomun zülallarla əlaqəli bölgələrini müəyyən etmək üçün jel elektroforezi ilə birlikdə etiketləmə və parçalanma istifadə edir.
- MNase-seq (mikrookokal nukleaz ardıcıllığı), genom boyu nükleosomların yerini tapmaq üçün bir mikrookokal nukleaz fermentindən istifadə edir.
- Xromosom konformasiyasının tutulması nüvədəki kromatinin məkan təşkilatını təyin edir və fiziki olaraq qarşılıqlı təsir göstərən genomik yerləri göstərir.
- MACC profilinin hazırlanması (Mikrokokal Nükleazın Mövcudluğu Profili), kromatinin mövcudluğunu müəyyənləşdirmək və genomun həm açıq, həm də qapalı bölgələrində nükleosomları və histon olmayan DNT-bağlayıcı zülalları xəritələşdirmək üçün mikrokokal nuklaz tərəfindən kromatin həzminin bir sıra titrlənməsindən istifadə edir[28].
Xromatin və düyünlər
[redaktə | vikimətni redaktə et]Dekondensasiya olunmuş interfazalı xromosomların mahiyyətcə işarəsiz qalması bir sirr idi. Təbii ümid budur ki, ikiqat telli DNT hissələrinin bir-birindən keçməsinə imkan verən tip II DNT topoizomerazları olduqda, bütün xromosomlar topoloji tarazlıq vəziyyətinə çatmalıdır. Xromosomal ərazilər əmələ gətirən çox sıxlıqda olan fazalararası xromosomlarda topoloji tarazlıq yüksək düyünlü xromatin liflərinin əmələ gəlməsinə səbəb olmalıdır[29]. Bununla birlikdə, xromosomların (3C) konformasiyasını tutma üsulları, interfaazalı xromosomlarda genom məsafəsi ilə təmasların məhv edilməsinin qlobusun qırışmış vəziyyəti ilə praktik olaraq eynisini təşkil etdiyini, uzun polimerlər meydana gəlmədən yoğuşduqda meydana gəldiyini göstərir. hər hansı bir qovşaq. Düyünlərin çox sıx olduğu kromatindən çıxarılması, sistemi topoloji tarazlıqdan çıxarmaq üçün yalnız enerji verməməli, həm də topoizomerazın vasitəçiliyi ilə parçalanmaları düyünlər əvəzinə düyünlərin effektiv şəkildə ayrılması üçün aktiv bir proses tələb edir. düyünləri daha da mürəkkəbləşdirir. Xromatin ilmə ekstruziyası prosesinin interfazalı xromosomlarda xromatin liflərinin aktiv birləşməsi üçün ideal olduğu göstərilmişdir[30]. DNT kondensasiyası, in vitro və ya in vivo şəkildə DNA molekullarının sıxlaşma prosesinə aiddir. [1] DNT qablaşdırma mexanizminin təfərrüatları, canlı sistemlərdə gen tənzimlənməsi prosesində işləməsi üçün vacibdir. Kondensasiya olunmuş DNT tez-tez klassik seyreltilmiş məhlul konsepsiyasından proqnozlaşdırıla bilməyən inanılmaz xüsusiyyətlərə malikdir. Beləliklə, in vitro DNT kondensasiyası fizika, biyokimya və biologiyadakı bir çox proses üçün nümunə sistemdir. [2] Bundan əlavə, DNT kondensasiyasının tibb və biotexnologiyada bir çox potensial tətbiqi vardır. [bir]
DNT-nin diametri təxminən 2 nm-dir və genişlənmiş tək bir molekulun uzunluğu orqanizmə görə bir neçə on santimetrə çata bilər. DNT cüt sarmalının bir çox xüsusiyyəti şəkər-fosfat onurğa sütununun mexaniki xüsusiyyətləri, fosfatlar arasındakı elektrostatik itələmə (DNT ikiqat sarmalın hər 0,17 nm üçün orta hesabla bir elementar mənfi yük daşıyır), üstəlik hər bir fərdi ipin əsasları ilə iplik-iplik qarşılıqlı əlaqələri. DNT ən sərt təbii polimerlərdən biridir, eyni zamanda ən uzun molekullardan biridir. Bu o deməkdir ki, uzun məsafələrdə DNT-yə çevik bir ip, qısa bir miqyasda isə sərt bir çubuq kimi baxıla bilər. Bir bağ hortumu kimi, təsadüfi olaraq paketdən çıxarılan DNT, sifariş verildiyindən daha çox həcm alır. Riyazi olaraq, üç ölçülü fəzada təsadüfi səpələnən qarşılıqlı əlaqəsi olmayan çevik bir zəncir üçün ucdan uca məsafə polimer uzunluğunun kvadrat kökü kimi ölçülür. DNA kimi həqiqi polimerlər üçün bu, çox kobud bir təxmindir; vacib olan in vivo DNA üçün mövcud yerin məhluldakı sərbəst diffuziya vəziyyətində tutacağı yerdən qat-qat az olmasıdır. Həcm məhdudiyyətlərinin öhdəsindən gəlmək üçün DNT ionlar və digər molekullarla uyğun həll şəraitində özünü qablaşdıra bilər. Ümumiyyətlə DNT kondensasiyası "genişlənmiş DNT zəncirlərinin yalnız bir və ya bir neçə molekulu ehtiva edən kompakt sifarişli hissəciklərə çevrilməsi" olaraq təyin edilir [3]. Bu tərif bir çox in vitro vəziyyətə aiddir və eyni zamanda bakteriyalardakı DNT kondensasiyasının "mövcud həcmin bir hissəsini tutan nisbətən konsentrasiyalı, kompakt bir vəziyyətin qəbul edilməsi" kimi tərifinə yaxındır [4]. Ökaryotlarda DNT-nin ölçüsü və iştirak edən digər oyunçuların sayı daha böyükdür və DNT molekulu, bir çox DNT qablaşdırma səviyyəsindən yalnız birincisi olan milyonlarla nizamlanmış nükleoprotein hissəciklərini, nükleosomları meydana gətirir. [bir]
Xromatin: alternativ tərifi
[redaktə | vikimətni redaktə et]Valter Fleminq tərəfindən hazırlanan bu terminin bir neçə mənası var:
- Sadə və qısa tərif: xromatin, DNT makromolekullarının və protein (və RNT) makromolekullarının makromolekulyar kompleksidir. Zülallar DNT-ni qablaşdırır və sifariş edir və hüceyrə nüvəsindəki funksiyasını idarə edir[31]
- Biokimyaların iş tərifi: xromatin, eukariotların parçalanmış interfaz nüvələrindən çıxarılan bir DNT protein RNT kompleksidir. Nüvədə mövcud olan bir çox maddənin hansının çıxarılan materialın bir hissəsini təşkil edəcəyi qismən hər tədqiqatçıın istifadə etdiyi metoddan asılıdır. Üstəlik, xromatinin tərkibi və xüsusiyyətləri, müəyyən bir hüceyrə tipinin inkişafı zamanı və hüceyrə dövrünün müxtəlif mərhələlərində bir hüceyrə növündən digərinə dəyişir[31]
- DNA + histon = Xromatin tərifi: Hüceyrənin nüvəsindəki DNT cüt zənciri, histonlar adlanan xüsusi zülallarla doludur. Yaranan protein DNT kompleksinə xromatin deyilir. Xromatinin əsas struktur vahidi nükleosomdur[32].
Birinci tərif, molekulu yoğuran hər hansı bir DNT ilə əlaqələndirən zülallardan istifadə edərək bakteriyalar və arxeylər kimi həyatın digər sahələrində "xromatin" tərifinə imkan verir. Bu zülallara ümumiyyətlə nukleoidlə əlaqəli zülallar (NAP) deyilir; nümunələrə HU ilə AsnC / LrpC daxildir. Bundan əlavə, bəzi arxeylər həqiqətən homoloji olan eukarotik histonlardan zülallardan nükleosomlar istehsal edirlər.
İstinadlar
[redaktə | vikimətni redaktə et]- ↑ Monday, Tanmoy. "Characterization of the RNA content of chromatin". Genome Res. 20 (7). July 2010: 899–907. doi:10.1101/gr.103473.109. PMC 2892091. PMID 20404130.
- ↑ Hansen, Jeffrey. "Human mitotic chromosome structure: what happened to the 30-nm fibre?". The EMBO Journal. 31 (7). March 2012: 1621–1623. doi:10.1038/emboj.2012.66. PMC 3321215. PMID 22415369.
- ↑ Dame, R.T. "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Molecular Microbiology. 56 (4). May 2005: 858–870. doi:10.1111/j.1365-2958.2005.04598.x. PMID 15853876.
- ↑ Bernstein BE, Mikkelsen TS, Xie X, Kamal M, Huebert DJ, Cuff J, Fry B, Meissner A, Wernig M, Plath K, Jaenisch R, Wagschal A, Feil R, Schreiber SL, Lander ES. "A bivalent chromatin structure marks key developmental genes in embryonic stem cells". Cell. 125 (2). April 2006: 315–26. doi:10.1016/j.cell.2006.02.041. ISSN 0092-8674. PMID 16630819.
- ↑ Portoso M, Cavalli G. The Role of RNAi and Noncoding RNAs in Polycomb Mediated Control of Gene Expression and Genomic Programming // RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Caister Academic Press. 2008. ISBN 978-1-904455-25-7. 2012-01-02 tarixində arxivləşdirilib. İstifadə tarixi: 2021-06-17.
- ↑ Annunziato, Anthony T. "DNA Packaging: Nucleosomes and Chromatin". Scitable. Nature Education. 2022-07-31 tarixində arxivləşdirilib. İstifadə tarixi: 2015-10-29.
- ↑ Robinson DJ; Fairall L; Huynh VA; Rhodes D. "EM measurements define the dimensions of the "30-nm" chromatin fiber: Evidence for a compact, interdigitated structure". Proceedings of the National Academy of Sciences of the United States of America. 103 (17). April 2006: 6506–11. Bibcode:2006PNAS..103.6506R. doi:10.1073/pnas.0601212103. PMC 1436021. PMID 16617109.
- ↑ Wong H, Victor JM, Mozziconacci J. Chen P (redaktor). "An All-Atom Model of the Chromatin Fiber Containing Linker Histones Reveals a Versatile Structure Tuned by the Nucleosomal Repeat Length". PLoS ONE. 2 (9). September 2007: e877. Bibcode:2007PLoSO...2..877W. doi:10.1371/journal.pone.0000877. PMC 1963316. PMID 17849006. Şablon:Open access
- ↑ Nicodemi M, Pombo A. "Models of chromosome structure" (PDF). Curr. Opin. Cell Biol. 28. June 2014: 90–5. doi:10.1016/j.ceb.2014.04.004. PMID 24804566. 2022-04-12 tarixində arxivləşdirilib (PDF). İstifadə tarixi: 2021-06-17.
- ↑ Nicodemi M, Panning B, Prisco A. "A thermodynamic switch for chromosome colocalization". Genetics. 179 (1). May 2008: 717–21. arXiv:0809.4788. doi:10.1534/genetics.107.083154. PMC 2390650. PMID 18493085.
- ↑ Bohn M, Heermann DW. "Diffusion-driven looping provides a consistent framework for chromatin organization". PLOS ONE. 5 (8). 2010: e12218. Bibcode:2010PLoSO...512218B. doi:10.1371/journal.pone.0012218. PMC 2928267. PMID 20811620.
- ↑ Lodish, Harvey F. Molecular Cell Biology (8th). New York: W. H. Freeman and Company. 2016. səh. 339. ISBN 978-1-4641-8339-3.
- ↑ Poonperm, R; Takata, H; Hamano, T; Matsuda, A; Uchiyama, S; Hiraoka, Y; Fukui, K. "Chromosome Scaffold is a Double-Stranded Assembly of Scaffold Proteins". Scientific Reports. 5. 1 July 2015: 11916. doi:10.1038/srep11916. PMC 4487240. PMID 26132639.
- ↑ Xing H, Vanderford NL, Sarge KD. "The TBP-PP2A mitotic complex bookmarks genes by preventing condensin action". Nat. Cell Biol. 10 (11). November 2008: 1318–23. doi:10.1038/ncb1790. PMC 2577711. PMID 18931662.
- ↑ Allfrey VG, Faulkner R, Mirsky AE. "Acetylation and Methylation of Histones and Their Possible Role in the Regulation of RNA Synthesis". Proc. Natl. Acad. Sci. U.S.A. 51 (5). May 1964: 786–94. Bibcode:1964PNAS...51..786A. doi:10.1073/pnas.51.5.786. PMC 300163. PMID 14172992.
- ↑ Kaochar S, Tu BP. "Gatekeepers of chromatin: Small metabolites elicit big changes in gene expression". Trends Biochem. Sci. 37 (11). November 2012: 477–83. doi:10.1016/j.tibs.2012.07.008. PMC 3482309. PMID 22944281.
- ↑ De Vries M, Ramos L, Housein Z, De Boer P. "Chromatin remodelling initiation during human spermiogenesis". Biol Open. 1 (5). May 2012: 446–57. doi:10.1242/bio.2012844. PMC 3507207. PMID 23213436.
- ↑ Murugesapillai D, McCauley MJ, Maher LJ, Williams MC. "Single-molecule studies of high-mobility group B architectural DNA bending proteins". Biophysical Reviews. 9 (1). February 2017: 17–40. doi:10.1007/s12551-016-0236-4. PMC 5331113. PMID 28303166.
- ↑ Liu B, Yip RK, Zhou Z. "Chromatin remodeling, DNA damage repair and aging". Curr. Genomics. 13 (7). 2012: 533–47. doi:10.2174/138920212803251373. PMC 3468886. PMID 23633913.
- ↑ Sellou H, Lebeaupin T, Chapuis C, Smith R, Hegele A, Singh HR, Kozlowski M, Bultmann S, Ladurner AG, Timinszky G, Huet S. "The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage". Mol. Biol. Cell. 27 (24). 2016: 3791–3799. doi:10.1091/mbc.E16-05-0269. PMC 5170603. PMID 27733626.
- ↑ Haince JF, McDonald D, Rodrigue A, Déry U, Masson JY, Hendzel MJ, Poirier GG. "PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites". J. Biol. Chem. 283 (2). 2008: 1197–208. doi:10.1074/jbc.M706734200. PMID 18025084.
- ↑ Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM. "DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139". J. Biol. Chem. 273 (10). 1998: 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ↑ Mailand N, Bekker-Jensen S, Faustrup H, Melander F, Bartek J, Lukas C, Lukas J. "RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins". Cell. 131 (5). 2007: 887–900. doi:10.1016/j.cell.2007.09.040. PMID 18001824.
- ↑ Luijsterburg MS, Acs K, Ackermann L, Wiegant WW, Bekker-Jensen S, Larsen DH, Khanna KK, van Attikum H, Mailand N, Dantuma NP. "A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure". EMBO J. 31 (11). 2012: 2511–27. doi:10.1038/emboj.2012.104. PMC 3365417. PMID 22531782.
- ↑ Giresi, Paul G.; Kim, Jonghwan; McDaniell, Ryan M.; Iyer, Vishwanath R.; Lieb, Jason D. "FAIRE (Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin". Genome Research. 17 (6). 2007-06-01: 877–885. doi:10.1101/gr.5533506. ISSN 1088-9051. PMC 1891346. PMID 17179217.
- ↑ Galas, D. J.; Schmitz, A. "DNAse footprinting: a simple method for the detection of protein-DNA binding specificity". Nucleic Acids Research. 5 (9). 1978-09-01: 3157–3170. doi:10.1093/nar/5.9.3157. ISSN 0305-1048. PMC 342238. PMID 212715.
- ↑ Cui, Kairong; Zhao, Keji. Genome-wide approaches to determining nucleosome occupancy in metazoans using MNase-Seq. Methods in Molecular Biology. 833. 2012-01-01. 413–419. doi:10.1007/978-1-61779-477-3_24. ISBN 978-1-61779-476-6. ISSN 1940-6029. PMC 3541821. PMID 22183607.
- ↑ Buenrostro, Jason D.; Giresi, Paul G.; Zaba, Lisa C.; Chang, Howard Y.; Greenleaf, William J. "Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position". Nature Methods. 10 (12). 2013-12-01: 1213–1218. doi:10.1038/nmeth.2688. ISSN 1548-7105. PMC 3959825. PMID 24097267.
- ↑ Mieczkowski J, Cook A, Bowman SK, Mueller B, Alver BH, Kundu S, Deaton AM, Urban JA, Larschan E, Park PJ, Kingston RE, Tolstorukov MY. "MNase titration reveals differences between nucleosome occupancy and chromatin accessibility". Nature Communications. 7. 2016-05-06: 11485. Bibcode:2016NatCo...711485M. doi:10.1038/ncomms11485. PMC 4859066. PMID 27151365.
- ↑ Buenrostro, Jason D.; Giresi, Paul G.; Zaba, Lisa C.; Chang, Howard Y.; Greenleaf, William J. "Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position". Nature Methods. 10 (12). 2013-12-01: 1213–1218. doi:10.1038/nmeth.2688. ISSN 1548-7105. PMC 3959825. PMID 24097267.
- ↑ 1 2 Racko D, Benedetti F, Goundaroulis D, Stasiak A. "Chromatin Loop Extrusion and Chromatin Unknotting". Polymers. 10 (10). 2018: 1126–1137. doi:10.3390/polym10101126. PMC 6403842. PMID 30961051.
- ↑ "Thomas Hunt Morgan and His Legacy". Arxivləşdirilib 2017-10-20 at the Wayback Machine Nobelprize.org. 7 Sep 2012