Triptofan sintaza
| Triptofan sintaza | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 } | |||||||||
| Identifikatori | |||||||||
| EC broj | 4.2.1.20 | ||||||||
| CAS broj | 9014-52-2 | ||||||||
| Baze podataka | |||||||||
| IntEnz | IntEnz pregled | ||||||||
| BRENDA | BRENDA unos | ||||||||
| ExPASy | NiceZyme pregled | ||||||||
| KEGG | KEGG unos | ||||||||
| MetaCyc | metabolički put | ||||||||
| PRIAM | profil | ||||||||
| PDB strukture | RCSB PDB PDBj PDBe PDBsum | ||||||||
| Ontologija gena | AmiGO / QuickGO | ||||||||
| |||||||||
Triptofan sintaza ili triptofan sintetaza je enzim koji katalizira dva finalna koraka u biosintezi triptofana.[1] Nađena je kod Eubacteria,[2] Archaebacteria,[3] Protista,[4] Fungi,[5] i biljaka.[6] Međutim, nema je kod životinja.[7][8][9]
Triptofan sintaza nalazi se obično u obliku α2β2 tetramera. Podjedinica α katalizira reverzibilno formiranje indola i gliceraldehid-3-fosfata (G3P) iz indol-3-glicerol fosfata (IGP). Podjedinica β katalizira nepovratnu kondenzaciju indola i serina kada se formira triptofan i piridoksal fosfat (PLP), zavisno od reakcije. Svako alfa aktivno mjesto povezano je sa β aktivnim mjestom dugim 25 angstrema u vidu hidrofobnog kanala koji sadrži enzim. To olakšava širenje formiranog indola na α aktivna mesta direktno na beta aktivna mesta u procesu poznatom kao kanalisanje supstrata.[10] Aktivno mjesto triptofan sintaze je alosterno upareno.[11]
Struktura
[uredi | uredi izvor]Podjedinice: Triptofan sintaze obično postoji kao α-ββ-α kompleks. Podjedinice α i β imaju molarne mase od 27, odnosno 43 kDa. Podjedinica α ima konformaciju TIM barel (TIM-bure), a β ima konformaciju nabora tipa II i vezanje uz aktivno mjesto za monovalentne katione.[12] Njihovo okupljanje u kompleks dovodi do strukturnih promjena u obje podjedinice što rezultira recipročnom aktivacijom. Postoje dva glavna mehanizma za međupodjediničnu komunikaciju. Prvo, u interakciju ulaze COMM domen β-podjedinice i α-petlja2 α-podjedinice. Osim toga, postoji interakcija između αGly181 i βSer178 ostataka.[13] The active sites are regulated allosterically and undergo transitions between open, inactive, and closed, active, states.[11]
Indol-3-glicerol-vežuže mjesto: Vidi sliku 1.
Indol i serin-vežuće mjesto: Vidi sliku 1.
Hidrofobni kanal: Aktivna mjesta α i β odvojena su hidrofobnim kanalom dužine 25 angstrema, koji sadrži enzim koji omogućava širenje indola. Ako kanal ne postoji, indol formiran pri α aktivnom mjestu bi brzo difundirao dalje i izgubio se u ćeliji jer je hidrofoban i lahko prolazi kroz membrane. Kao takav, kanal je od suštinskog značaja za funkciju enzimskog kompleksa.[14]
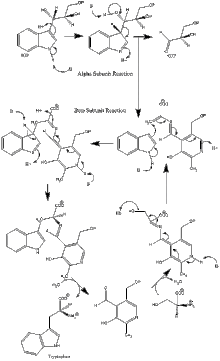
Mehanizam djelovanja
[uredi | uredi izvor]Reakcija α podjedinice: Podjedinica α katalizira formiranje indola i G3P, putem retro-aldolnog cijepanja IGP. Pritom se za αGlu49 i αAsp60 smatra da su direktno uključeni u katalizu kao što je gore prikazano.[10] Stopa ograničavajućeg koraka određena je je izomerizacijom IGP.[15] Vidi sliku 2.
Reakcija β podjedinice: Podjedinica β katalizira reakciju β-premještanja, u kojem se indol i serin kondenziraju i formiraju triptofan u reakciji nastanka PLP. Za βLys87, βGlu109 i βSer377 smatra se da su direktno uključeni u katalizu kao što je prikazano.[10] Ponovo, tačan mehanizam nije konačno utvrđen. Vidi sliku 2! Neto reakcija: Vidi sliku 3.


Biološka funkcija
[uredi | uredi izvor]Triptofan sintaza nalazi se obično u Eubakteria, Archaebacteria, Protista, gljiva i biljaka. Nema je kodd životinja, kao što su ljudi. Triptofan je jedna od standardnih aminokiselina, od ukupno dvadeset i jedne od devet esencijalnih aminokiselina za ljude. Kao takav, triptofan je neophodna komponenta ljudske ishrane.
Medicinski značaj
[uredi | uredi izvor]Budući da ljudi nemaju triptofan sintaze, ovaj enzim je istraživan kao potencijalna meta lijekova.[16] Međutim, smatra se da bakterije imaju alternativne mehanizme za proizvodnju aminokiselina koje mogu učiniti ovaj pristup manje efikasnim. U svakom slučaju, čak i ako lijek samo slabi bakterije, može i dalje biti koristan za sprečavanje bakterija kada su već ugrožene u neprijateljskom okruženju domaćina. Kao takva, inhibicija putem triptofan sintaze zajedno sa ostalim PLP-enzimima u metabolizmu aminokiselina ima potencijal da pomogne u rješavanju zdravstvenih problema.[17]
Inhibicija triptofan syntaze i ostalih enzima PLP grupe u metabolizmu aminokiselina, ukazuje na mogućnost liječenja bolesti kao što su: tuberkuloza[16];
- očne i genitalne infekcije[18];
- kriptosporidioza[16];
Pored toga, triptofan sintaza može poslužiti kao preventivno sredstvo protiv korova, odnosno
Evolucija
[uredi | uredi izvor]Smatra se da je u ranoj u evoluciji došlo do duplikacije gena trpB2. Jedna kopija ušla je u TRP operon kao trpB2i omogućavajući njegovo ispoljavanje sa trpA. TrpB2i formira prolazni kompleks sa TrpA i u procesu neusmjereno aktiviranog TrpA . Drugi primjerak je ostje van kao trpB2o i ispunjava postojeću ulogu ili ima novu, kao što djeluje u spašavanju proteina za indol. TrpB2i evoluirao je u TrpB1, koji formira stalni kompleksa sa trpA rezultirajući u dvosmjernoj aktivaciji. Prednost indolskog spašavanje proteina opada i TrpB gen je izgubljen. Konačno, TrpB1 i TrpA geni su spojeni, što rezultira u formiranju dvofunkcionalnog enzima.[20]
Historijski značaj
[uredi | uredi izvor]Triptofan sintaza bila je prvi od enzima za koje se utvrdilo da imaju dvije katalitske sposobnosti koje su intenzivno proučavane. To je ujedno bio i prvi identifikovni enzim koji koristi supstratno kanalisanje. Kao takav, ovaj enzim je intenzivno proučavan i predmet je od velikog interesa[10]
Također pogledajte
[uredi | uredi izvor]Reference
[uredi | uredi izvor]- ^ Dunn MF, Niks D, Ngo H, Barends TR, Schlichting I (2008). "Tryptophan synthase: the workings of a channeling nanomachine". Trends in Biochemical Sciences. 33 (6): 254–64. doi:10.1016/j.tibs.2008.04.008. PMID 18486479.CS1 održavanje: upotreba parametra authors (link)
- ^ Jablonski P, Jablonski L, Pintado O, Sriranganathan N, Howde C (1996). "Tryptophan synthase: Identification of Pasteurella multocida tryptophan synthase B-subunit by antisera against strain PI059". Microbiology. 142: 115–21. doi:10.1099/13500872-142-1-115. PMID 8581158.CS1 održavanje: upotreba parametra authors (link)
- ^ Lazcano A, Diaz-Villgomez E, Mills T, Oro J (1995). "On the levels of enzymatic substrate specificity: Implications for the early evolution of metabolic pathways". Advances in Space Research. 15 (3): 345–56. doi:10.1016/S0273-1177(99)80106-9. PMID 11539248.CS1 održavanje: upotreba parametra authors (link)
- ^ Anderson I, Watkins R, Samuelson J, Spencer D, Majoros W, Grey M, Loftus B (august 2005). "Gene Discovery in the Acanthamoeba castellanii Genome". Protist. 156 (2): 203–14. doi:10.1016/j.protis.2005.04.001. PMID 16171187.CS1 održavanje: upotreba parametra authors (link)
- ^ ,Ireland C, Peekhaus N, Lu P, Sangari R, Zhang A, Masurekar P, An Z (april 2008). "The tryptophan synthetase gene TRP1 of Nodulisporium sp.: molecular characterization and its relation to nodulisporic acid A production". Appl Microbiol Biotechnol. 79 (3): 451–9. doi:10.1007/s00253-008-1440-3. PMID 18389234.CS1 održavanje: upotreba parametra authors (link)
- ^ Sanjaya, Hsiao PY, Su RC, Ko SS, Tong CG, Yang RY, Chan MT (2008). "Overexpression of Arabidopsis thaliana tryptophan synthase beta 1 (AtTSB1) in Arabidopsis and tomato confers tolerance to cadmium stress". Plant Cell Environ. 31 (8): 1074–85. doi:10.1111/j.1365-3040.2008.01819.x. PMID 18419734.CS1 održavanje: više imena: authors list (link)
- ^ Bajrović K, Jevrić-Čaušević A., Hadžiselimović R., Eds. (2005). Uvod u genetičko inženjerstvo i biotehnologiju. Institut za genetičko inženjerstvo i biotehnologiju (INGEB) Sarajevo. ISBN 9958-9344-1-8.CS1 održavanje: više imena: authors list (link)
- ^ Alberts B.; et al. (2002). Molecular Biology of the Cell, 4th Ed. Garland Science. ISBN 0-8153-4072-9. Eksplicitna upotreba et al. u:
|author=(pomoć) - ^ Eckert SC, Kubler E, Hoffmann B, Braus GH (juni 2000). "The tryptophan synthase-encoding trpB gene of Aspergillus nidulans is regulated by the cross-pathway control system". Mol Gen Genet. 263 (5): 867–76. doi:10.1007/s004380000250. PMID 10905354.CS1 održavanje: upotreba parametra authors (link)
- ^ a b c d Raboni S, Bettati S, Mozzarelli A (april 2009). "Tryptophan synthase: a mine for enzymologists". Cell Mol Life Sci. 66 (14): 2391–403. doi:10.1007/s00018-009-0028-0. PMID 19387555.CS1 održavanje: upotreba parametra authors (link)
- ^ a b Fatmi MQ, Ai R, Chang CA (septembar 2009). "Synergistic regulation and ligand-induced conformational changes of tryptophan synthase". Biochemistry. 48 (41): 9921–31. doi:10.1021/bi901358j. PMID 19764814.CS1 održavanje: upotreba parametra authors (link)
- ^ Grishin NV, Phillips MA, Goldsmith EJ (juli 1995). "Modeling of the spatial structure of ornithine decarboxylases". Protein Sci. 4 (7): 1291–304. doi:10.1002/pro.5560040705. PMC 2143167. PMID 7670372.CS1 održavanje: upotreba parametra authors (link)
- ^ Schneider TR, Gerhardt E, Lee M, Liang PH, Anderson KS, Schlichting I (april 1998). "Loop closure and intersubunit communication in tryptophan synthase". Biochemistry. 37 (16): 5394–406. doi:10.1021/bi9728957. PMID 9548921.CS1 održavanje: upotreba parametra authors (link)
- ^ Huang X, Holden HM, Raushel FM (2001). "Channeling of Substrates and Intermediates in Enzyme-Catalyzes Reactions". Annu Rev Biochem. 70: 149–80. doi:10.1146/annurev.biochem.70.1.149. PMID 11395405. Referenca sadrži prazan nepoznati parametar:
|month=(pomoć)CS1 održavanje: upotreba parametra authors (link) - ^ Anderson KS, Miles EW, Johnson KA (maj 1991). "Serine modulates substrate channeling in tryptophan synthase. A novel intersubunit triggering mechanism". J Biol Chem. 266 (13): 8020–33. PMID 1902468.CS1 održavanje: upotreba parametra authors (link)
- ^ a b c Chaudhary K, Roos DS (septembar 2005). "Protozoan genomics for drug discovery". Nat Biotechnol. 23 (9): 1089–91. doi:10.1038/nbt0905-1089. PMID 16151400.CS1 održavanje: upotreba parametra authors (link)
- ^ Becker D, Selbach M, Rollenhagen C, Ballmaier M, Meyer TF, Mann M, Bumann D (2006). "Robust Salmonella metabolism limits possibilities for new antimicrobials". Nature. 440 (7082): 303–7. doi:10.1038/nature04616. PMID 16541065.CS1 održavanje: upotreba parametra authors (link)
- ^ Caldwell HD, Wood H, Crance D, Baily R (juni 2003). "Polymorphisms in Chlamydia trachomatis tryptophan synthase genes differentiate between genital an ocular isolates". J Clin Invest. 111 (11): 1757–69. doi:10.1172/JCI17993. PMC 156111. PMID 12782678.CS1 održavanje: upotreba parametra authors (link)
- ^ Kulik V, Hartmann E, Weyand M, Frey M, Gierl A, Niks D, Dunn MF, Schlichting I (septembar 2005). "On the structural basis of the catalytic mechanism and the regulation of the α-subunit of tryptophan synthase from Salmonella typhimurium and BXI from maize, two evolutionarily related enzymes". J Mol Biol. 352 (3): 608–20. doi:10.1016/j.jmb.2005.07.014. PMID 16120446.CS1 održavanje: upotreba parametra authors (link)
- ^ Leopoldseder S, Hettwer S, Sterner R (novembar 2006). "Evolution of Multi-Enzyme Complexes: The Case of Tryptophan Synthase". Biochemistry. 45 (47): 14111–9. doi:10.1021/bi061684b. PMID 17115706.CS1 održavanje: upotreba parametra authors (link)