ARN no codificant
Un ARN no codificant (ARNnc) és una molècula d'ARN funcional que no es tradueix en una proteïna. Sinònims emprats menys sovint són els anomenats en anglès npcRNA (non-protein-coding RNA), nmRNA (non-messenger RNA), snmRNA (small non-messenger RNA) i fRNA (functional RNA). El terme small RNA (sRNA) es fa servir comunament per a petits ARNnc de bacteris. La seqüència d'àcid desoxiribonucleic des de la qual és transcrit l'ARNnc com a producte final sol anomenar-se gen d'ARN no codificant. Aquest tipus de molècula es troba, majoritàriament en la forma d'ARN de transferència (ARNt) o ARN ribosòmic (ARNr).
Els gens d'ARN no codificant inclouen ARNs abundants i importants funcionalment, com l'ARN transferent (ARNt) i ARN ribosòmic (ARNr) i altres ARN com els snoARN (ARN petit nucleolar, small nucleolar RNA), miARN (micro ARN), siARN (ARN petit d'interferència, small interfering RNA), piARN (ARN d'interacció amb Piwi, Piwi-interacting RNA) i el ARNlnc (ARNnc llarg, long non coding RNA, lncRNA) que inclou exemples com el Xist i HOTAIR.
El nombre d'ARNnc codificats en el genoma humà és desconegut; no obstant això, recents estudis transcriptòmics i bioinformàtics suggereixen l'existència de milers de ncARNs.[1][2][3][4] Com que la funció de molts dels ncARNs identificats recentment no ha estat confirmada, és possible que molts siguin no funcionals.[5]
Història i descobriment
[modifica]Els àcids nucleics van ser descoberts per primera vegada en 1868 per Friedrich Miescher[6] i pel 1939 ja havien implicat l'ARN a la síntesi de proteïnes.[7] Dues dècades més tard, Francis Crick va predir un component de l'ARN funcional que controlava la traducció; argumentava que l'ARN encaixa millor amb el transcrit d'ARNm que un polipèptid pur.[8]
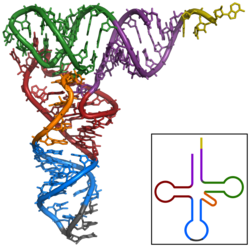
El primer ARN no codificant a ser caracteritzat va ser un ARNt d'alanina trobat al llevat de pastisseria; la seva estructura va ser publicada en 1965.[9] Per a produir una mostra (purificada) d'ARNt d'alanina, Robert W. Holleyet al. va utilitzar 140 kg de llevat de pastisseria comercial per obtenir només 1 g d'ARNtAla pur per a l'anàlisi.[10] L'ARNt de 80 nucleòtids va ser seqüenciat sent primer digerit amb ribonucleasa pancreàtica (produint fragments acabats en citosina o uridina) i després amb ribonucleasa takadiastasa Tl (produint fragments acabats en guanosina). La cromatografia i identificació dels extrems 5' i 3' va ajudar després a ordenar els fragments per establir la seqüència d'ARN.[10] De les tres estructures proposades originalment per a aquest ARNt,[9] l'estructura en forma de trèvol va ser proposada independentment a les següents publicacions.[11][12][13][14] L'estructura secundària en forma de trèvol va ser concretada seguint anàlisis cristal·logràfiques de raigs X duts a terme per dos grups independents de recerca en 1974.[15][16]
L'ARN ribosomal va ser el següent a ser descobert, seguit per l'UARN a principis dels anys 80. Des d'aleshores, el descobriment de nous ARNs no codificants ha continuat amb els snoARNs, Xist, CRISPR i molts més.[17] Addicions recents notables inclouen riboswitches i el miARN, el descobriment del mecanisme de l'ARNi descobert per Craig C. Mello and Andrew Fire (guanyadors el 2006 del Premi Nobel de Medicina o Fisiologia).[18]
Funcions biològiques del ncARN
[modifica]Els ARN no codificants pertanyen a diversos grups i estan implicats en molts processos cel·lulars. Aquests van des dels ncARNs d'importància central que es conserven durant tota o quasi tota la vida cel·lular fins a ncARNs més transitoris, que són específics per a una o unes poques espècies relacionades. Es creu que els ncARNs més conservats són fòssils o relíquies moleculars de LUCA i el món ARN.[19][20][21]

Paper a la traducció
[modifica]
Molts dels ncARNs essencials i abundants conservats estan implicats en la traducció. Les partícules de ribonucleoproteïna anomenades ribosomes són les “fàbriques” on la traducció té lloc a la cèl·lula. El ribosoma conté més del 60% d'ARN ribosomal, format per 3 ncARNs en procariotes i 4 ncARNs en eucariotes. Els ARNs ribosomals catalitzen la traducció de seqüències de nucleòtids a proteïna. Un altre tipus de ncARNs, els ARNs transferents, formen una “molècula adaptadora” entre l'ARNm i les proteïnes. Els snoARNs H/ACA box i C/D box són ncARNs que es troben en archaea i eucariotes i la ARNasa MRP està restringida als eucariotes; ambdós grups de ncARN estan implicats en la maduració de l'ARNr. Els snoARNs guien modificacions covalents d'ARNr, ARNt i snARNs, la ARNasa MRP trenca l'espaiador 1 transcrit intern entre 18S u 5,8S rARNs. El ncARN ubiquitinitzat, ARNasa P, és un parent evolutiu de la ARNasa MRP.[23] L'ARNasa P madura seqüències d'ARNt generant extrems 5' madurs d'ARNt i trencant els elements líders 5' dels tARNs precursors. Un altre RNP ubiquitinitzat anomenat SRP reconeix i transporta noves proteïnes al reticle endoplasmàtic en eucariotes i la membrana plasmàtica en procariotes. En bacteris, l'ARN transferent-missatger (tmARN) és un RNP implicat en rescatar ribosomes estancats, etiquetant polipèptids incomplets i promovent la degradació d'ARNm en mal estat.
ncARN en splicing d'ARN
[modifica]
En eucariotes el spliceosoma realitza les reaccions de splicing, essencials per retirar les seqüències d'Introns. Aquest procés és necessari per a la formació d'ARNm madur. L'spliceosoma és un altre RNP normalment conegut com a snRNP o tri-snRNP. Hi ha dues formes diferents de spliceosoma, la major i la menor. Els components de ncARN del spliceosoma major són U1, U2, U3, U4 i U5. Els components de ncARN del spliceosoma menor són U11, U12, U5, U4atac i U6atac. Un altre grup d'introns pot catalitzar la seva pròpia eliminació dels trànscrits hostes. S'anomenen self-splicing ARNs i es divideixen en dos grups principals, el grup I d'introns catalítics i el grup II d'introns catalítics. Aquests ncARNs catalitzen la seva pròpia escissió des dels precursors d'ARNm, ARNt i ARNr en una gran diversitat d'organismes. En mamífers, s'ha descobert que els snoARNs poden regular l'splicing alternatiu de l'ARNm. Per exemple, l'snoARN HBII-52 regula l'splicing del receptor de serotonina 2C.[24] En nematodes, el SmY ncARN sembla estar implicat en el trans-splicing de l'ARNm.
ncARNs en la regulació genètica
[modifica]L'expressió de molts milers de gens està regulada per ncARNs. Aquesta regulació pot donar-se en trans o cis.
Trans-acting ncARNs
[modifica]En eucariotes majors, els microARNs regulen l'expressió genètica. Un sol miARN pot reduir els nivells d'expressió de centenars de gens. El mecanisme pel qual les molècules de miARN madur actuen és a través de molècules parcialment complementàries a un o més mARNs, generalment en 3'UTRs. La funció principal dels miARNs és inhibir l'expressió genètica. S'ha demostrat que la ncARN ARNasa P també influeix sobre l'expressió genètica. En el nucli humà, l'ARNasa P és necessària per a la transcripció normal i eficient de diversos ncARNs transcrits per l'ARN polimerasa III. Aquests inclouen gens d'ARNt, 5S ARNr, SRP ARN i U6 snARN. L'ARNasa P exerceix el seu paper a la transcripció associant-se amb la Pol III i la cromatina dels gens actius d'ARNt i 5S ARNr.[25] S'ha demostrat que el 7SK ARN, un ncARN metazoal, actua com un regulador negatiu del factor d'elongació P-TEFb de l'ARN polimerasa II, i que aquesta activitat està influïda per vies de resposta a l'estrès. El ncARN bacterià, 6S ARN, s'associa específicament amb l'holoenzim ARN polimerasa, que conté el factor específic sigma 70. Aquesta interacció reprimeix l'expressió d'un promotor dependent de sigma 70 durant la fase estacionària. Un altre ncARN bacteria, l'OxyS ARN, reprimeix la traducció associant-se a seqüències de Shine-Dalgarno i així ocloent la unió dels ribosomes. L'OxyS ARN és produït com a resposta a l'estrès oxidatiu en Escherichia coli. El B2 ARN és un petit trànscrit de ncARN polimerasa III que reprimeix la transcripció d'ARNm en resposta a un xoc de calor en cèl·lules de ratolí. El B2 ARN inhibeix la transcripció associant-se al nucli de la Pol II. A través d'aquesta interacció, el B2 ARN s'uneix a complexos de preiniciació en el promotor i bloqueja la síntesi d'ARN.[26]
Un estudi recent ha mostrat que just l'acte de transcripció de la seqüència de ncARN pot tenir influencia sobre l'expressió genètica. La transcripció a ncARN de l'ARN polimerasa II es necessita per a la remodelació de cromatina en els Schizosaccharomyces pombe. La cromatina adopta progressivament un configuració oberta, així com diverses espècies de ncARN són transcrits.[27]
Cis-acting ncARNs
[modifica]Un nombre de ncARNs estan units als 5'UTRs dels gens codificants de proteïnes i influeixen la seva expressió de diverses maneres. Per exemple, un riboswitch pot unir dírectament una molècula diana petita i aquesta unió afecta l'activitat genètica. Es troben seqüències d'ARN líder a la zona superior del primer gen dels operons biosintetitzadors d'aminoàcids. Aquests elements d'ARN formen una de les dues possibles estructures en regions codificant seqüències peptídiques molt curtes que són riques en l'aminoàcid final produït per l'operó. Una estructura degradadora es forma quan existeix un excés de l'aminoàcid regulador i el moviment ribosòmic sobre el trànscrit líder no s'impedeix. Quan hi ha una deficiència d'ARNt carregat de l'aminoàcid regulador, el ribosoma actiu s'atura i es forma l'estructura no-degradativa. Això permet a l'ARN polimerasa transcriure l'operó. Els ARN líders coneguts són l'operó líder d'histidina, l'operó líder de leucina, l'operó líder de treonina i l'operó líder de triptòfan.
Els IRE (“Iron reponse elements”, elements de resposta al ferro) estan units per IRP (Iron response proteins”, proteïnes de resposta al ferro). Els IRE es troben als UTRs de diferents mARNs els productes dels quals estan involucrats al metabolisme del ferro. Quan la concentració de ferro és baixa, les IRPs uneixen l'IRE ARNm de ferritina per la repressió de la seva traducció.
IRES (“Internal ribosome entry sites”, llocs d'entrada interns dels ribosomes) són una estructura d'ARN que permeten l'inici de la traducció al mig de la seqüència d'ARNm com a part del procés de síntesi proteica.
ncARNs i defensa del genoma
[modifica]Els piARNs (“Piwi-interacting ARNs”, ARN que interaccionen amb el Piwi) expressats a les mostres de mamífers i a cèl·lules somàtiques, formen complexos proteics d'ARN amb proteïnes Piwi. Aquests complexos piARNs estan lligats a la inhibició de la transcripció genètica dels retrotransposons i altres elements genètics de les cèl·lules de la línia germinal, especialment aquelles implicades a l'espermatogènesi. Els CRISPR (“Clustered Regularly Interspaced Short Palindromic Repeats”) són trobats repetides vegades al DNA de molts bacteris. Les repeticions estan separades per espais de longitud similar. S'ha estat demostrat que aquests espais poden derivar d'infeccions víriques i, conseqüentment, poden ajudar a les cèl·lules a protegir-se d'infeccions.
ncARNS i l'estructura cromosòmica
[modifica]La telomerasa és un enzim RNP que afegeix repeticions de seqüències específiques de DNA a regions telomèriques, les quals es troben al final dels cromosomes d'eucariotes. Els telòmers contenen DNA condensat, donant estabilitat als cromosomes. L'enzim és una transcriptasa inversa que duu ARN telomerasa, que és utilitzada per allargar els telòmers, que es fan més curts després de cada cicle de replicació. Xist (“X-inactive-specific transcript”, transcrit inactiu específic de X) és un gen ncARN llarg dels cromosomes X dels mamífers placentaris que actua com un efector de la inactivació del procés de formació de cossos Barr pel cromosoma X. Un ARN amb sentit invers, Tsix, és un regulador negatiu de Xist. Els cromosomes X amb falta d'expressió de Tsix (i amb grans nivell d'expressió del Xist) són inactivats més sovint que els cromosomes normals. Als Drosophilids, que també utilitzen un sistema de determinació de sexe XY, els ARNs roX (ARN del cromosoma X) estan involucrats en la compensació mitjançant dosis.[28] Tant Xist com roX presenten una regulació epigenètica de la transcripció a través de la captació d'enzims modificadors d'histones.
ARN bifuncional
[modifica]Els ARNs bifuncionals són ARNs que tenen dues funcions diferents.[29][30] També són coneguts com a ARNs amb funció dual. La majoria dels ARNs bifuncionals coneguts són mARNs que codifiquen una proteïna i ncARNs. Això no obstant, també hi ha un creixent nombre de ncARNs que se situen en dues categories diferents de ncARN. Per exemple, H/ACA box snoARN i miARN.[31][32]
Dos exemples ben coneguts d'ARNs bifuncionals són el SgrS ARN i el ARNIII. Això no obstant, es coneixen també altres ARNs bifuncionals que existeixen: SRA,[33] VegT ARN,[34][35] Oskar ARN[36] i enod 40.[37]
ncARNs i malalties
[modifica]Proteïnes, mutacions i desequilibris del repertori de ncARNs al cos poden produir una gran varietat de malalties.
Càncer
[modifica]Molts ncARNs mostren uns patrons d'expressió anormals als teixits cancerígens, tenint un paper important dins de l'epigenètica del càncer. Aquests inclouen miARNs,[38] ARNm-like ncARNs llargs,[39][40] GAS5[41] SNORD 50,[42] ARN telomerasa i Y ARNs.[43] Els miARNs estan involucrats en una llarga escala regulatòria de molts gens codificadors de proteïnes;[44][45] els YARN són importants per a la iniciació de la replicació de DNA;[46] les ARN telomerases que funciona com a primer per la telomerasa, un RNP que allarga les regions telomèriques del final del cromosoma. La funció directa del ARNm-like ncARNs llarg és menys clara.
S'ha demostrat que les mutacions de la línia germinal als precursors primaris del miR-16-1 i al miR-15 són més freqüents als pacients amb leucèmia limfoide crònica que a les poblacions control.[47][48]
S'ha suggerit que un SNP estrany (rs11614913) que està damunt la hsa-mir-196a2 està associat al carcinoma pulmonar de cèl·lules petites.[49] De la mateixa forma, un cribat de 17 miARNs que s'ha predit que regulen un nombre de variacions de gens associats al càncer de pit va presentar les variacions al miR17 i miR-30c-1. Aquests pacients no eren portadors de mutacions BRCA1 ni BRCA2, habilitant la possibilitat que el càncer de pit hereditari pot ser causat per variacions d'aquests miARNs.[50]
Síndrome Prader-Willi
[modifica]L'eliminació de les 48 còpies del C/D box snoARN SNORD116 ha estat demostrada com a primera causa de la síndrome de Parder-Willi.[51][52][53] Prader-Willi és un trastorn en el desenvolupament associat a problemes a l'aprenentatge i trastorns alimentaris. SNORD116 té llocs diana potencials entre un nombre de gens codificadors de proteïnes i pot tenir un paper a la regulació alternativa de l'splicing.[54]
Autisme
[modifica]El locus cromosomal que conté el grup genètic SNORD115 snARN ha estat duplicat en aproximadament un 5% dels individus amb característiques autistes.[55] [56] Un ratolí modificat genèticament per a tenir la duplicació del grup SNORD115 manifesta comportament autista.[57]
Hipoplàsia cartílag-cabell
[modifica]S'ha demostrat que mutacions entre ARNses MRP causa hipoplàsia cartílag-cabell, una malaltia associatda a un grup de símptomes tals com baixa estatura, escàs cabell, anormalitats esquelètiques i una supressió del sistema immunitari que és freqüent entre els amish i els finlandesos.[58][59][60] La variant millor caracteritzada és una transició A-G al nucleòtid 70 que està a una regió corbada de dues bases 5' d'un pseudoknot (estructura secundària del DNA) conservat. De totes maneres, moltes altres mutacions entre ARNasa MRP també causen hipoplàsia cartílag-cabell.
Malaltia d'Alzheimer
[modifica]El DNA en sentit oposat BACE1-AS està transcrit des de la banda oposada del BACE1 i una regulació progressiva als pacients amb Alzheimer.[61] BACE1-AS regula l'expressió de BACE1 Mitjançant l'increment de l'estabilitat del BACE1 ARNm i generant BACE1 addicional a causa d'un mecanisme post-traduccional de prealimentació. Pel mateix mecanisme també s'augmenten les concentracions de beta-amieloide, el principal constituent de les plaques senils. Les concentracions BACE1-AS són elevades en individus amb la malaltia d'Alzheimer i en ratolins transgènics amb la proteïna precursora de l'amiloide.
miR-96 i pèrdua de l'oïda
[modifica]Variacions en la regió d'inici del miR-96 madur s'ha associat a una malaltia autosòmica dominant de progressiva pèrdua d'oïda en humans i ratolins. Els ratolins homozigots mutants estaven profundament sords, sense mostrar resposta per part de la còclea. Els humas i els ratolins heterozigots progressivament perden la seva habilitat per escoltar.[62] [63] [64]
Distinció entre ARN funcional (fARN) i ncARN
[modifica]Moltes publicacions[65][66][67] han començat a utilitzar el terme fARN, en oposició al ncARN, per descriure regions funcionals a nivell d'ARN que tal vegada podrien o no podrien mantenir separats ARN transcripts. A més a més, cada ncARN és un fARN, però existeixen fARN (tals com riboswitches, elements SEICS i altres regions cis-reguladores) que no són ncARN. Ara el terme fARN pot també incloure ARNm, ja que aquest ARN codifica proteïnes i, per tant, és funcional. A més, ARNs evolucionats artificialment també estan englobats pel terme fARN. Algunes publicacions[17] afirmen que els termes ncARN i fARN són gairebé sinònims.
Referències
[modifica]- ↑ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S, Patel S, Long J, Stern D, Tammana H, Helt G, Sementchenko V, Piccolboni A, Bekiranov S, Bailey DK, Ganesh M, Ghosh S, Bell I, Gerhard DS, Gingeras TR «Transcriptional maps of 10 human chromosomes at 5-nucleotide resolution.». Science, 308, 5725, 2005, pàg. 1149–54. DOI: 10.1126/science.1108625. PMID: 15790807.
- ↑ ENCODE Project Consortium; Birney, E; Stamatoyannopoulos, JA; Dutta, A; Guigó, R; Gingeras, TR; Margulies, EH; Weng, Z; Snyder, M «Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project.». Nature, 447, 7146, 2007, pàg. 799–816. DOI: 10.1038/nature05874. PMC: 2212820. PMID: 17571346.
- ↑ Washietl S, Pedersen JS, Korbel JO, Stocsits C, Gruber AR, Hackermüller J, Hertel J, Lindemeyer M, Reiche K, Tanzer A, Ucla C, Wyss C, Antonarakis SE, Denoeud F, Lagarde J, Drenkow J, Kapranov P, Gingeras TR, Guigó R, Snyder M, Gerstein MB, Reymond A, Hofacker IL, Stadler PF «Structured RNAs in the ENCODE selected regions of the human genome.». Genome Res, 17, 6, 2007, pàg. 852–64. DOI: 10.1101/gr.5650707. PMC: 1891344. PMID: 17568003.
- ↑ van Bakel H, Nislow C, Blencowe BJ, Hughes TR «Most “dark matter” transcripts are associated with known genes». PLoS Biol, 8, 5, 2010, pàg. e1000371. Arxivat de l'original el 2014-08-03. DOI: 10.1371/journal.pbio.1000371. PMC: 2872640. PMID: 20502517 [Consulta: 4 desembre 2010].
- ↑ Hüttenhofer A, Schattner P, Polacek N «Non-coding RNAs: hope or hype?». Trends Genet, 21, 5, 2005, pàg. 289–97. DOI: 10.1016/j.tig.2005.03.007. PMID: 15851066.
- ↑ Dahm R «Friedrich Miescher and the discovery of DNA». Dev. Biol., 278, 2, febrer 2005, pàg. 274–88. DOI: 10.1016/j.ydbio.2004.11.028. PMID: 15680349 [Consulta: 3 setembre 2010].
- ↑ Caspersson T, Schultz J «Pentose nucleotides in the cytoplasm of growing tissues». Nature, 143, 1939, pàg. 602–3. DOI: 10.1038/143602c0.
- ↑ CRICK FH «On protein synthesis». Symp. Soc. Exp. Biol., 12, 1958, pàg. 138–63. PMID: 13580867 [Consulta: 3 setembre 2010].
- ↑ 9,0 9,1 HOLLEY RW, APGAR J, EVERETT GA, et al. «STRUCTURE OF A RIBONUCLEIC ACID». Science, 147, març 1965, pàg. 1462–5. DOI: 10.1126/science.147.3664.1462. PMID: 14263761 [Consulta: 3 setembre 2010].
- ↑ 10,0 10,1 «The Nobel Prize in Physiology or Medicine 1968». Nobel Foundation. [Consulta: 28 juliol 2007].
- ↑ Madison JT, Everett GA, Kung H «Nucleotide sequence of a yeast tyrosine transfer RNA.». Science, 153, 735, 1966, pàg. 531–4. DOI: 10.1126/science.153.3735.531. PMID: 5938777.
- ↑ Zachau HG, Dütting D, Feldmann H, Melchers F, Karau W «Serine specific transfer ribonucleic acids. XIV. Comparison of nucleotide sequences and secondary structure models». Cold Spring Harb. Symp. Quant. Biol., 31, 1966, pàg. 417–24. PMID: 5237198 [Consulta: 3 setembre 2010].
- ↑ Dudock BS, Katz G, Taylor EK, Holley RW «Primary structure of wheat germ phenylalanine transfer RNA». Proc. Natl. Acad. Sci. U.S.A., 62, 3, març 1969, pàg. 941-5. DOI: 10.1073/pnas.62.3.941. PMC: 223689. PMID: 5257014 [Consulta: 3 setembre 2010].
- ↑ Cramer F, Doepner H, Haar F VD, Schlimme E, Seidel H «On the conformation of transfer RNA». Proc. Natl. Acad. Sci. U.S.A., 61, 4, desembre 1968, pàg. 1384–91. DOI: 10.1073/pnas.61.4.1384. PMC: 225267. PMID: 4884685 [Consulta: 3 setembre 2010].
- ↑ Ladner JE, Jack A, Robertus JD, et al. «Structure of yeast phenylalanine transfer RNA at 2.5 A resolution». Proc. Natl. Acad. Sci. U.S.A., 72, 11, novembre 1975, pàg. 4414–8. DOI: 10.1073/pnas.72.11.4414. PMC: 388732. PMID: 1105583 [Consulta: 3 setembre 2010].
- ↑ Kim SH, Quigley GJ, Suddath FL, et al. «Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain». Science, 179, 70, 1973, pàg. 285–8. DOI: 10.1126/science.179.4070.285. PMID: 4566654.
- ↑ 17,0 17,1 Eddy SR «Non-coding RNA genes and the modern RNA world». Nat. Rev. Genet., 2, 12, desembre 2001, pàg. 919–29. DOI: 10.1038/35103511. PMID: 11733745 [Consulta: 3 setembre 2010].
- ↑ Daneholt, Bertil. «Advanced Information: RNA interference». The Nobel Prize in Physiology or Medicine 2006. [Consulta: 25 gener 2007].
- ↑ Jeffares DC, Poole AM, Penny D «Relics from the RNA world.». J Mol Evol, 46, 1, 1998, pàg. 18–36. DOI: 10.1007/PL00006280. PMID: 9419222.
- ↑ Poole AM, Jeffares DC, Penny D «The path from the RNA world.». J Mol Evol, 46, 1, 1998, pàg. 1–17. DOI: 10.1007/PL00006275. PMID: 9419221.
- ↑ Poole A, Jeffares D, Penny D «Early evolution: prokaryotes, the new kids on the block.». Bioessays, 21, 10, 1999, pàg. 880–9. DOI: 10.1002/(SICI)1521-1878(199910)21:10<880::AID-BIES11>3.0.CO;2-P. PMID: 10497339.
- ↑ Ban N, Nissen P, Hansen J, Moore P, Steitz T «The complete atomic structure of the large ribosomal subunit at 2.4 ångström resolution». Science, 289, 5481, 2000, pàg. 905–20. DOI: 10.1126/science.289.5481.905. PMID: 10937989.
- ↑ Zhu Y, Stribinskis V, Ramos KS, Li Y «Sequence analysis of RNase MRP RNA reveals its origination from eukaryotic RNase P RNA». RNA, 12, 5, 2006, pàg. 699–706. DOI: 10.1261/ARN.2284906. PMC: 1440897. PMID: 16540690.
- ↑ Kishore S, Stamm S «The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C». Science, 311, 5758, 2006, pàg. 230–231. DOI: 10.1126/science.1118265. PMID: 16357227.
- ↑ Reiner R, Ben-Asouli Y, Krilovetzky I, Jarrous N «A role for the catalytic ribonucleoprotein RNAse P in RNA polymerase III transcription.». Genes Dev, 20, 12, 2006, pàg. 1621–35. DOI: 10.1101/gad.386706. PMC: 1482482. PMID: 16778078.
- ↑ Espinoza CA, Allen TA, Hieb AR, Kugel JF, Goodrich JA «B2 RNA binds directly to RNA polymerase II to repress transcript synthesis.». Nat Struct Mol Biol, 11, 9, 2004, pàg. 822–9. DOI: 10.1038/nsmb812. PMID: 15300239.
- ↑ Hirota K, Miyoshi T, Kugou K, Hoffman CS, Shibata T, Ohta K «Stepwise chromatin remodelling by a cascade of transcription initiation of non-coding RNAs.». Nature, 456, 7218, 2008, pàg. 130–4. DOI: 10.1038/nature07348. PMID: 18820678.
- ↑ Park Y, Kelley RL, Oh H, Kuroda MI, Meller VH «Extent of chromatin spreading determined by roX RNA recruitment of MSL proteins.». Science, 298, 5598, 2002, pàg. 1620–3. DOI: 10.1126/science.1076686. PMID: 12446910.
- ↑ Wadler CS, Vanderpool CK «A dual function for a bacterial small RNA: SgrS performs base pairing-dependent regulation and encodes a functional polypeptide.». Proc Natl Acad Sci USA, 104, 51, 2007, pàg. 20454–9. DOI: 10.1073/pnas.0708102104. PMC: 2154452. PMID: 18042713.
- ↑ Dinger ME, Pang KC, Mercer TR, Mattick JS «Differentiating protein-coding and noncoding RNA: challenges and ambiguities.». PLoS Comput Biol, 4, 11, 2008, pàg. e1000176. DOI: 10.1371/journal.pcbi.1000176. PMC: 2518207. PMID: 19043537.
- ↑ Saraiya AA, Wang CC «snoRNA, a novel precursor of microARN in Giardia lamblia.». PLoS Pathog, 4, 11, 2008, pàg. e1000224. DOI: 10.1371/journal.ppat.1000224. PMC: 2583053. PMID: 19043559.
- ↑ Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N, Meister G «A human snoRNA with microRNA-like functions.». Mol Cell, 32, 4, 2008, pàg. 519–28. DOI: 10.1016/j.molcel.2008.10.017. PMID: 19026782.
- ↑ Leygue E «Steroid receptor RNA activator (SRA1): unusual bifaceted gene products with suspected relevance to breast cancer.». Nucl Recept Signal, 5, 2007, pàg. e006. DOI: 10.1621/nrs.05006. PMC: 1948073. PMID: 17710122.
- ↑ Zhang J, King ML «Xenopus VegT RNA is localized to the vegetal cortex during oogenesis and encodes a novel T-box transcription factor involved in mesodermal patterning.». Development, 122, 12, 1996, pàg. 4119–29. PMID: 9012531.
- ↑ Kloc M, Wilk K, Vargas D, Shirato Y, Bilinski S, Etkin LD «Potential structural role of non-coding and coding RNAs in the organization of the cytoskeleton at the vegetal cortex of Xenopus oocytes.». Development, 132, 15, 2005, pàg. 3445–57. DOI: 10.1242/dev.01919. PMID: 16000384.
- ↑ Jenny A, Hachet O, Závorszky P, Cyrklaff A, Weston MD, Johnston DS, Erdélyi M, Ephrussi A «A translation-independent role of oskar RNA in early Drosophila oogenesis.». Development, 133, 15, 2006, pàg. 2827–33. DOI: 10.1242/dev.02456. PMID: 16835436.
- ↑ Gultyaev AP, Roussis A «Identification of conserved secondary structures and expansion segments in enod40 RNAs reveals new enod40 homologues in plants.». Nucleic Acids Res, 35, 9, 2007, pàg. 3144-52. DOI: 10.1093/nar/gkm173. PMC: 1888808. PMID: 17452360.
- ↑ Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, Sweet-Cordero A, Ebert BL, Mak RH, Ferrando AA, Downing JR, Jacks T, Horvitz HR, Golub TR «MicroRNA expression profiles classify human cancers». Nature, 435, 7043, 2005, pàg. 834–8. DOI: 10.1038/nature03702. PMID: 15944708.
- ↑ Pibouin L, Villaudy J, Ferbus D, Muleris M, Prospéri MT, Remvikos Y, Goubin G «Cloning of the mRNA of overexpression in colon carcinoma-1: a sequence overexpressed in a subset of colon carcinomas». Cancer Genet Cytogenet, 133, 1, 2002, pàg. 55–60. DOI: 10.1016/S0165-4608(01)00634-3. PMID: 11890990.
- ↑ Fu X, Ravindranath L, Tran N, Petrovics G, Srivastava S «Regulation of apoptosis by a prostate-specific and prostate cancer-associated noncoding gene, PCGEM1». DNA Cell Biol, 25, 3, 2006, pàg. 135–41. DOI: 10.1089/dna.2006.25.135. PMID: 16569192.
- ↑ Mourtada-Maarabouni M, Pickard MR, Hedge VL, Farzaneh F, Williams GT «GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer.». Oncogene, 28, 2, 2009, pàg. 195–208. DOI: 10.1038/onc.2008.373. PMID: 18836484.
- ↑ Dong XY, Guo P, Boyd J, Sun X, Li Q, Zhou W, Dong JT «Implication of snoRNA U50 in human breast cancer.». J Genet Genomics, 36, 8, 2009, pàg. 447–54. DOI: 10.1016/S1673-8527(08)60134-4. PMC: 2854654. PMID: 19683667.
- ↑ Christov CP, Trivier E, Krude T «Noncoding human Y RNAs are overexpressed in tumours and required for cell proliferation». Br J Cancer, 98, 5, 2008, pàg. 981–8. DOI: 10.1038/sj.bjc.6604254. PMC: 2266855. PMID: 18283318.
- ↑ Farh KK, Grimson A, Jan C, Lewis BP, Johnston WK, Lim LP, Burge CB, Bartel DP «The widespread impact of mammalian MicroRNAs on mRNA repression and evolution». Science, 310, 5755, 2005, pàg. 1817–21. DOI: 10.1126/science.1121158. PMID: 16308420.
- ↑ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM «Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs». Nature, 433, 7027, 2005, pàg. 769–73. DOI: 10.1038/nature03315. PMID: 15685193.
- ↑ Christov CP, Gardiner TJ, Szüts D, Krude T «Functional requirement of noncoding Y RNAs for human chromosomal DNA replication». Mol Cell Biol, 26, 18, 2006, pàg. 6993-7004. DOI: 10.1128/MCB.01060-06. PMC: 1592862. PMID: 16943439.
- ↑ Calin GA, Ferracin M, Cimmino A, et al «A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia». N. Engl. J. Med., 353, 17, octubre 2005, pàg. 1793–801. Arxivat de l'original el 2009-02-27. DOI: 10.1056/NEJMoa050995. PMID: 16251535 [Consulta: 3 desembre 2010].
- ↑ Calin, GA; Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K, Rassenti L, Kipps T, Negrini M, Bullrich F, Croce CM «Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia». Proc Natl Acad Sci USA, 99, 24, 2002, pàg. 15524–15529. DOI: 10.1073/pnas.242606799. PMC: 137750. PMID: 12434020.
- ↑ Hu Z, Chen J, Tian T, Zhou X, Gu H, Xu L, Zeng Y, Miao R, Jin G, Ma H, Chen Y, Shen H «Genetic variants of miRNA sequences and non-small cell lung cancer survival». J Clin Invest, 118, 7, 2008, pàg. 2600–8. DOI: 10.1172/JCI34934. PMC: 2402113. PMID: 18521189.
- ↑ Shen J, Ambrosone CB, Zhao H «Novel genetic variants in microRNA genes and familial breast cancer». Int J Cancer, 124, 5, 2009, pàg. 1178–82. DOI: 10.1002/ijc.24008. PMID: 19048628.
- ↑ Sahoo T, del Gaudio D, German JR, Shinawi M, Peters SU, Person RE, Garnica A, Cheung SW, Beaudet AL «Prader–Willi phenotype caused by paternal deficiency for the HBII-85 C/D box small nucleolar RNA cluster.». Nat Genet, 40, 6, 2008, pàg. 719–21. DOI: 10.1038/ng.158. PMC: 2705197. PMID: 18500341.
- ↑ Ding F, Li HH, Zhang S, Solomon NM, Camper SA, Cohen P, Francke U «SnoRNA Snord116 (Pwcr1/MBII-85) deletion causes growth deficiency and hyperphagia in mice.». PLoS ONE, 3, 3, 2008, pàg. e1709. DOI: 10.1371/journal.pone.0001709. PMC: 2248623. PMID: 18320030.
- ↑ Ding F, Prints Y, Dhar MS, Johnson DK, Garnacho-Montero C, Nicholls RD, Francke U «Lack of Pwcr1/MBII-85 snoRNA is critical for neonatal lethality in Prader–Willi syndrome mouse models.». Mamm Genome, 16, 6, 2005, pàg. 424–31. DOI: 10.1007/s00335-005-2460-2. PMID: 16075369.
- ↑ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A «snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions.». Gene, 408, 1-2, 2008, pàg. 172–9. DOI: 10.1016/j.gene.2007.10.037. PMID: 18160232.
- ↑ Bolton PF, Veltman MW, Weisblatt E, et al. «Chromosome 15q11-13 abnormalities and other medical conditions in individuals with autism spectrum disorders». Psychiatr. Genet., 14, 3, setembre 2004, pàg. 131–7. DOI: 10.1097/00041444-200409000-00002. PMID: 15318025.
- ↑ Cook EH, Scherer SW «Copy-number variations associated with neuropsychiatric conditions». Nature, 455, 7215, octubre 2008, pàg. 919–23. DOI: 10.1038/nature07458. PMID: 18923514.
- ↑ Nakatani J, Tamada K, Hatanaka F, et al. «Abnormal behavior in a chromosome-engineered mouse model for human 15q11-13 duplication seen in autism». Cell, 137, 7, juny 2009, pàg. 1235–46. DOI: 10.1016/j.cell.2009.04.024. PMID: 19563756.
- ↑ Ridanpää M, van Eenennaam H, Pelin K, Chadwick R, Johnson C, Yuan B, vanVenrooij W, Pruijn G, Salmela R, Rockas S, Mäkitie O, Kaitila I, de la Chapelle A «Mutations in the RNA component of RNase MRP cause a pleiotropic human disease, cartilage-hair hypoplasia.». Cell, 104, 2, 2001, pàg. 195–203. DOI: 10.1016/S0092-8674(01)00205-7. PMID: 11207361.
- ↑ Martin AN, Li Y «RNase MRP RNA and human genetic diseases.». Cell Res, 17, 3, 2007, pàg. 219–26. DOI: 10.1038/sj.cr.7310120. PMID: 17189938.
- ↑ Kavadas FD, Giliani S, Gu Y, Mazzolari E, Bates A, Pegoiani E, Roifman CM, Notarangelo LD «Variability of clinical and laboratory features among patients with ribonuclease mitochondrial RNA processing endoribonuclease gene mutations.». J Allergy Clin Immunol, 122, 6, 2008, pàg. 1178–84. DOI: 10.1016/j.jaci.2008.07.036. PMID: 18804272.
- ↑ Faghihi MA, Modarresi F, Khalil AM, Wood DE, Sahagan BG, Morgan TE, Finch CE, St Laurent G, Kenny PJ, Wahlestedt C «Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase.». Nat Med, 14, 7, 2008, pàg. 723–30. DOI: 10.1038/nm1784. PMC: 2826895. PMID: 18587408.
- ↑ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA «Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss.». Nat Genet, 41, 5, 2009, pàg. 609–13. DOI: 10.1038/ng.355. PMID: 19363479.
- ↑ Lewis MA, Quint E, Glazier AM, Fuchs H, De Angelis MH, Langford C, van Dongen S, Abreu-Goodger C, Piipari M, Redshaw N, Dalmay T, Moreno-Pelayo MA, Enright AJ, Steel KP «An ENU-induced mutation of miR-96 associated with progressive hearing loss in mice.». Nat Genet, 41, 5, 2009, pàg. 614–8. DOI: 10.1038/ng.369. PMC: 2705913. PMID: 19363478.
- ↑ Soukup GA «Little but loud: Small RNAs have a resounding affect on ear development.». Brain Res, 1277, 2009, pàg. 104–14. DOI: 10.1016/j.brainres.2009.02.027. PMC: 2700218. PMID: 19245798.
- ↑ Richard J. Carter, Inna Dubchak, Stephen R. Holbrook «A computational approach to identify genes for functional RNAs in genomic sequences». Nucleic Acids Research, 29, 19, 2001, pàg. 3928–3938. PMC: 60242. PMID: 11574674.
- ↑ Jakob Skou Pedersen, Gill Bejerano, Adam Siepel, Kate Rosenbloom, Kerstin Lindblad-Toh, Eric S. Lander, Jim Kent, Webb Miller, David Haussler «Identification and Classification of Conserved RNA Secondary Structures in the Human Genome». PLOS Computational Biology, 2, 4, 2006, pàg. e33. DOI: 10.1371/journal.pcbi.0020033. PMC: 1440920. PMID: 16628248.[Enllaç no actiu]
- ↑ Tomas Babak, Benjamin J Blencowe, Timothy R Hughes «Considerations in the identification of functional RNA structural elements in genomic alignments». BMC Bioinformatics, 8, 8, 2007, pàg. 33. DOI: 10.1186/1471-2105-8-21. PMC: 1783863. PMID: 17244370.
Enllaços externs
[modifica]- The Rfam Database Arxivat 2012-12-05 at Archive.is — una llista precisa de families de ncARNs relacionats.
- NONCODE.org Arxivat 2011-02-22 a Wayback Machine. — una base de dades gratuïta de tots els tipus de ncARNs (excepte tARNs i rARNs).