Basenexzisionsreparatur

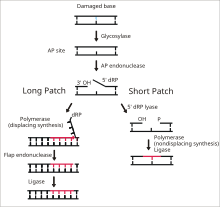

Basenexzisionsreparatur (englisch base excision repair, BER) bezeichnet einen Mechanismus der DNA-Reparatur von bestimmten DNA-Schäden, bei der eine Nukleinbase ausgetauscht oder ein Einzelstrangbruch geschlossen wird.
Eigenschaften
[Bearbeiten | Quelltext bearbeiten]Die Basenexzisionsreparatur repariert DNA-Schäden an Nukleinbasen, die aus Oxidation, Desaminierung und Alkylierung resultieren sowie abasische Stellen.[1][2] Die BER repariert sowohl abasische Stellen als auch Einzelstrangbrüche,[3] sowohl an genomischer DNA als auch an mtDNA.[4] Sie ist zudem bei der Immunantwort an der somatischen Hypermutation und dem Klassenwechsel beteiligt.[5] Die Aktivität der BER wird durch posttranslationale Modifikationen reguliert.[6] Defekte in der BER treten bei Krebs, neurodegenerativen Erkrankungen, Entwicklungsstörungen, Immunschwäche und beim Altern auf.[7]
Die BER beginnt mit einer von 11 DNA-Glykosylasen, welche die veränderte Nukleinbase hydrolysiert.[1] Ohne Nukleinbase wird die Position als abasische Stelle bezeichnet.[1][8] Anschließend folgen vier weitere Enzyme: Endonuklease, DNA-Lyase, DNA-Polymerase und DNA-Ligase.[9][10] Danach wird durch eine Endonuklease das Phosphodiester-Rückgrat der DNA geschnitten, gefolgt von einer Kürzung der Enden durch die DNA-Lyase. Die Lücke wird durch die DNA-Polymerase mit neuen Nukleotiden aufgefüllt und der Einzelstrangbruch wird durch die DNA-Ligase geschlossen.[11]
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c H. E. Krokan, M. Bjørås: Base excision repair. In: Cold Spring Harbor perspectives in biology. Band 5, Nummer 4, April 2013, S. a012583, doi:10.1101/cshperspect.a012583, PMID 23545420, PMC 3683898 (freier Volltext).
- ↑ Dhara Gohil, Altaf H. Sarker, Rabindra Roy: Base Excision Repair: Mechanisms and Impact in Biology, Disease, and Medicine. In: International Journal of Molecular Sciences. 2023, Band 24, Nummer 18, S. 14186 doi:10.3390/ijms241814186. PMID 37762489. PMC 10531636 (freier Volltext).
- ↑ S. Zhao, S. Tadesse, D. Kidane: Significance of base excision repair to human health. In: International review of cell and molecular biology. Band 364, 2021, S. 163–193, doi:10.1016/bs.ircmb.2021.05.002, PMID 34507783.
- ↑ K. Allkanjari, R. A. Baldock: Beyond base excision repair: an evolving picture of mitochondrial DNA repair. In: Bioscience Reports. Band 41, Nummer 10, Oktober 2021, S. , doi:10.1042/BSR20211320, PMID 34608928, PMC 8527207 (freier Volltext).
- ↑ S. S. Wallace: Base excision repair: a critical player in many games. In: DNA repair. Band 19, Juli 2014, S. 14–26, doi:10.1016/j.dnarep.2014.03.030, PMID 24780558, PMC 4100245 (freier Volltext).
- ↑ R. J. Carter, J. L. Parsons: Base Excision Repair, a Pathway Regulated by Posttranslational Modifications. In: Molecular and Cellular Biology. Band 36, Nummer 10, Mai 2016, S. 1426–1437, doi:10.1128/MCB.00030-16, PMID 26976642, PMC 4859697 (freier Volltext).
- ↑ J. M. Hinz, W. Czaja: Facilitation of base excision repair by chromatin remodeling. In: DNA repair. Band 36, Dezember 2015, S. 91–97, doi:10.1016/j.dnarep.2015.09.011, PMID 26422134, PMC 4688104 (freier Volltext).
- ↑ A. Prakash, S. Doublié: Base Excision Repair in the Mitochondria. In: Journal of cellular biochemistry. Band 116, Nummer 8, August 2015, S. 1490–1499, doi:10.1002/jcb.25103, PMID 25754732, PMC 4546830 (freier Volltext).
- ↑ W. A. Beard, J. K. Horton, R. Prasad, S. H. Wilson: Eukaryotic Base Excision Repair: New Approaches Shine Light on Mechanism. In: Annual review of biochemistry. Band 88, Juni 2019, S. 137–162, doi:10.1146/annurev-biochem-013118-111315, PMID 31220977, PMC 8956022 (freier Volltext).
- ↑ N. N. Hindi, N. Elsakrmy, D. Ramotar: The base excision repair process: comparison between higher and lower eukaryotes. In: Cellular and molecular life sciences : CMLS. Band 78, Nummer 24, Dezember 2021, S. 7943–7965, doi:10.1007/s00018-021-03990-9, PMID 34734296.
- ↑ Y. J. Kim, D. M. Wilson: Overview of base excision repair biochemistry. In: Current molecular pharmacology. Band 5, Nummer 1, Januar 2012, S. 3–13, doi:10.2174/1874467211205010003, PMID 22122461, PMC 3459583 (freier Volltext).