Νουκλεόσωμα

Το νουκλεόσωμα είναι η βασική δομική μονάδα συσκευασίας του DNA[1] στους ευκαρυώτες. Τη δομή ενός νουκλεοσώματος συνιστά ένα τμήμα DNA που τυλίγεται γύρω από οκτώ ιστονικές πρωτεΐνες,[2] προσομοιάζοντας ένα νήμα τυλιγμένο γύρω από ένα μασούρι.
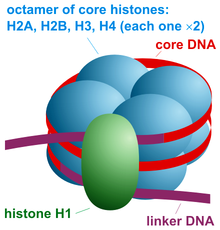
Το πυρηνικό σωμάτιο νουκλεοσώματος αποτελείται από περίπου 146 ζεύγη βάσεων (bp) DNA[3] τυλιγμένου ως αριστερόστροφο σπείραμα με 1,67 υπερελικωμένες στροφές γύρω από ένα οκταμερές ιστονών, το οποίο αποτελείται από 2 μόρια καθεμίας από τις ιστόνες H2A, H2B, H3 και H4.[4] Τα πυρηνικά σωμάτια συνδέονται με τμήματα συνδετικού DNA, το οποίο μπορεί να έχει μήκος έως και περίπου 80 bp. Τεχνικά, ένα νουκλεόσωμα ορίζεται ως το πυρηνικό σωμάτιο μαζί με μία από αυτές τις συνδετικές περιοχές. Ωστόσο, ο όρος νουκλεόσωμα συχνά ταυτίζεται μόνο με το πυρηνικό σωμάτιο.[5] Χάρτες τοποθεσίας των νουκλεοσωμάτων ολόκληρου του γονιδιώματος είναι πλέον διαθέσιμοι για πολλούς οργανισμούς-μοντέλα, συμπεριλαμβανομένου του ήπατος και εγκεφάλου ποντικού.[6]
Οι συνδετικές ιστόνες όπως η Η1 και οι ισομορφές της εμπλέκονται στη συμπύκνωση της χρωματίνης και βρίσκονται στη βάση του νουκλεοσώματος κοντά στην είσοδο και έξοδο του DNA, δεσμευόμενες με τη συνδετική περιοχή του DNA.[7] Τα μη συμπυκνωμένα νουκλεοσώματα χωρίς τη συνδετική ιστόνη προσομοιάζουν "χάντρες σε μια χορδή DNA" σε ένα ηλεκτρονικό μικροσκόπιο.[8]
Τα νουκλεοσώματα πιστεύεται ότι φέρουν επιγενετικά κληρονομήσιμες πληροφορίες με τη μορφή ομοιοπολικών τροποποιήσεων των κύριων ιστονών τους. Οι θέσεις των νουκλεοσωμάτων στο γονιδίωμα δεν είναι τυχαίες και είναι σημαντικό να είναι γνωστή η θέση καθενός νουκλεοσώματος, διότι αυτό καθορίζει την προσβασιμότητα του DNA στις ρυθμιστικές πρωτεΐνες.[9]
Τα νουκλεοσώματα παρατηρήθηκαν για πρώτη φορά ως σωματίδια στο ηλεκτρονικό μικροσκόπιο από τους Ντον και Άντα Όλινς το 1974,[10] ενώ η ύπαρξη και η δομή τους (ως οκταμερή ιστονών που περιβάλλονται από περίπου 200 ζεύγη βάσεων DNA) προτάθηκαν από τον Ρότζερ Κορνμπεργκ.[11] [12] Ο ρόλος του νουκλεοσώματος ως γενικού καταστολέα γονιδίων καταδείχθηκε in vitro από τους Λορχ et al.,[13] και in vivo από τους Χαν και Γκρανστάιντο, το 1987 και το 1988 αντίστοιχα.[14]
Το DNA πρέπει να συσκευαστεί σε νουκλεοσώματα ώστε να χωρέσει στον πυρήνα του κυττάρου.[15] Εκτός από το πακετάρισμα σε νουκλεοσώματα, η ευκαρυωτική χρωματίνη συμπιέζεται περαιτέρω με αναδίπλωση σε μια σειρά πιο σύνθετων δομών, σχηματίζοντας τελικά ένα χρωμόσωμα.
Σε αντίθεση με τα περισσότερα ευκαρυωτικά κύτταρα, τα ώριμα σπερματοκύτταρα χρησιμοποιούν σε μεγάλο βαθμό πρωταμίνες για τη συσκευασία του γονιδιωματικού τους DNA, πιθανότατα για να επιτύχουν ακόμη υψηλότερη αναλογία συμπύκνωσης.[16] Ισοδύναμα ιστόνης και μια απλοποιημένη δομή χρωματίνης έχουν επίσης βρεθεί στα Αρχαία,[17] υποδηλώνοντας ότι οι ευκαρυώτες δεν είναι οι μόνοι οργανισμοί που χρησιμοποιούν νουκλεοσώματα.
Δομή
[Επεξεργασία | επεξεργασία κώδικα]Δομή του πυρηνικού σωματίου
[Επεξεργασία | επεξεργασία κώδικα]
Το πυρηνικό σωμάτιο νουκλεοσώματος (NCP)
[Επεξεργασία | επεξεργασία κώδικα]Το πυρηνικό σωμάτιο νουκλεοσώματος (φαίνεται στο σχήμα αριστερά) αποτελείται από περίπου 146 ζεύγη βάσεων (bp) DNA [3] τυλιγμένου σε 1,67 αριστερόστροφες υπερελικωμένες συστροφές γύρω από το οκταμερές των ιστονών, το οποίο αποτελείται από 2 αντίγραφα καθεμίας από τις πυρηνικές ιστόνες H2A, H2B, H3 και Η4. Τα γειτονικά νουκλεοσώματα ενώνονται με ένα τμήμα ελεύθερου DNA που ονομάζεται συνδετικό DNA (linker DNA), το οποίο κυμαίνεται από 10-80 bp σε μήκος ανάλογα με το είδος και τον τύπο του ιστού.[17] Η συνολική κατασκευή δημιουργεί έναν κύλινδρο διαμέτρου 11 nm και ύψους 5,5 nm.


Τα πυρηνικά σωμάτια νουκλεοσώματος παρατηρούνται όταν η χρωματίνη στη μεσόφαση υποβάλλεται σε επεξεργασία ώστε να ξεδιπλωθεί μερικώς. Η προκύπτουσα εικόνα, σε ένα ηλεκτρονικό μικροσκόπιο, είναι "χάντρες σε μια χορδή". Η χορδή είναι το DNA, ενώ κάθε χάντρα στο νουκλεόσωμα είναι ένα πυρηνικό σωματίδιο. Το πυρηνικό σωματίδιο νουκλεοσώματος αποτελείται από DNA και ιστονικές πρωτεΐνες.[21]
Μερική πέψη της χρωματίνης με DNAαση αποκαλύπτει τη δομή της με τα νουκλεοσώματα. Επειδή τμήματα DNA των πυρηνικών σωματιδίων νουκλεοσώματος είναι λιγότερο προσβάσιμα για τη DNAαση από ό,τι τα συνδετικά τμήματα, το DNA πέπτεται σε θραύσματα μήκους ίσου με το πολλαπλάσιο της απόστασης μεταξύ των νουκλεοσωμάτων (180, 360, 540 ζεύγη βάσεων κ.λπ.). Ως εκ τούτου, ένα πολύ χαρακτηριστικό μοτίβο παρόμοιο με μια σκάλα γίνεται ορατό κατά την ηλεκτροφόρηση πηκτώματος αυτού του DNA.[18] Τέτοια πέψη μπορεί να συμβεί επίσης υπό φυσικές συνθήκες κατά την απόπτωση (προγραμματισμένος κυτταρικός θάνατος), διότι η αυτοαποδόμηση του DNA είναι συνήθως ο ρόλος της.
Αλληλεπιδράσεις πρωτεϊνών εντός του νουκλεοσώματος
[Επεξεργασία | επεξεργασία κώδικα]Οι πυρηνικές ιστονικές πρωτεΐνες περιέχουν ένα χαρακτηριστικό δομικό μοτίβο που ονομάζεται «πτυχή ιστόνης» και αποτελείται από τρεις α-έλικες (α1-3) που χωρίζονται από δύο βρόχους (L1-2). Σε διάλυμα, οι ιστόνες σχηματίζουν ετεροδιμερή Η2Α-Η2Β και ετεροτετραμερή Η3-Η4. Οι ιστόνες διμερίζονται γύρω από τις μακρές α2 έλικες σε αντι-παράλληλο προσανατολισμό και, στην περίπτωση των Η3 και Η4, δύο τέτοια διμερή σχηματίζουν μια δέσμη 4 ελίκων που σταθεροποιείται από εκτεταμένη αλληλεπίδραση Η3-Η3'. Το διμερές Η2Α / Η2Β συνδέεται στο τετραμερές Η3 / Η4 λόγω αλληλεπιδράσεων μεταξύ των Η4 και Η2Β, οι οποίες περιλαμβάνουν τον σχηματισμό ενός υδρόφοβου συμπλέγματος.[4] Το οκταμερές των ιστονών σχηματίζεται από ένα κεντρικό τετραμερές Η3 / Η4 που περικλείεται ανάμεσα σε δύο διμερή Η2Α / Η2Β. Λόγω του εξαιρετικά βασικού φορτίου και των τεσσάρων πυρηνικών ιστονών, το οκταμερές ιστόνης είναι σταθερό μόνο παρουσία DNA ή πολύ υψηλών συγκεντρώσεων άλατος.
Αλληλεπιδράσεις Ιστόνης - DNA
[Επεξεργασία | επεξεργασία κώδικα]Το νουκλεόσωμα περιέχει πάνω από 120 άμεσες αλληλεπιδράσεις πρωτεΐνης-DNA και αρκετές εκατοντάδες μεσολαβούμενες από νερό. [22] Οι άμεσες αλληλεπιδράσεις πρωτεΐνης - DNA δεν κατανέμονται ομοιόμορφα στην επιφάνεια του οκταμερούς και βρίσκονται σε διακριτές θέσεις. Αυτές οφείλονται στον σχηματισμό δύο τύπων θέσεων πρόσδεσης DNA εντός του οκταμερούς, τη θέση α1α1, η οποία χρησιμοποιεί την έλικα α1 από δύο παρακείμενες ιστόνες, και τη θέση L1L2 που σχηματίζεται από τους βρόχους L1 και L2. Δεσμοί άλατος και υδρογόνου μεταξύ τόσο των βασικών ομάδων και υδροξυλομάδων της πλευρικής αλυσίδας, όσο και των αμιδίων της κύριας αλυσίδας με τα φωσφορικά του σκελετού του DNA, συνιστούν το μεγαλύτερο μέρος των αλληλεπιδράσεων με το DNA. Αυτό είναι σημαντικό, δεδομένου ότι η ευρεία κατανομή των νουκλεοσωμάτων κατά μήκος των γονιδιωμάτων απαιτεί αυτά να αποτελούν μη ειδικής αλληλουχίας παράγοντες πρόσδεσης στο DNA. Αν και τα νουκλεοσώματα τείνουν να προτιμούν ορισμένες αλληλουχίες DNA από άλλες,[23] είναι ικανά να συνδέονται πρακτικά με οποιαδήποτε αλληλουχία, κάτι το οποίο πιστεύεται ότι οφείλεται στην ευελιξία του σχηματισμού αυτών των αλληλεπιδράσεων στις οποίες μεσολαβεί το νερό. Επιπλέον, συμβαίνουν μη πολικές αλληλεπιδράσεις μεταξύ των πρωτεϊνικών πλευρικών αλυσίδων και των ομάδων δεοξυριβόζης. Επίσης, μια πλευρική αλυσίδα αργινίνης παρεμβάλλεται εντός της μικρής αύλακας του DNA και στις 14 θέσεις στις οποίες αντικρίζει την επιφάνεια του οκταμερούς. Η κατανομή και η ισχύς των θέσεων πρόσδεσης DNA γύρω από την επιφάνεια του οκταμερούς παραμορφώνει το DNA εντός του πυρήνα του νουκλεοσώματος. Το DNA κάμπτεται ανομοιόμορφα και περιέχει επίσης ελαττωματικές συστροφές. Η συστροφή ελεύθερου DNA μορφής Β σε διάλυμα είναι 10,5 bp ανά στροφή. Ωστόσο, η συνολική συστροφή του νουκλεοσωμικού DNA είναι μόνο 10,2 bp ανά στροφή, κυμαινόμενη από τιμή 9,4 έως 10,9 bp ανά στροφή.
Περιοχές της ουράς ιστόνης
[Επεξεργασία | επεξεργασία κώδικα]Οι επεκτάσεις ουράς των ιστονών αποτελούν έως και το 30% κατά μάζα των ιστονών, αλλά δεν είναι ορατές στις κρυσταλλικές δομές των νουκλεοσωμάτων λόγω της υψηλής ενδογενούς ευκαμψίας τους και πιστεύεται ότι σε μεγάλο βαθμό δεν έχουν δομή.[24] Οι αμινοτελικές ουρές των ιστονών Η3 και Η2Β διέρχονται από ένα κανάλι που σχηματίζεται από τις μικρές αύλακες των δύο αλυσίδων DNA, προεξέχοντας από το DNA κάθε 20 bp. Η αμινοτελική ουρά της ιστόνης Η4, από την άλλη, έχει μια περιοχή υψηλά βασικών αμινοξέων (16-25), η οποία, στην κρυσταλλική δομή, αλληλεπιδρά με την εξαιρετικά όξινη επιφανειακή περιοχή ενός διμερούς H2A-H2B ενός άλλου νουκλεοσώματος, κάτι που ενδεχομένως σχετίζεται με τη δομή ανώτερης τάξης των νουκλεοσωμάτων. Αυτή η αλληλεπίδραση θεωρείται ότι συμβαίνει και υπό φυσιολογικές συνθήκες και υποδηλώνει ότι η ακετυλίωση της ουράς της Η4 παραμορφώνει τη δομή ανώτερης τάξης της χρωματίνης.
Δομή ανώτερης τάξης
[Επεξεργασία | επεξεργασία κώδικα]
Η οργάνωση του DNA που επιτυγχάνεται από το νουκλεόσωμα δεν μπορεί να εξηγήσει πλήρως τη συσκευασία του DNA που παρατηρείται στον κυτταρικό πυρήνα. Απαιτείται περαιτέρω συμπύκνωση της χρωματίνης στον πυρήνα του κυττάρου, αλλά δεν είναι ακόμη καλά κατανοητή. Η τρέχουσα κατανόηση[25] είναι ότι τα επαναλαμβανόμενα νουκλεοσώματα με το παρεμβαλλόμενο "συνδετικό" DNA σχηματίζουν μια ίνα 10 nm και περιγράφονται ως "χάντρες σε μια χορδή" που έχουν αναλογία συσκευασίας περίπου πέντε έως δέκα.[17] Μια αλυσίδα νουκλεοσωμάτων μπορεί να οργανωθεί σε μια ίνα 30 nm, μια συμπαγή δομή με αναλογία συσκευασίας ~50 της οποίας ο σχηματισμός εξαρτάται από την παρουσία της Η1 ιστόνης.
Έχει παρουσιαστεί μια κρυσταλλική δομή ενός τετρανουκλεοσώματος και έχει χρησιμοποιηθεί για τη δημιουργία μιας προτεινόμενης δομής έλικας της ίνας των 30 nm.[26] Υπάρχει ακόμη αμφιβολία σχετικά με αυτό το μοντέλο, καθώς είναι ασυμβίβαστο με πρόσφατα δεδομένα ηλεκτρονικής μικροσκοπίας.[27] Πέρα από αυτό, η δομή της χρωματίνης είναι ελάχιστα κατανοητή, αλλά κλασικά προτείνεται ότι οι ίνες των 30 nm διατάσσονται σε βρόχους κατά μήκος μιας κεντρικής πρωτεϊνικής σκαλωσιάς για να σχηματίσουν μεταγραφικά ενεργή ευχρωματίνη. Περαιτέρω συμπύκνωση οδηγεί σε μεταγραφικά ανενεργή ετεροχρωμίνη.
Δυναμική
[Επεξεργασία | επεξεργασία κώδικα]Αν και το νουκλεόσωμα είναι ένα πολύ σταθερό σύμπλοκο DNA-πρωτεΐνης, δεν είναι στατικό και έχει αποδειχθεί ότι υφίσταται έναν αριθμό διαφορετικών δομικών αναδιατάξεων, συμπεριλαμβανομένης της ολίσθησης και της έκθεσης θέσεων DNA. Ανάλογα με το πλαίσιο, τα νουκλεοσώματα μπορούν να αναστέλλουν ή να διευκολύνουν τη σύνδεση του μεταγραφικού παράγοντα. Οι θέσεις των νουκλεοσωμάτων ελέγχονται από τρεις σημαντικές συνεισφορές: Πρώτον, η εγγενής συγγένεια δέσμευσης του οκταμερούς ιστονών εξαρτάται από την αλληλουχία DNA. Δεύτερον, το νουκλεόσωμα μπορεί να μετατοπιστεί ή να στρατολογηθεί μέσω της ανταγωνιστικής ή συνεργατικής σύνδεσης άλλων πρωτεϊνικών παραγόντων. Τρίτον, το νουκλεόσωμα μπορεί να μετατοπιστεί ενεργά από εξαρτώμενα από ΑΤΡ σύμπλοκα αναδιαμόρφωσης.[28]
Συναρμολόγηση νουκλεοσωμάτων in vivo
[Επεξεργασία | επεξεργασία κώδικα]Τα νουκλεοσώματα είναι η βασική μονάδα συσκευασίας του DNA και κατασκευάζονται από ιστονικές πρωτεΐνες γύρω από τις οποίες τυλίγεται το DNA. Χρησιμεύουν ως σκαλωσιά για τον σχηματισμό δομής χρωματίνης ανώτερης τάξης καθώς και για ένα στρώμα ρυθμιστικού ελέγχου της γονιδιακής έκφρασης. Τα νουκλεοσώματα συναρμολογούνται γρήγορα στο πρόσφατα συντεθειμένο DNA πίσω από τη διχάλα αντιγραφής.
H3 και H4
[Επεξεργασία | επεξεργασία κώδικα]Οι ιστόνες Η3 και Η4 από αποσυναρμολογημένα παλιά νουκλεοσώματα διατηρούνται κοντά και κατανέμονται τυχαία στο πρόσφατα συντεθειμένο DNA.[29] Συναρμολογούνται από το σύμπλεγμα του παράγοντα συναρμολόγησης χρωματίνης 1 (CAF-1), το οποίο αποτελείται από τρεις υπομονάδες (p150, p60 και p48).[30] Πρόσφατα συντεθειμένες H3 και H4 συναρμολογούνται από τον παράγοντα συναρμολόγησης ζεύξης αντιγραφής (RCAF). Ο RCAF περιέχει την υπομονάδα Asf1, η οποία συνδέεται με τις πρόσφατα συντεθειμένες πρωτεΐνες H3 και H4.[31] Οι παλιές πρωτεΐνες H3 και H4 διατηρούν τις χημικές τους τροποποιήσεις, γεγονός που συμβάλλει στη μετάδοση της επιγενετικής υπογραφής. Οι πρόσφατα συντεθειμένες πρωτεΐνες Η3 και Η4 ακετυλιώνονται σταδιακά σε διαφορετικά κατάλοιπα λυσίνης ως μέρος της διαδικασίας ωρίμανσης της χρωματίνης.[32] Πιστεύεται επίσης ότι οι παλιές πρωτεΐνες Η3 και Η4 στα νέα νουκλεοσώματα στρατολογούν ένζυμα τροποποίησης των ιστονών που σημαίνουν τις νέες ιστόνες, συμβάλλοντας στην επιγενετική μνήμη.
H2A και H2B
[Επεξεργασία | επεξεργασία κώδικα]Σε αντίθεση με τις παλιές H3 και H4, οι παλιές ιστονικές πρωτεΐνες H2A και H2B απελευθερώνονται και αποικοδομούνται. Επομένως, πρόσφατα συναρμολογημένες πρωτεΐνες Η2Α και Η2Β ενσωματώνονται σε νέα νουκλεοσώματα.[33] Οι Η2Α και Η2Β συναρμολογούνται σε διμερή τα οποία στη συνέχεια φορτώνονται σε νουκλεοσώματα από την πρωτεΐνη συναρμολόγησης νουκλεοσωμάτων 1 (ΝΑΡ-1), η οποία επίσης βοηθά στην ολίσθηση των νουκλεοσωμάτων.[34] Τα νουκλεοσώματα αποδιατάσσονται επίσης από εξαρτώμενα από ΑΤΡ σύμπλοκα αναδιαμόρφωσης νουκλεοσωμάτων που περιέχουν ένζυμα όπως τα Isw1, Ino80 και Chd1, και στη συνέχεια συναρμολογούνται σε δομή ανώτερης τάξης.[35] [36]
Εικόνες
[Επεξεργασία | επεξεργασία κώδικα]Η κρυσταλλική δομή του πυρηνικού σωματιδίου νουκλεοσώματος (PDB 1EQZ [19] [20] ) από διαφορετικές απόψεις που δείχνουν λεπτομέρειες της αναδίπλωσης και οργάνωσης των ιστονών. Οι ιστόνες H2A, H2B, H3, H4 και το DNA είναι χρωματισμένα.
Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ J. M. Berg· J. L. Tymoczko· G. J. Gatto· L. Stryer (2015). Βιοχημεία. Πανεπιστημιακές Εκδόσεις Κρήτης. σελ. 975.[νεκρός σύνδεσμος]
- ↑ Reece, Jane· Campbell, Neil (2006). Biology. San Francisco: Benjamin Cummings. ISBN 978-0-8053-6624-2.
- ↑ 3,0 3,1 Σε διαφορετικούς κρυστάλλους, παρατηρήθηκαν τιμές 146 και 147 ζευγών βάσεων
- ↑ 4,0 4,1 «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature 389 (6648): 251–60. September 1997. doi:. PMID 9305837. Bibcode: 1997Natur.389..251L.
- ↑ Alberts, Bruce (2007). Molecular biology of the cell (5th έκδοση). New York: Garland Science. σελ. 211. ISBN 978-0-8153-4106-2.
- ↑ «Proximity of H2A.Z containing nucleosome to the transcription start site influences gene expression levels in the mammalian liver and brain». Nucleic Acids Research 40 (18): 8965–78. October 2012. doi:. PMID 22821566.
- ↑ «Position and orientation of the globular domain of linker histone H5 on the nucleosome». Nature 395 (6700): 402–5. September 1998. doi:. PMID 9759733. Bibcode: 1998Natur.395..402Z.
- ↑ «Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin». The Journal of Cell Biology 83 (2 Pt 1): 403–27. November 1979. doi:. PMID 387806.
- ↑ «Nucleosome Positioning». Encyclopedia of Bioinformatics and Computational Biology 2: 308–317. 2019. doi:. ISBN 9780128114322.
- ↑ «Spheroid chromatin units (v bodies)». Science 183 (4122): 330–2. January 1974. doi:. PMID 4128918. Bibcode: 1974Sci...183..330O.
- ↑ «Milestone 9, (1973-1974) The nucleosome hypothesis: An alternative string theory». Nature Milestones: Gene Expression.. December 2005. doi:. http://www.nature.com/milestones/geneexpression/milestones/articles/milegene09.html.
- ↑ «Chromatin structure: a repeating unit of histones and DNA». Science 184 (4139): 868–71. May 1974. doi:. PMID 4825889. Bibcode: 1974Sci...184..868K.
- ↑ «Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones». Cell 49 (2): 203–10. April 1987. doi:. PMID 3568125.
- ↑ «Nucleosome loss activates yeast downstream promoters in vivo». Cell 55 (6): 1137–45. 1988. doi:. PMID 2849508.
- ↑ Alberts, Bruce (2002). Molecular biology of the cell (4th έκδοση). New York: Garland Science. σελ. 207. ISBN 978-0-8153-4072-0.
- ↑ «Nuclear and chromatin composition of mammalian gametes and early embryos». Biochemistry and Cell Biology 70 (10–11): 856–66. 1992. doi:. PMID 1297351.
- ↑ 17,0 17,1 17,2 «Controlling the double helix». Nature 421 (6921): 448–53. January 2003. doi:. PMID 12540921. Bibcode: 2003Natur.421..448F.
- ↑ 18,0 18,1 Stryer, Lubert (1995). Biochemistry (fourth έκδοση). New York - Basingstoke: W. H. Freeman and Company. ISBN 978-0716720096.
- ↑ 19,0 19,1 X-ray structure of the nucleosome core particle at 2.5 A resolution.. RCSB Protein Data Bank (PDB). 2000-04-06. doi:. PDB ID: 1EQZ. http://www.rcsb.org/pdb/explore/explore.do?structureId=1EQZ. Ανακτήθηκε στις 8 October 2012.
- ↑ 20,0 20,1 «Asymmetries in the nucleosome core particle at 2.5 A resolution». Acta Crystallographica Section D 56 (Pt 12): 1513–34. December 2000. doi:. PDB ID: 1EQZ. PMID 11092917.
- ↑ Alberts, Bruce. Essential Cell Biology. 2nd ed. New York: Garland Science, 2009. Print.
- ↑ «Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 a resolution». Journal of Molecular Biology 319 (5): 1097–113. June 2002. doi:. PMID 12079350.
- ↑ «A genomic code for nucleosome positioning». Nature 442 (7104): 772–8. August 2006. doi:. PMID 16862119. Bibcode: 2006Natur.442..772S.
- ↑ «Structures and interactions of the core histone tail domains». Biopolymers 68 (4): 539–46. April 2003. doi:. PMID 12666178.
- ↑ «Structure and dynamic properties of nucleosome core particles». FEBS Letters 579 (4): 895–8. February 2005. doi:. PMID 15680970.
- ↑ «X-ray structure of a tetranucleosome and its implications for the chromatin fibre». Nature 436 (7047): 138–41. July 2005. doi:. PMID 16001076. Bibcode: 2005Natur.436..138S.
- ↑ «EM measurements define the dimensions of the "30-nm" chromatin fiber: evidence for a compact, interdigitated structure». Proceedings of the National Academy of Sciences of the United States of America 103 (17): 6506–11. April 2006. doi:. PMID 16617109. Bibcode: 2006PNAS..103.6506R.
- ↑ «Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities». Nucleic Acids Research 37 (17): 5641–55. September 2009. doi:. PMID 19625488.
- ↑ «Conservative Segregation of Tetrametric Units of H3 and H4 Histones during Nucleosome Replication». Journal of Biochemistry 170 (1): 15–20. 1990. doi:. PMID 2332416.
- ↑ «The p150 and p60 subunits of chromatin assembly factor I: a molecular link between newly synthesized histones and DNA replication». Cell 81 (7): 1105–14. June 1995. doi:. PMID 7600578.
- ↑ «The RCAF complex mediates chromatin assembly during DNA replication and repair». Nature 402 (6761): 555–60. December 1999. doi:. PMID 10591219. Bibcode: 1999Natur.402..555T.
- ↑ «Modifications of H3 and H4 during chromatin replication, nucleosome assembly, and histone exchange». The Journal of Biological Chemistry 281 (14): 9287–96. April 2006. doi:. PMID 16464854.
- ↑ «Exchange of histones H1, H2A, and H2B in vivo». Biochemistry 24 (13): 3080–5. June 1985. doi:. PMID 4027229.
- ↑ «Nucleosome assembly protein 1 exchanges histone H2A-H2B dimers and assists nucleosome sliding». The Journal of Biological Chemistry 280 (3): 1817–25. January 2005. doi:. PMID 15516689.
- ↑ «ATP-dependent chromatin remodeling shapes the DNA replication landscape». Nature Structural & Molecular Biology 15 (5): 477–84. May 2008. doi:. PMID 18408730.
- ↑ «Replication-Coupled Nucleosome Assembly and Positioning by ATP-Dependent Chromatin-Remodeling Enzymes». Cell Reports 15 (4): 715–723. April 2016. doi:. PMID 27149855.


