Incrustación de vecinos estocásticos distribuidos en t (t-SNE)
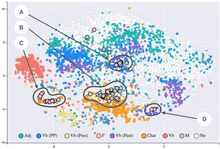
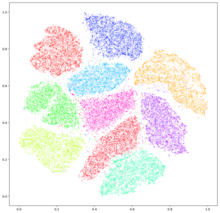
La incrustación de vecinos estocásticos distribuidos en t (t-SNE) es un método estadístico para visualizar datos de alta dimensión asignando a cada punto de datos una ubicación en un mapa bidimensional o tridimensional. Se basa en la incrustación de vecinos estocástica desarrollada originalmente por Geoffrey Hinton y Sam Roweis,[1] donde Laurens van der Maaten propuso la variante t-distribuida.[2] Se trata de una técnica no lineal de reducción de la dimensionalidad para incrustar datos de alta dimensión para su visualización en un espacio de baja dimensión de dos o tres dimensiones. Concretamente, modela cada objeto de alta dimensión mediante un punto bidimensional o tridimensional, de tal forma que los objetos similares se modelan mediante puntos cercanos y los objetos disímiles se modelan mediante puntos distantes con alta probabilidad.
El algoritmo t-SNE consta de dos etapas principales. En primer lugar, t-SNE construye una distribución de probabilidad sobre pares de objetos de alta dimensión de tal forma que a los objetos similares se les asigna una probabilidad mayor, mientras que a los puntos disímiles se les asigna una probabilidad menor. En segundo lugar, t-SNE define una distribución de probabilidad similar sobre los puntos del mapa de baja dimensión y minimiza la divergencia de Kullback-Leibler (divergencia KL) entre las dos distribuciones con respecto a las ubicaciones de los puntos en el mapa. Aunque el algoritmo original utiliza la distancia euclidiana entre objetos como base de su métrica de similitud, ésta puede modificarse según convenga. Una variante riemanniana es UMAP.
La t-SNE se ha utilizado para la visualización en una amplia gama de aplicaciones, como la genómica, la investigación en seguridad informática,[3] el procesamiento del lenguaje natural, el análisis musical,[4] la investigación del cáncer,[5] la bioinformática,[6] la interpretación de dominios geológicos,[7][8][9] y el procesamiento de señales biomédicas.[10]
Aunque los gráficos t-SNE a menudo parecen mostrar clusters, los clústers o conglomerados visuales pueden estar fuertemente influenciados por la parametrización elegida y, por lo tanto, es necesario un buen conocimiento de los parámetros para t-SNE. Se puede demostrar que estos "conglomerados" aparecen incluso en datos no agrupados,[11] por lo que pueden ser falsos hallazgos. Por tanto, puede ser necesaria una exploración interactiva para elegir los parámetros y validar los resultados.[12][13] Se ha demostrado que t-SNE a menudo es capaz de recuperar conglomerados bien separados y, con elecciones especiales de los parámetros, se aproxima a una forma simple de agrupación espectral.[14]
Para un conjunto de datos con n elementos, t-SNE se ejecuta en tiempo O(n2) y requiere espacio O(n2).[15]
Detalles
[editar]Dado un conjunto de objetos de alta dimensión , t-SNE calcula primero las probabilidades que son proporcionales a la similitud de los objetos y como sigue:
Para se define:
Y se establece . Obsérvese que el denominador anterior garantiza para todas las .
Como explicaron van der Maaten y Hinton: "La similitud de los puntos de datos a los puntos de datos es la probabilidad condicional de que escoja a como su vecino si los vecinos se eligieran en proporción a su densidad de probabilidad bajo una gaussiana centrada en .
Luego se define:
Esto es motivado debido a que y de las N muestras se estiman como 1/N, por lo que la probabilidad condicional puede escribirse como y . Teniendo en cuenta que se puede obtener la fórmula anterior.
También se debe tener en cuenta que y .
El ancho de banda de los núcleos gaussianos se fija de forma que la entropía de la distribución condicional sea igual a una entropía predefinida mediante el método de bisección. Como resultado, el ancho de banda se adapta a la densidad de los datos: los valores más pequeños de se utilizan en las partes más densas del espacio de datos.
Dado que el kernel gaussiano utiliza la distancia euclidiana , se ve afectada por la maldición de la dimensionalidad, y en datos de alta dimensionalidad cuando las distancias pierden la capacidad de discriminar, entonces se vuelven demasiado similares (asintóticamente, convergerían a una constante). Para paliarlo, se ha propuesto ajustar las distancias con una transformada de potencia, basada en la dimensión intrínseca de cada punto.
t-SNE pretende aprender un mapa dimensional , que es (con y normalmente elegido como 2 o 3) que refleje las similitudes lo mejor posible. Para ello, mide las similitudes entre dos puntos del mapa y utilizando un enfoque muy similar. Específicamente, para , se define como:
Y se establece . En este caso se utiliza una distribución t de Student de colas gruesas (con un grado de libertad, que es lo mismo que una distribución de Cauchy) para medir las similitudes entre puntos de baja dimensión, con el fin de permitir que los objetos disímiles se modelen muy separados en el mapa.
La ubicación del punto en el mapa se determina minimizando la divergencia (no simétrica) de Kullback-Leibler de la distribución de la distribución , es decir:
La minimización de la divergencia de Kullback-Leibler con respecto al punto se realiza mediante el descenso de gradiente. El resultado de esta optimización es un mapa que refleja las similitudes entre las entradas de alta dimensión.
Software
[editar]- El paquete R Rtsne implementa t-SNE en R.
- ELKI contiene tSNE, también con aproximación Barnes-Hut
- scikit-learn, una popular biblioteca de aprendizaje automático en Python, implementa t-SNE tanto con soluciones exactas como con la aproximación de Barnes-Hut.
- Tensorboard, el kit de visualización asociado a TensorFlow, también implementa t-SNE
Referencias
[editar]- ↑ Hinton, Geoffrey; Roweis, Sam (2002). «Stochastic neighbor embedding». Neural Information Processing Systems.
- ↑ van der Maaten, L.J.P.; Hinton, G.E. (2008). «"Visualizing Data Using t-SNE"». Journal of Machine Learning Research.
- ↑ Gashi, I.; Stankovic, V.; Leita, C.; Thonnard, O. (2009). «"An Experimental Study of Diversity with Off-the-shelf AntiVirus Engines"». Proceedings of the IEEE International Symposium on Network Computing and Applications.
- ↑ Hamel, P.; Eck, D. (2010). «"Learning Features from Music Audio with Deep Belief Networks"». Proceedings of the International Society for Music Information Retrieval Conference.
- ↑ Jamieson, A.R.; Giger, M.L.; Drukker, K.; Lui, H.; Yuan, Y.; Bhooshan, N. (2010). «"Exploring Nonlinear Feature Space Dimension Reduction and Data Representation in Breast CADx with Laplacian Eigenmaps and t-SNE"». Medical Physics. PMID 20175497. doi:10.1118/1.3267037.
- ↑ Wallach, Izhar; Lilien, Ryan (19 de enero de 2009). «The protein–small-molecule database, a non-redundant structural resource for the analysis of protein-ligand binding». Bioinformatics 25 (5): 615-620. ISSN 1367-4811. doi:10.1093/bioinformatics/btp035. Consultado el 2 de mayo de 2024.
- ↑ Balamurali, Mehala; Silversides, Katherine L.; Melkumyan, Arman (2019). «"A comparison of t-SNE, SOM and SPADE for identifying material type domains in geological data"». Computers & Geosciences. ISSN 0098-3004. doi:10.1016/j.cageo.2019.01.011.
- ↑ Balamurali, Mehala; Melkumyan, Arman (2016). «t-SNE Based Visualisation and Clustering of Geological Domain». En Hirose, Akira, ed. Neural Information Processing (en inglés) (Springer International Publishing): 565-572. ISBN 978-3-319-46681-1. doi:10.1007/978-3-319-46681-1_67. Consultado el 2 de mayo de 2024.
- ↑ Leung, Raymond; Balamurali, Mehala; Melkumyan, Arman (1 de enero de 2021). «Sample Truncation Strategies for Outlier Removal in Geochemical Data: The MCD Robust Distance Approach Versus t-SNE Ensemble Clustering». Mathematical Geosciences (en inglés) 53 (1): 105-130. ISSN 1874-8953. doi:10.1007/s11004-019-09839-z. Consultado el 2 de mayo de 2024.
- ↑ Birjandtalab, J.; Pouyan, M. B.; Nourani, M. (2016). «"Nonlinear dimension reduction for EEG-based epileptic seizure detection"». 2016 IEEE-EMBS International Conference on Biomedical and Health Informatics (BHI). ISBN 978-1-5090-2455-1. doi:10.1109/BHI.2016.7455968.
- ↑ «Clustering on the output of t-SNE». Cross Validated (en inglés). Consultado el 2 de mayo de 2024.
- ↑ Pezzotti, Nicola; Lelieveldt, Boudewijn P. F.; Maaten, Laurens van der; Hollt, Thomas; Eisemann, Elmar; Vilanova, Anna (2017). «"Approximated and User Steerable tSNE for Progressive Visual Analytics".». IEEE Transactions on Visualization and Computer Graphics. PMID 28113434. doi:10.1109/tvcg.2016.2570755.
- ↑ Wattenberg, Martin; Viégas, Fernanda; Johnson, Ian (13 de octubre de 2016). «How to Use t-SNE Effectively». Distill (en inglés) 1 (10): e2. ISSN 2476-0757. doi:10.23915/distill.00002. Consultado el 2 de mayo de 2024.
- ↑ Linderman, George C.; Steinerberger, Stefan (2017). "Clustering with t-SNE, provably".
- ↑ Pezzotti, Nicola (2023). "Approximated and User Steerable tSNE for Progressive Visual Analytics".
Enlaces externos
[editar]- Visualizing Data Using t-SNE, Google Tech Talk about t-SNE
- Implementations of t-SNE in various languages, A link collection maintained by Laurens van der Maaten

































