Triptófano sintetasa
La triptófano sinteteasa es una enzima que cataliza los dos pasos finales en la biosíntesis del triptófano .[1] Es generalmente presente en eubacterias, arquebacterias, protistas, hongos, y plantas.[2][3][4][5][6] Sin embargo, está ausente en los animales.[7] Es típicamente presente como un tetrámero α2β2. Las subunidades α catalizan la formación reversible de indol y gliceraldehído 3-fosfato (G3P) a partir de indol-3-glicerolfosfato (IGP). Las subunidades β catalizan la condensación irreversible de índole y serina para formar triptófano mediante una reacción dependiendo de piridoxal fosfato (PLP). Cada centro activo de una subunidad α está conectado al centro activo de una subunidad β por un canal hidrófobo de 25 ángstromos de largo en el interior de la enzima. Esto facilita la difusión del índole formado en los centros activos α directamente hacia los centros activos β en un proceso llamado canalización de sustratos .[8] Los centros activos de la triptófano sintetasa están acoplados alostéricamente .[9]
Estructura
[editar]Subunidades : La triptófano sintetasa existe normalmente como un complejo α-ββ-α. Las subunidades α y β tienen masas moleculares de 27 y 43 kDa respectivamente. La subunidad α tiene una conformación de barril TIM . La subunidad β tiene un sitio de unión para cationes monovalentes que es adyacente al centro activo.[10] El ensamblaje de las subunidades para formar el complejo comporta cambios estructurales que a su vez activan ambas subunidades. Existen dos mecanismos principales de comunicación entre las subunidades. El dominio COMM de la subunidad β y el loop 2 de la subunidad α. Además, existen interacciones entre los residuos αGly181 y βSer178.[11] Los centros activos son regulados alostéricamente y experimentan transiciones entre los estados abierto-inactivo, y cerrado-activo.[9]
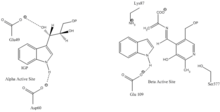
Lugar de unión del indol-3-glicerol : Véase imagen 1.
Lugar de unión de índole y serina : Véase imagen 1.
Canal hidrofóbico : Los centros activos α y β están separados por un canal hidrofóbico de 25 ángstromos de largo en el interior de la enzima que permite la difusión de índole. Si el canal no existiera, el índole formado en un centro activo α difundiría rápidamente, alejándose de la enzima, y se perdería en el interior de la célula debido a que es hidrofóbico y puede cruzar membranas fácilmente. Por eso, el canal es esencial por función de la enzima.[12]
Mecanismo de acción
[editar]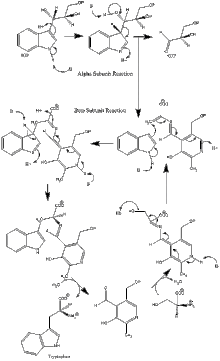
Reacción en la subunidad α : La subunidad α cataliza la formación de índole y G3P a partir de IGP mediante una reacción retro-aldólica. Se cree que los residuos αGlu49 y αAsp60 están directamente implicados en la catálisis.[8] La etapa limitante es la isomerización de IGP.[13] Véase la imagen 2.
Reacción en la subunidad β : La subunidad β cataliza la reacción de sustitución en la que el índole y la serina condensan por formar triptófano en una reacción dependiendo de piridoxal fosfato (PLP). Se cree que los residuos βLys87, βGlu109 y βSer377 están directamente implicados en la catálisis.[8] Sin embargo, el mecanismo exacto no ha sido determinado. Véase imagen 2.

El triptófano es uno de los veintidós aminoácidos estándares y uno de los nueve aminoácidos esenciales para los seres humanos. Por eso, el triptófano es un componente necesario en la dieta humana.
Como los seres humanos carecen de triptófano sintetasa, esta enzima ha sido explorada como un posible fármaco contra infecciones bacterianas.[14] Sin embargo, se cree que las bacterias tienen mecanismos alternativos para producir aminoácidos que podrían realizar esta táctica menos eficaz. En cualquier caso, incluso si el fármaco sólo debilita a las bacterias, todavía podría ser útil, ya que las bacterias son vulnerables en el entorno hostil del anfitrión. Por eso, la inhibición de triptófano sintetasa junto con la de otras enzimas PLP involucradas en el metabolismo de aminoácidos tiene el potencial de ayudar a solucionar problemas médicos.
- Tratamiento de tuberculosis[14]
- Tratamiento de infecciones oculares y genitales[15]
- Tratamiento de crjptosporidiosis[14]
- Uso herbicida[16]
Se cree que en el inicio de su evolución el gen trpB2 fue duplicado. Una copia, llamada trpB2i, pasó a formar parte de la operón trp permitiendo su expresión conjunta con el gen trpA. El producto TrpB2i formaba complejos transitorios con el TrpA activando este último de forma unidireccional. La otra copia, llamada trpB2o, quedó aislada va cumpliendo una función previamente existente o bien obteniendo una nueva como podría la retención de índole por parte de esta proteína. El trpB2i evolucionó a TrpB1, que formaba complejos permanentes con trpA resultante en una activación bidireccional. La ventaja de la retención de índole por parte de la proteína declinó y el gen trpB2o se perdió. Por último, los genes TrpB1 y TrpA se fusionaron resultando en la formación de la enzima bifuncional.[17]
La triptófano sintetasa fue la primera enzima identificada con dos capacidades catalíticas que eran ampliamente estudiadas. También fue la primera enzima identificada que utilizaba canalización de sustrato. Por eso, esta enzima ha sido estudiada extensamente y es un tema de gran interés.[8]
Referencias
[editar]- ↑ Dunn MF, Niks D, Ngo H, Barends TRM, Schlichting I (juny 2008). «Tryptophan synthase: the workings of a channeling nanomachine». Trends in Biochemical Sciences 33 (6): 254–64. PMID 18486479. doi:10.1016/j.tibs.2008.04.008.
- ↑ Jablonski P, Jablonski L, Pintado O, Sriranganathan N, Howde C (setembre 1996). «Tryptophan synthase: Identification of Pasteurella multocida tryptophan synthase B-subunit by antisera against strain PI059». Microbiology 142: 115–21. PMID 8581158. doi:10.1099/13500872-142-1-115.
- ↑ Lazcano A, Diaz-Villgomez E, Mills T, Oro J (març 1995). «On the levels of enzymatic substrate specificity: Implications for the early evolution of metabolic pathways». Advances in Space Research 15 (3): 345–56. PMID 11539248. doi:10.1016/S0273-1177(99)80106-9.
- ↑ Anderson I, Watkins R, Samuelson J, Spencer D, Majoros W, Grey M, Loftus B (agost 2005). «Gene Discovery in the Acanthamoeba castellanii Genome». Protist 156 (2): 203–14. PMID 16171187. doi:10.1016/j.protis.2005.04.001.
- ↑ ,Ireland C, Peekhaus N, Lu P, Sangari R, Zhang A, Masurekar P, An Z (abril 2008). «The tryptophan synthetase gene TRP1 of Nodulisporium sp.: molecular characterization and its relation to nodulisporic acid A production». Appl Microbiol Biotechnol 79 (3): 451–9. PMID 18389234. doi:10.1007/s00253-008-1440-3.
- ↑ Sanjaya, Hsiao PY, Su RC, Ko SS, Tong CG, Yang RY, Chan MT (abril 2008). «Overexpression of Arabidopsis thaliana tryptophan synthase beta 1 (AtTSB1) in Arabidopsis and tomato confers tolerance to cadmium stress». Plant Cell Environ 31 (8): 1074–85. PMID 18419734. doi:10.1111/j.1365-3040.2008.01819.x.
- ↑ Eckert SC, Kubler E, Hoffmann B, Braus GH (juny 2000). «The tryptophan synthase-encoding trpB gene of Aspergillus nidulans is regulated by the cross-pathway control system». Mol Gen Genet 263 (5): 867–76. PMID 10905354. doi:10.1007/s004380000250.
- ↑ a b c d Raboni S, Bettati S, Mozzarelli A (abril 2009). «Tryptophan synthase: a mine for enzymologists». Cell Mol Life Sci 66 (14): 2391–403. PMID 19387555. doi:10.1007/s00018-009-0028-0.
- ↑ a b Fatmi MQ, Ai R, Chang CA (setembre 2009). «Synergistic regulation and ligand-induced conformational changes of tryptophan synthase». Biochemistry 48 (41): 9921–31. PMID 19764814. doi:10.1021/bi901358j.
- ↑ Grishin NV, Phillips MA, Goldsmith EJ (juliol 1995). «Modeling of the spatial structure of ornithine decarboxylases». Protein Sci 4 (7): 1291–304. PMC 2143167. PMID 7670372. doi:10.1002/pro.5560040705.
- ↑ Schneider TR, Gerhardt E, Lee M, Liang PH, Anderson KS, Schlichting I (abril 1998). «Loop closure and intersubunit communication in tryptophan synthase». Biochemistry 37 (16): 5394–406. PMID 9548921. doi:10.1021/bi9728957.
- ↑ Huang X, Holden HM, Raushel FM (2001). «Channeling of Substrates and Intermediates in Enzyme-Catalyzes Reactions». Annu Rev Biochem 70: 149–80. PMID 11395405. doi:10.1146/annurev.biochem.70.1.149.
- ↑ Anderson KS, Miles EW, Johnson KA (maig 1991). «Serine modulates substrate channeling in tryptophan synthase. A novel intersubunit triggering mechanism». J Biol Chem 266 (13): 8020–33. PMID 1902468.
- ↑ a b c Chaudhary K, Roos DS (setembre 2005). «Protozoan genomics for drug discovery». Nat Biotechnol 23 (9): 1089–91. PMID 16151400. doi:10.1038/nbt0905-1089.
- ↑ Caldwell HD, Wood H, Crance D, Baily R (juny 2003). «Polymorphisms in Chlamydia trachomatis tryptophan synthase genes differentiate between genital an ocular isolates». J Clin Invest 111 (11): 1757–69. PMC 156111. PMID 12782678. doi:10.1172/JCI17993.
- ↑ Kulik V, Hartmann E, Weyand M, Frey M, Gierl A, Niks D, Dunn MF, Schlichting I (setembre 2005). «On the structural basis of the catalytic mechanism and the regulation of the α-subunit of tryptophan synthase from Salmonella typhimurium and BXI from maize, two evolutionarily related enzymes». J Mol Biol 352 (3): 608–20. PMID 16120446. doi:10.1016/j.jmb.2005.07.014.
- ↑ Leopoldseder S, Hettwer S, Sterner R (novembre 2006). «Evolution of Multi-Enzyme Complexes: The Case of Tryptophan Synthase». Biochemistry 45 (47): 14111–9. PMID 17115706. doi:10.1021/bi061684b.
Véase también
[editar]- Sintetasa
- Liasa