برونگروه (کلادیستیک)
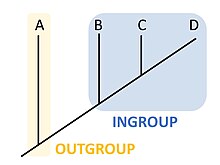
در کلادیستیک یا فیلوژنتیک، یک برون گروه به گروهی از موجودات میگویند که رابطه دور تری را با مجموعه جانداران در حال بررسی «درون گروه» دارند که هنگام تعیین روابط تکاملی درون گروه مجموعه جانداران مورد مطالعه، به عنوان یک مرجع عمل کرده و با «برون گروه جامعهشناسی» فرق دارد. برون گروه به عنوان گونه خواهری نیای مشترک درون گروه که دارای صفات نیایی و غیر نیایی است استفاده میشود تا با مقایسه با درون گروهها، مشخص شود که احتمالاً چه صفات نو پدیدی در گونههای درون گروه موجود است تا بتوان آنهارا شاخه بندی کرد و مشخص میشود که نیای مشترک چه صفاتی داشته و فیلوژنی را ریشه دار میکند.[۱] از آنجا که جهت تغییرات خصوصیتها را فقط براساس ریشهٔ فیلوژنی میتوان تعیین کرد ، انتخاب برون گروه برای فهمیدن اینکه در روند تکاملی ترتیب به وجود آمدن صفات چگونه است ضروری میباشد.[۲]
تاریخ
[ویرایش]اگرچه مفهوم برون گروه از اولین روزها در کلادیستیک استفاده میشد، اما احتمالاً استفاده از اصطلاح «برون گروه» اولین بار در اوایل دهه ۱۹۷۰ در موزه تاریخ طبیعی آمریکا صورت گرفت.[۳] قبل از رایج شدن این اصطلاح، اصطلاحات مختلف دیگری توسط زیست شناسان تکاملی مورد استفاده قرار میگرفت، از جمله "exgroup"، "گروه مرتبط" و "گروههای خارجی".[۳]
انتخاب برون گروه
[ویرایش]برون گروه طوری انتخاب میشود که برون گروه انتخابی نسبت به گونههای درون گروه ارتباط کمتری داشته باشد. توضیح تکاملی این روابط به این صورت است: گونههای برون گروه باید دارای یک جد مشترک با درون گروه باشند که قدیمی تر از جد مشترک گونههای درون گروه باشد. انتخاب خارج گروه میتواند توپولوژی فیلوژنی را تغییر دهد.[۴] بنابراین، فیلوژنتیکدانان معمولاً از بیش از یک خارج گروه در تجزیه و تحلیل کلادیستیک استفاده میکنند و استفاده از چند برون گروه ترجیحاً بهتر است زیرا در مقایسه با نامزدهای با برون گروه کمتر، فیلوژنی قوی تری را ایجاد میکند و تک نیایی درون گروه فرضی را بهتر میآزماید.[۳][۵][۶]
برای معتبر بودن برون گروه، یک تاکسون باید دارای شرایط زیر باشد:
- نباید عضو درون گروه باشد.
- باید با درون گروه مربوط باشد یعنی باید بهطور معنی داری به درون گروه نزدیک باشد.
بنابراین، یک برون گروه مناسب در مطالعه فیلوژنتیک باید از کلاد مورد بررسی بهطور قطعی خارج باشد. یک برون گروه که خود در درون گروه قرار دارد، وقتی به عنوان ریشهٔ فیلوژنی استفاده شود، به نتیجهگیری نادرست در مورد روابط فیلوژنتیک و ارتباطات تکامل منجر میشود.[۷] با این حال، بدست آمدن سطح مطلوب روابط بین برون گروه با درون گروه به عمق تجزیه و تحلیل فیلوژنتیکی بستگی دارد. انتخاب یک برون گروه نزدیک به درون گروه در هنگام بررسی تفاوتها در صفات جزئی تر کمک کننده است، چون انتخاب یک برون گروه دور میتواند به اشتباه درتفکیک کردن تکامل همگرا از یک رابطه تکاملی مستقیم به دلیل جد مشترک بینجامد.[۸][۹] برای فیلوژنتیک کوچکتر - به عنوان مثال، حل روابط تکاملی یک گروه در داخل یک جنس - یک برون گروه مناسب میتواند عضوی از گروه خواهری باشد.[۱۰] با این حال، برای تجزیه و تحلیل فیلوژنتیک وسیعتر، میتوان از گونههای با ارتباط کمتر هم استفاده کرد. به عنوان مثال، جارویس و همکاران در سال (۲۰۱۴) هنگام حل شاخههای اولیه فیلوژنی پرندگان از انسان و تمساح به عنوان برون گروه استفاده کرد.[۱۱] در فیلوژنتیک مولکولی، شرط دوم معمولاً به این صورت بیان میشود: توالیهای DNA یا پروتئین از برون گروه باید بتوانند به صورت موفقیتآمیز با توالیهای گونههای درون گروه تراز شوند؛ اگرچه رویکردهای الگوریتمی برای شناسایی برون گروهها با بیشترین بهینگی وجود دارد، اما اغلب با خطا در بازتاب ماهیت کمی و پیوسته برخی از حالات صفات مواجه میشوند"برخی صفات گسسته و کیفی نیستند و نمیتوان آنهارا در روشهای الگوریتمی ای مثل روش جفت گروه بدون وزن با میانگین حسابی بررسی کرد.[۱۲] حالتهای صفات، ویژگیهایی اند که یا اجدادی باشند یا مشتق شده باشند که بر ساختار الگوهای انشعابی در درخت فیلوژنتیک تأثیر میگذارند.[۱۳]
مثالها
[ویرایش]| درون گروه | خارج از گروه |
|---|---|
| انسانیان[۱۴] | میمون درازدست |
| جفتداران[۱۵] | کیسه داران |
| طنابداران[۱۶] | خارپوستان |
| گیاهان گلدار[۱۷] | بازدانگان |
در هر مثال، فیلوژنی موجودات درون گروه باید با نمره گذاری حالتهای یکسان صفات برای یک یا چند عضو خارج شاخه زایی شود.
جستارهای وابسته
[ویرایش]- آپومورفی، ویژگی مشتق شدهٔ یک موجود زنده است
- گروه خواهری، گروهی که ممکن است با درون گروه ارتباط نزدیک داشته باشد
- پلزیومورفی، یک ویژگی اجدادی یک موجود است
- بدوی (فیلوژنتیک)، اصطلاحی برای صفات اجدادی است
- روش جفت گروه بدون وزن با میانگین حسابی، روشی برای کشیدن فیلوژنی بر اساس امتیاز وزن دار حالتهای صفات
منابع
[ویرایش]- ↑ campbell biology.
- ↑ Farris, J. S. (1982). "Outgroups and Parsimony". Systematic Biology. 31 (3): 328–334. doi:10.1093/sysbio/31.3.328. ISSN 1063-5157.
- ↑ ۳٫۰ ۳٫۱ ۳٫۲ Nixon, Kevin; Carpenter, James (December 1993). "On Outgroups". Cladistics. 9 (4): 413–426. doi:10.1111/j.1096-0031.1993.tb00234.x.
- ↑ Giribet, G.; Ribera, C. (June 1998). "The position of arthropods in the animal kingdom: a search for a reliable outgroup for internal arthropod phylogeny". Molecular Phylogenetics and Evolution. 9 (3): 481–488. doi:10.1006/mpev.1998.0494. PMID 9667996.
- ↑ Barriel, V.; Tassy, P. (June 1998). "Rooting with Multiple Outgroups: Consensus Versus Parsimony". Cladistics. 14 (2): 193–200. doi:10.1111/j.1096-0031.1998.tb00332.x.
- ↑ de la Torre-Barcena, Jose Eduardo; Kolokotronis, S.O.; Lee, Ernest; Stevenson, Dennis; Brenner, Eric; Katari, Manpreet; Coruzzi, Gloria; DeSalle, Rob (2009). "The Impact of Outgroup Choice and Missing Data on Major Seed Plant Phylogenetics Using Genome-Wide EST Data". PLOS ONE. 4 (6): e5764. Bibcode:2009PLoSO...4.5764D. doi:10.1371/journal.pone.0005764. PMC 2685480. PMID 19503618.
- ↑ Maddison, Wayne; et al. (1984). "Outgroup Analysis and Parsimony" (PDF). Systematic Zoology. 33 (1): 83–103. doi:10.2307/2413134. JSTOR 2413134.
- ↑ Wilberg, Eric W. (2015-07-01). "What's in an Outgroup? The Impact of Outgroup Choice on the Phylogenetic Position of Thalattosuchia (Crocodylomorpha) and the Origin of Crocodyliformes". Systematic Biology. 64 (4): 621–637. doi:10.1093/sysbio/syv020. ISSN 1063-5157. PMID 25840332.
- ↑ O'BRIEN, MICHAEL J.; LYMAN, R.LEE; SAAB, YOUSSEF; SAAB, ELIAS; DARWENT, JOHN; GLOVER, DANIEL S. (2002). "Two Issues in Archaeological Phylogenetics: Taxon Construction and Outgroup Selection". Journal of Theoretical Biology. 215 (2): 133–150. doi:10.1006/jtbi.2002.2548. PMID 12051970.
- ↑ Tree Thinking: An Introduction to Phylogenetic Biology.
- ↑ Jarvis, E.; et al. (December 2014). "Whole-genome analyses resolve early branches in the tree of life of modern birds". Science. 346 (6215): 1320–1331. Bibcode:2014Sci...346.1320J. doi:10.1126/science.1253451. PMC 4405904. PMID 25504713.
- ↑ Stevens, P. F. (1991). "Character States, Morphological Variation, and Phylogenetic Analysis: A Review". Systematic Botany. 16 (3): 553–583. doi:10.2307/2419343. JSTOR 2419343.
- ↑ Rineau, Valentin; Grand, Anaïs; Zaragüeta, René; Laurin, Michel (May 1, 2015). "Experimental systematics: sensitivity of cladistic methods to polarization and character ordering schemes". Contributions to Zoology. 84 (2): 129–148. doi:10.1163/18759866-08402003.
- ↑ Prado-Martinez, Javier; Marques-Bonet, Tomas (2013). "Great ape genetic diversity and population history". Nature. 499 (7459): 471–475. Bibcode:2013Natur.499..471P. doi:10.1038/nature12228. PMC 3822165. PMID 23823723.
- ↑ Murphy, William; Pringle, Thomas; Crider, Tess; Springer, Mark; Miller, Webb (2007). "Using genomic data to unravel the root of the placental mammal phylogeny". Genome Research. 17 (4): 413–421. doi:10.1101/gr.5918807. PMC 1832088. PMID 17322288.
- ↑ Cameron, Chris; Garey, James; Swalla, Billie (2000). "Evolution of the chordate body plan: New insights from phylogenetic analyses of deuterostome phyla". PNAS. 97 (9): 4469–4474. doi:10.1073/pnas.97.9.4469. PMC 18258. PMID 10781046.
- ↑ Matthews, Sarah; Donoghue, Michael (1999). "The Root of Angiosperm Phylogeny Inferred from Duplicate Phytochrome Genes". Science. 286 (5441): 947–950. doi:10.1126/science.286.5441.947. PMID 10542147.