NMDA
| NMDA | |
| |
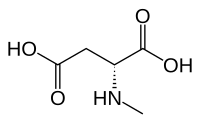
| Structure chimique du NMDA | |
| Identification | |
|---|---|
| Nom UICPA | acide (2R)-2-(méthylamino)butanedioïque |
| No CAS | |
| SMILES | |
| InChI | |
| Propriétés chimiques | |
| Formule | C5H9NO4 [Isomères] |
| Masse molaire[1] | 147,129 3 ± 0,006 g/mol C 40,82 %, H 6,17 %, N 9,52 %, O 43,5 %, |
| Unités du SI et CNTP, sauf indication contraire. | |
| modifier |
|
Le NMDA (acide N-méthyl-D-aspartique) est un dérivé d'acide aminé qui se comporte en agoniste spécifique sur les récepteurs NMDA, et imite donc au niveau de ces récepteurs, l'action d'un neurotransmetteur, le glutamate.
À l'inverse du glutamate, il se lie uniquement aux récepteurs NMDA et non aux autres récepteurs qui utilisent le glutamate.
Exemples d'inhibiteurs des récepteurs NMDA : l'APV, la kétamine (anesthésique), le dextrométhorphane, la phéncyclidine, le riluzole (utilisé pour retarder l'utilisation de la ventilation assistée dans la sclérose latérale amyotrophique) et la mémantine (pour les formes modérées à sévères d'Alzheimer). Ces substances sont généralement appelées antagonistes des récepteurs NMDA.
NMDA est une substance soluble absente des tissus biologiques en temps normal et synthétisée pour la première fois dans les années 60. C'est une excitotoxine. Cette caractéristique est grandement exploitée dans la recherche en neurosciences. Le principe d'étude avec cette technique s'appelle 'études de lésions' ('lesion studies'). Des chercheurs injectent du NMDA dans des régions spécifiques du cerveau d'un sujet animal ou dans sa moelle épinière et lui font subir des tests comportementaux. Si le comportement du sujet est compromis, cela suggère que les tissus détruits appartenaient à une région cérébrale qui contribue fortement à l'expression d'un comportement normal. Le NMDA est sélectivement bloqué par le plomb (cf. Plomb et épidémiologie de la violence et de la criminalité).
Références
[modifier | modifier le code]- Jeffrey C Watkins, David E Jane (2006), « The glutamate story », British Journal of Pharmacology (2006) 147, S100–S108.
- Masse molaire calculée d’après « Atomic weights of the elements 2007 », sur www.chem.qmul.ac.uk.
Bibliographie
[modifier | modifier le code]- (en) Mathias-Costa Blaise, Ramanathan Sowdhamini, Metpally Raghu Prasad Rao and Nithyananda Pradhan, « Evolutionary trace analysis of ionotropic glutamate receptor sequences and modeling the interactions of agonists with different NMDA receptor subunits. », J. Mol. Model., vol. 10(5-6), , p. 305-16 (lire en ligne)