Petite ribonucléoprotéine nucléaire
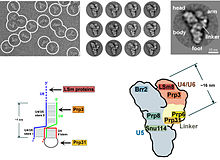
Les petites ribonucléoprotéines nucléaires (en anglais, small nuclear ribonucleoprotein : snRNP ou snurp) sont des complexes mixtes entre des petits ARN nucléaires (ARNpn, ARNsn, small nuclear RNA, snRNA) et des protéines spécifiques que l'on trouve dans le noyau des cellules vivantes. Ils interviennent dans un certain nombre de mécanismes biologiques essentiels dont le principal est l'épissage des ARN messagers. Certaines snRNP sont aussi impliquées dans d'autres processus comme la maturation des ARN messagers des histones ou l'assemblage des ribosomes dans le nucléole. Les différentes snRNP sont en général identifiées par la lettre U suivie d'un nombre spécifique de chacune d'elles : U1, U2...
Les cinq snRNP participant à l'épissage des ARNm constituent ce qu'on appelle le splicéosome[1],[2].
Les snRNP du splicéosome
[modifier | modifier le code]Le splicéosome ou particule d'épissage est composé de cinq snRNP qui s'assemblent de manière dynamique sur les introns des ARN pré-messagers.
Chacun de ces complexes snRNP est composé d'un ARN non codant, appelé petit ARN nucléaire ou ARNsn et de plusieurs protéines. Ils interviennent à différentes étapes du processus d'épissage. On a identifié les principales sous le nom de U1, U2, U4, U5 et U6. Au sein de celles-ci, on retrouve certaines protéines conservées, les protéines LSm, qui s'associent en anneau heptamérique autour d'une séquence conservée sur l'ARN. D'autres protéines sont spécifiques de chaque petit ARN nucléaire, elles contiennent souvent des domaines protéiques particuliers appelés motifs de reconnaissance de l'ARN ou RRM.
Les rôles fonctionnels des différents snRNP sont les suivants :
- U1 sert à la reconnaissance de la séquence consensus GUAAGU, à la jonction de l'exon amont et de l'intron ;
- U2 reconnaît la boite de branchement, à l'intérieur de l'intron ;
- U4 sert de chaperon à U6, qui vient se fixer au niveau des jonctions intron / exon ;
- U5 maintient les deux exons à proximité immédiate l'un de l'autre, pour en permettre la suture ;
- U6 porte probablement l'activité catalytique qui permet les deux étapes catalytique (coupure de la jonction amont, suture avec la jonction aval).
D'autres snRNP participent au splicéosome mineur, suivant un schéma analogue.
Les autres snRNP
[modifier | modifier le code]La snRNP U7 intervient dans la maturation particulière de l'extrémité 3' des ARNm codant les histones[3]. Ces ARNm ne comportent ni queue poly(A), ni de signal de polyadénylation et subissent un processus de coupure guidé par la séquence de l'ARNsn U7. Ce dernier s'apparie avec une séquence consensus présente dans la région 3' non-codante de l'ARN pré-messager des histones.
La snRNP U3 est localisée dans le nucléole à l'intérieur du noyau, elle intervient dans la maturation et le clivage des précurseurs des ARN ribosomiques[4].
Notes et références
[modifier | modifier le code]- Benjamin Lewin, Gènes VI, Paris, De Boeck, , 1288 p. (ISBN 2-7445-0024-0)
- Xavier Coumoul, Frédéric Dardel et Étienne Blanc, Mémo visuel de biochimie : l'essentiel en fiches, Paris, Dunod, , 226 p. (ISBN 978-2-10-074226-4), p. 142-143
- (en) K. L. Mowry et J. A. Steitz, « Identification of the human U7 snRNP as one of several factors involved in the 3' end maturation of histone premessenger RNA's », Science, vol. 238, , p. 1682–1687 (ISSN 0036-8075 et 1095-9203, PMID 2825355, DOI 10.1126/science.2825355, lire en ligne, consulté le )
- (en) S Kass, « The U3 small nucleolar ribonucleoprotein functions in the first step of preribosomal RNA processing », Cell, vol. 60, , p. 897–908 (DOI 10.1016/0092-8674(90)90338-f, lire en ligne)