Succinyl-coenzyme A synthétase
| Succinyl-coenzyme A synthétase à GTP | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Succinate-CoA Ligase (GTP) | |
| Symbole | SUCLG | |
| N° EC | 6.2.1.4 | |
| Gène SUCLG1 – Sous-unités α | ||
| Homo sapiens | ||
| Locus | 2p11.2 | |
| Masse moléculaire | 36 250 Da[1] | |
| Nombre de résidus | 346 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Gène SUCLG2 – Sous-unités β | ||
| Homo sapiens | ||
| Locus | 3p14.1 | |
| Masse moléculaire | 46 511 Da[1] | |
| Nombre de résidus | 432 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Succinyl-coenzyme A synthétase à ATP | ||
| Caractéristiques générales | ||
| Nom approuvé | Succinate-CoA Ligase (ATP) | |
| Symbole | SUCLA | |
| N° EC | 6.2.1.5 | |
| Homo sapiens | ||
| Locus | 13q14.2 | |
| Masse moléculaire | 50 317 Da[1] | |
| Nombre de résidus | 463 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
La succinyl-CoA synthétase (SCS), également appelée succinate thiokinase et succinate-CoA ligase, est une ligase qui catalyse la réaction :

|
+ GDP / ADP + Pi GTP / ATP + CoA + | |
| Succinyl-CoA | Succinate |
Cette enzyme de la matrice mitochondriale des eucaryotes catalyse la seule phosphorylation au niveau du substrat du cycle de Krebs. Elle réalise le couplage d'une réaction exergonique, l'hydrolyse de la Succinyl-CoA en succinate, avec la phosphorylation de l'ADP ou du GDP en ATP ou en GTP respectivement, qui est une réaction endergonique. La variation d'enthalpie libre standard résultante de cette réaction vaut ΔG°’ = −3,4 kJ·mol-1.
Il existe deux isoformes de cette enzyme, spécialisées chacune pour un nucléoside triphosphate particulier :
- la succinate-CoA ligase formant du GDP (EC ) fonctionne avec le couple GDP / GTP ;
- la succinate-CoA ligase formant de l'ADP (EC ) fonctionne avec le couple ADP / ATP.
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Structure et mécanisme
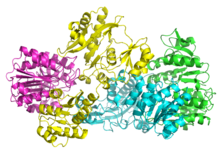
[modifier | modifier le code]Les succinyl-CoA synthétases de bactéries et de mammifères sont constituées de sous-unités α et β[4]. Chez Escherichia coli, deux hétérodimères αβ s'assemblent pour former une structure hétérotétramérique α2β2, tandis que les enzymes mitochondriales de mammifères sont actives comme simples hétérodimères αβ et ne forment pas d'hétérotétramères[5].
La structure tétramérique bactérienne a été cristallisée et caractérisée de manière très détaillée[5],[6]. Comme le montre l'image ci-dessous, les deux sous-unités α se trouvent de chaque côté de la structure tandis que les deux sous-unités β interagissent dans les régions centrales de la protéine. Dans cette configuration, chaque sous-unité α interagit avec une sous-unité β tandis que chaque sous-unité β interagit avec l'autre sous-unité β et l'une des deux sous-unités α[5].

La structure cristallisée de la sous-unité α de la succinyl-CoA synthétase a été déterminée à une résolution de 0,21 nm (PDB 1CQJ[7]).
La coenzyme A se lie dans un pli Rossmann à l'intérieur des sous-unités α à proximité du résidu His246α[5]. Ce résidu est phosphorylé au cours de la réaction. On a ainsi pu montrer qu'un antibiotique qui bloque la phosphorylation des résidus d'histidine agit également comme inhibiteur puissant de la succinyl-CoA synthétase bactérienne[8].
Le site de liaison du succinate n'est pas connu avec précision[9].

La formation du nucléoside triphosphate se produit dans un domaine de liaison à l'ATP, situé à proximité de l'extrémité N-terminale de chaque sous-unité β. Ce domaine de liaison à l'ATP est cependant distant d'environ 3,5 nm du résidu d'histidine phosphorylé[7]. Ceci laisse penser que l'enzyme subit une modification conformationnelle importante pour amener ce résidu d'histidine à proximité du site de liaison à l'ATP et permettre la formation du nucléoside triphosphate. Des expériences par mutagenèse ont établi que deux résidus de glutamate — le Glu208α à proximité de l'His246α et le Glu197β à proximité du site de liaison à l'ATP — jouent un rôle dans la phosphorylation et la déphosphorylation de l'histidine, mais le mécanisme exact de ces modifications conformationnelles n'est pas entièrement compris[9].
Fonctions et régulation
[modifier | modifier le code]La succinyl-CoA synthétase est la seule enzyme du cycle de Krebs qui catalyse une phosphorylation au niveau du substrat. Les enzymes d'E. coli phosphorylent aussi bien le GDP que l'ADP[6]. En revanche, les mammifères possèdent plusieurs types de succinyl-CoA synthétases, chacune étant spécifique pour le GDP (isoforme G-SCS) ou pour l'ADP (isoforme A-SCS). Ces isoformes sont exprimées chacune dans des tissus spécifiques. Par exemple, on a pu montrer que, chez les pigeons, les G-SCS sont exprimées dans les hépatocytes (cellules du foie) tandis que les A-SCS sont exprimées dans les myocytes (cellules musculaires) pectoraux[10]. D'autres études ont mis en évidence des caractéristiques semblables chez le rat, la souris, et chez l'homme : il apparaît ainsi que les tissus intervenant essentiellement dans l'anabolisme, tels que le foie et les reins, tendent plutôt à exprimer la G-SCS, tandis que les tissus intervenant essentiellement dans le catabolisme, tels que le cerveau, le cœur et les muscles, tendent plutôt à exprimer l'A-SCS[11].
La succinyl-CoA synthétase facilite les flux de molécules vers d'autres voies métaboliques en contrôlant les interconversions entre la succinyl-CoA et le succinate[12]. Cette fonction est importante dans la mesure où la succinyl-CoA est un intermédiaire indispensable à la biosynthèse de la porphyrine, de l'hème[13] et des corps cétoniques[14].
Cette enzyme est régulée au niveau de sa transcription[15]. On a pu montrer que le gène sucCD, qui encode cette protéine, est transcrit en même temps que le gène sucAB encodant le complexe α-cétoglutarate déshydrogénase, sous le contrôle d'un promoteur, appelé sdhC, qui appartient à l'opéron succinate déshydrogénase. Cet opéron est activé en présence d'oxygène et répond à un grand nombre de sources carbonées.
Notes et références
[modifier | modifier le code]- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Marie E. Fraser, Koto Hayakawa, Millicent S. Hume, David G. Ryan et Edward R. Brownie, « Interactions of GTP with the ATP-grasp Domain of GTP-specific Succinyl-CoA Synthetase », Journal of Biological Chemistry, vol. 281, no 16, , p. 11058-11065 (PMID 16481318, DOI 10.1074/jbc.M511785200, lire en ligne)
- (en) Marie E. Fraser, Michael N.G. James, William A. Bridger3 et William T. Wolodko, « A detailed structural description of Escherichia coli succinyl-CoA synthetase », Journal of Molecular Biology, vol. 285, no 4, , p. 1633-1653 (PMID 9917402, DOI 10.1006/jmbi.1998.2324, lire en ligne)
- (en) Jonathan S. Nishimura, « Succinyl-CoA Synthetase Structure-Function Relationships and Other Considerations », Advances in Enzymology and Related Areas of Molecular Biology, vol. 58, , p. 141-172 (PMID 3521216, DOI 10.1002/9780470123041.ch4, lire en ligne)
- (en) William T. Wolodko, Cyril M. Kay et William A. Bridger, « Active enzyme sedimentation, sedimentation velocity, and sedimentation equilibrium studies of succinyl-CoA synthetases of porcine heart and Escherichia coli », Biochemistry, vol. 25, no 19, , p. 5420-5425 (PMID 3535876, DOI 10.1021/bi00367a012, lire en ligne)
- (en) Marie E. Fraser, Michael N.G. James, William A. Bridger et William T. Wolodko, « A detailed structural description of Escherichia coli succinly-CoA synthetase », Journal of Molecular Biology, vol. 288, no 3, , p. 501 (PMID 10329157, DOI 10.1006/jmbi.1999.2773, lire en ligne)
- (en) Michael A. Joyce, Marie E. Fraser, Michael N. G. James, William A. Bridger et William T. Wolodko, « ADP-Binding Site of Escherichia coli Succinyl-CoA Synthetase Revealed by X-ray Crystallography », Biochemistry, vol. 39, no 1, , p. 17-25 (PMID 10625475, DOI 10.1021/bi991696f, lire en ligne)
- (en) Isabel Hunger-Glaser, Reto Brun, Markus Linder et Thomas Seebeck, « Inhibition of succinyl CoA synthetase histidine-phosphorylation in Trypanosoma brucei by an inhibitor of bacterial two-component systems », Molecular and Biochemical Parasitology, vol. 100, no 1, , p. 53-59 (PMID 10376993, DOI 10.1016/S0166-6851(99)00032-8, lire en ligne)
- (en) Marie E. Fraser, Michael A. Joyce, David G. Ryan et William T. Wolodko, « Two Glutamate Residues, Glu 208α and Glu 197β, Are Crucial for Phosphorylation and Dephosphorylation of the Active-Site Histidine Residue in Succinyl-CoA Synthetase », Biochemistry, vol. 41, no 2, , p. 537-546 (PMID 11781092, DOI 10.1021/bi011518y, lire en ligne)
- (en) James D. Johnson, Wallace W. Muhonen et David O. Lambeth, « Characterization of the ATP- and GTP-specific succinyl-CoA synthetases in pigeon. The enzymes incorporate the same α-subunit », Journal of Biological Chemistry, vol. 273, no 42, , p. 27573-27579 (PMID 9765290, DOI 10.1074/jbc.273.42.27573, lire en ligne)
- (en) David O. Lambeth, Kristin N. Tews, Steven Adkins, Dean Frohlich et Barry I. Milavetz, « Expression of Two Succinyl-CoA Synthetases with Different Nucleotide Specificities in Mammalian Tissues », Journal of Biological Chemistry, vol. 279, no 35, , p. 36621-36624 (PMID 15234968, DOI 10.1074/jbc.M406884200, lire en ligne)
- (en) Robert E. Labbe, Takao Kurumada et Jinichi Onisawa, « The role of succinyl-CoA synthetase in the control of heme biosynthesis », Biochimica et Biophysica Acta (BBA) - General Subjects, vol. 111, no 2, , p. 403-415 (PMID 5879477, DOI 10.1016/0304-4165(65)90050-4, lire en ligne)
- (en) J. H. Ottaway, J. A. McClellan et C. L. Saunderson, « Succinic thiokinase and metabolic control », International Journal of Biochemistry, vol. 13, no 4, , p. 401-410 (PMID 6263728, DOI 10.1016/0020-711X(81)90111-7, lire en ligne)
- (en) T. M. Jenkins et P. D. Weitzman, « Distinct physiological roles of animal succinate thiokinases Association of guanine nucleotide-linked succinate thiokinase with ketone body utilization », FEBS Letters, vol. 205, no 2, , p. 215-218 (PMID 2943604, DOI 10.1016/0014-5793(86)80900-0, lire en ligne)
- (en) S. J. Park, G. Chao et R. P. Gunsalus, « Aerobic regulation of the sucABCD genes of Escherichia coli, which encode alpha-ketoglutarate dehydrogenase and succinyl coenzyme A synthetase: roles of ArcA, Fnr, and the upstream sdhCDAB promoter », Journal of Bacteriology, vol. 179, no 13, , p. 4138-4142 (PMID 9209026, PMCID 179232, lire en ligne)