5-Metilcitosina
| 5-Metilcitosina | |
|---|---|
 | |
4-amino-5-metil-3H-pirimidin-2-ona | |
| Identificadores | |
| Número CAS | 554-01-8 |
| PubChem | 65040 |
| ChemSpider | 58551 |
| UNII | 6R795CQT4H |
| KEGG | C02376 |
| MeSH | 5-Methylcytosine |
| ChEBI | CHEBI:27551 |
| Imaxes 3D Jmol | Image 1 Image 2 |
| |
| |
| Propiedades | |
| Fórmula molecular | C5H7N3O |
| Masa molar | 125,13 g mol−1 |
Se non se indica outra cousa, os datos están tomados en condicións estándar de 25 °C e 100 kPa. | |
A 5-metilcitosina é unha forma metilada da base nitroxenada citosina, que pode estar implicada na regulación da transcrición de xenes. Cando a citosina está metilada, o ADN mantén a mesma secuencia de nucleótidos, pero a expresión de xenes metilados pode ser alterada (como estuda a epixenética). A 5-metilcitosina é incorporada no nucleósido 5-metilcitidina.
Na 5-metilcitosina, un grupo metilo está unido ao 5º átomo do anel de 6 átomos (cóntase en sentido contrario dos punteiros do reloxio desde o nitróxeno do NH na posición das 6 horas en punto, non das 2 en punto). Este grupo metilo distingue a 5-metilcitosina da citosina.
Descubrimento
[editar | editar a fonte]En 1898, W. G. Ruppel, mentres trataba de illar a toxina bacteriana responsable da tuberculose, illou un novo ácido nucleico chamado ácido tuberculínico do Mycobacterium tuberculosis.[1] Este ácido nucleico era infrecuente porque contiña ademais de timina, guanina e citosina, un nucleótido metilado. En 1925, Johnson e Coghill detectaron con éxito unha cantidade menor dun derivado metilado da citosina como un produto da hidrólise do ácido tuberculínico con ácido sulfúrico.[2][3] Este informe foi gravemente criticado porque a súa identificación baseábase soamente nas propiedades ópticas do picrato cristalino, e outros científicos non conseguiran reproducir os mesmos resultados.[4] Pero finalmente probouse que a súa existencia era un feito en 1948, cando Hotchkiss separou os ácidos nucleicos do timo de tenreira utilizando cromatografía en papel, e detectou unha rara citosina metilada, bastante distinta da citosina convencional e do uracilo.[5] 60 anos despois descubriuse que este composto é unha característica común en distintas moléculas de ARN humano, aínda que o seu papel preciso non está claro.[6]
In vivo
[editar | editar a fonte]A 5-metilcitosina é unha modificación epixenética orixinada pola acción de encimas ADN metiltransferases.
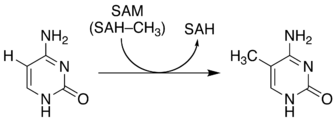
A función deste composto varía significativamente entre as especies:[7]
- En bacterias, a 5-metilcitosina pode encontrarse en diversos sitios, e a miúdo utilízase como marcador para protexer o ADN de ser cortado por encimas de restrición sensibles á metilación nativos.
- En plantas, a 5-metilcitosina aparece nas secuencias CpG, CpHpG e CpHpH (onde H = A, C ou T).
- En fungos e animais, a 5-metilcitosina aparece predominantemente nos dinucleótidos CpG. A maioría dos eucariotas metilan só unha pequena porcentaxe deses sitios, pero en vertebrados entre o 70 e o 80% das citosinas CpG están metiladas.
Aínda que a desaminación espontánea da citosina orixina uracilo, o cal é recoñecido e eliminado polos encimas de reparación do ADN (por estar nun lugar indebido), a desaminación da 5-metilcitosina forma timina. Esta conversión dunha base do ADN de citosina (C) a timina (T) pode causar unha mutación por transición. Ademais, a desaminación encimática activa da citosina ou da 5-metilcitosina pola familia APOBEC de citosina desaminases podería ter implicacións beneficiosas sobre varios procesos celulares e sobre a evolución dos organismos.[8] por outra parte, as implicacións da desaminación da 5-hidroximetilcitosina coñécense peor.
In vitro
[editar | editar a fonte]O grupo NH2 pode ser eliminado (desaminación) da 5-metilcitosina para formar timina co uso de reactivos como o ácido nitroso. A citosina desamínase a uracilo en similares condicións.
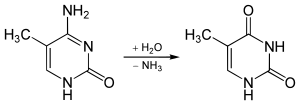
A 5-metilcitosina é resistente á desaminación por tratamento con bisulfito, o cal si pode desaminar residuos de citosina. Esta propiedade aprovéitase a miúdo para analizar os patróns de metilación de citosinas no ADN por medio de secuenciación por bisulfito.[9]
Notas
[editar | editar a fonte]- ↑ Matthews AP (2012). Physiological Chemistry. Williams & Wilkins Company. p. 167. ISBN 1130145379.
- ↑ Johnson TB, Coghill RD (1925). "The discovery of 5-methyl-cytosine in tuberculinic acid, the nucleic acid of the Tubercle bacillus". J Am Chem Soc 47 (11): 2838–2844. doi:10.1021/ja01688a030.
- ↑ Grosjean H (2009). Nucleic Acids Are Not Boring Long Polymers of Only Four Types of Nucleotides: A Guided Tour. Landes Bioscience.
- ↑ Vischer E, Zamenhof S, Chargaff E (1949). "Microbial nucleic acids: the desoxypentose nucleic acids of avian tubercle bacilli and yeast". J Biol Chem 77 (1): 429–438. PMID 18107446.
- ↑ Hotchkiss RD (1948). "The quantitative separation of purines, pyrimidines and nucleosides by paper chromatography". J Biol Chem 175 (1): 315–332. PMID 18107446.
- ↑ Squires JE, Patel HR, Nousch M, Sibbritt T, Humphreys DT, Parker BJ, Suter CM, Preiss T. (2012). "Widespread occurrence of 5-methylcytosine in human coding and non-coding RNA". Nucleic Acids Res 40 (11): 5023–5033. PMC 3367185. PMID 22344696. doi:10.1093/nar/gks144.
- ↑ Colot V, Rossignol JL (1999). "Eukaryotic DNA methylation as an evolutionary device". BioEssays 21 (5): 402–411. PMID 10376011. doi:10.1002/(SICI)1521-1878(199905)21:5<402::AID-BIES7>3.0.CO;2-B.
- ↑ Chahwan R., Wontakal S.N., and Roa S. (2010). "Crosstalk between genetic and epigenetic information through cytosine deamination". Trends in Genetics 26 (10): 443–448. PMID 20800313. doi:10.1016/j.tig.2010.07.005.
- ↑ Clark SJ, Harrison J, Paul CL, Frommer M (1994). "High sensitivity mapping of methylated cytosines". Nucleic Acids Res. 22 (15): 2990–2997. PMC 310266. PMID 8065911. doi:10.1093/nar/22.15.2990.
Véxase tamén
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Griffiths, Anthony J. F. (1999). An Introduction to genetic analysis. San Francisco: W.H. Freeman. Chapter 15: Gene Mutation. ISBN 0-7167-3520-2. (dispoñible en liña no National Center for Biotechnology Information de EUA)