ADN xirase
| ADN xirase | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
| ADN topoisomerase IIA dímero, humana | |||||||||
| Identificadores | |||||||||
| Número EC | 5.99.1.3 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| |||||||||
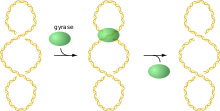
A ADN xirase (ou DNA xirase ou ADN topoisomerase II), ás veces denominada simplemente xirase, é un encima que alivia a tensión xerada no ADN cano o ADN bicatenario está sendo desenrolado por unha helicase.[1][2] Isto causa un superenrolamento negativo no ADN. A ADN xirase pertence a un tipo de encimas chamados topoisomerases que provocan cambios na topoloxía do ADN. A ADN xirase bacteriana é a diana de moitos antibióticos, como o ácido nalidíxico, a novobiocina, e a ciprofloxacina. Superenrola negativamene (ou relaxa superenrolamentos positivos) no ADN ao formar un bucle no molde para formar un cruzamento, despois corta unha das dobre hélices e pasa a outra a través dela antes de liberar a rotura, cambiando o índice de ligazón por dous en cada paso encimático. O índice de ligazón ou número de enlace (linking number) é un parámetro do superenrolamento que especifica o número de veces que dúas febras dun círculo de ADN bicatenario están interenroladas. Este proceso ocorre en bacterias, cuxo ADN circular é cortado pola ADN xirase e os dous extremos resultantes son despois retorcidos un sobre o outro e forman o superenrolamento. Unha xirase atopouse tamén no apicoplasto do parasito da malaria Plasmodium falciparum, un eucariota unicelular.[3][4]
A característica capacidade da xirase de introducir superenrolamentos negativos no ADN é o que permite que o ADN bacteriano teña superenrolamentos negativos libres. A habilidade da xirase de relaxar os superenrolamentos positivos entra en xogo durante a replicación do ADN e na transcrición procariótica. A natureza dextroxira da dobre hélice do ADN causa superenrolamentos positivos que se acumulan por diante dun encima que se transloca, que no caso da replicación do ADN, é unha ADN polimerase. A capacidade da xirase (e da topoisomerase IV) de relaxar superenrolamentos positivos permite que a tensión da superhélice por diante da polimerase se libere para que a replicación poida continuar.
A ADN xirase foi descuberta en 1976 por Gellert e colaboradores cando estaban estudando os factores necesarios para a integración do bacteriófago X en Escherichia coli.[5]
Modelo mecanoquímico da actividade da xirase
[editar | editar a fonte]Un estudo da molécula[6] caracterizou a actividade da xirase como unha función da tensión do ADN (forza aplicada) e do ATP, e propuxo un modelo mecanoquímico. Ao unirse ao ADN (o estado "xirase-ADN"), hai unha competición entre o enroscamento e a disociación do ADN, no que o incremento da tensión do ADN incrementa a probailidade de disociación. Despois do enroscamento e hidrólise do ATP, introdúcense dous enrolamentos negativos no molde, o que dá a oportunidade de que se produzan máis enrolamentos e superenrolamentos. O número de xiros de superhélice introducidos nun ADN circular inicialmente relaxado calculouse que é aproximadamente igual ao número de moléculas de ATP hidrolizadas pola xirase.[7] Por tanto, suxeriuse que se hidrolizan pola xirase dúas moléculas de ATP por ciclo de reacción, o que leva a introducir unha diferenza de ligazón de -2.[5]
Inhibición por antibióticos
[editar | editar a fonte]A xirase está presente nos procariotas e nalgúns eucariotas, pero estes encimas non son totalmente similares en estrutura ou secuencia, e teñen diferentes afinidades por distintas moléculas. Isto fai que a xirase sexa unha boa diana para antibióticos. Sábese que dúas clases de antibióticos poden inhibir a xirase, que son:
- As aminocoumarinas (como a novobiocina). As aminocoumarinas actúan por medio dunha inhibición competitiva da enerxía de transdución da ADN xirase ao unirse ao sitio activo con actividade ATPase situado na subunidade GyrB.
- As quinolonas (como o ácido nalidíxico e a ciprofloxacina). As quinolonas únense a estes encimas e impídenlles que fagan a descatenación do ADN en replicación. As bacterias resistentes ás quinolonas xeralmente teñen topoisomerases mutadas que resisten a unión das quinolonas.
A ADN xirase ten dúas subunidades, que á súa vez constan doutras dúas subunidades cada unha, é dicir, dúas subunidades A e dúas B. As subunidaes A e B únense xuntas ao ADN, hidrolizan o ATP, e introducen os superenrolamentos negativos. A subunidade A leva a cabo o corte no ADN, e a subunidade B introduce os superenrolamentos negativos, e despois a subunidade A volve a unir as febras cortadas. As fluoroquinolonas únense á subunidade A e interfiren coas súa función de cortar e volver a unir as febras.
A subunidade A é inactivada selectivamente por antibióticos como os ácidos nalidíxico e oxolínico. A subunidade B é inactivada selectivamente por antibióticos como a coumermicina A1 e a novobiocina. A inhibición de ambas as subunidades bloquea a actividade de superenrolamento.[8]
Notas
[editar | editar a fonte]- ↑ Wigley, D.B.; Davies, G.J.; Dodson, E.J.; Maxwell, A; Dodson, G (1991). "Crystal structure of an N-terminal fragment of the DNA gyrase B protein". Nature 351 (6328): 624–629. Bibcode:1991Natur.351..624W. PMID 1646964. doi:10.1038/351624a0.
- ↑ Morais Cabral, J. H.; Jackson, A. P.; Smith, C. V.; Shikotra, N; Maxwell, A; Liddington, R. C. (1997). "Crystal structure of the breakage-reunion domain of DNA gyrase". Nature 388 (6645): 903–6. Bibcode:1997Natur.388..903M. PMID 9278055. doi:10.1038/42294.
- ↑ "Molecular Cloning of Apicoplast-Targeted Plasmodium falciparum DNA Gyrase Genes: Unique Intrinsic ATPase Activity and ATP-Independent Dimerization of PfGyrB Subunit". EUKARYOTIC CELL: 398–412. 2007.
- ↑ "A unique 45 amino acid region in the Toprim domain of Plasmodium falciparum GyraseB is essential for its activity. (2009)". Eukaryotic Cell 11 (8): 1759–69.
- ↑ 5,0 5,1 Reece, Richard (1991). "DNA Gyrase: Structure and Function". Critical Reviews in Biochemistry and Molecular Biology 26 (3/4): 335–375. doi:10.3109/10409239109114072. pdf Arquivado 25 de setembro de 2019 en Wayback Machine.
- ↑ Gore J, Bryant Z, Stone MD, Nollmann M, Cozzarelli NR, Bustamante C, "Mechanochemical Analysis of DNA Gyrase Using Rotor Bead Tracking", Nature 2006 Jan 5 (Vol. 439): 100-104.
- ↑ Sugino, Akio (1980-10-07). "The Intrinsic ATPase of DNA Gyrase". Journal of Biological Chemistry 255 (13).
- ↑ Engle, E C; Manes, S H; Drlica, K. "Differential effects of antibiotics inhibiting gyrase". Journal of Bacteriology.