Marco de lectura
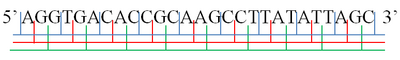
A·GGT·GAC·ACC·GCA·AGC·CTT·ATA·TTA·GC
AG·GTG·ACA·CCG·CAA·GCC·TTA·TAT·TAG·C
En bioloxía molecular, un marco de lectura é unha forma de dividir a secuencia de nucleótidos dunha molécula de ácido nucleico (ADN ou ARN) nun conxunto de tripletes consecutivos e non solapados. Cando estes tripletes igualan o número de aminoácidos ou sinais de parada durante a tradución de proteínas, denomínanse codóns.
Unha soa febra de molécula de ácido nucleico ten un extremo fosforilo, chamado extremo 5′ e un extremo hidroxilo ou extremo 3′. Estes definen a dirección 5′→3′. Hai tres marcos de lectura que se poden ler nesa rirección 5′→3′, cada un dos cales empeza nun nucleótido diferente do triplete. Nun ácido nucleico bicatenario, hai outros tres marcos de lectura posibles na outra febra, chamada febra complementaria na dirección 5′→3′ desa febra. Como as dúas febras dunha molécula de ácido nucleico bicatenario son antiparalelas, a dirección 5′→3′ da segunda febra correspóndese coa dirección 3′→5′ da outra febra.[1][2]
En xeral, como moito, un só marco de lectura nunha sección determinada dun ácido nucleico é bioloxicamente relevante (marco de lectura aberto). Algúns transcritos virais poden traducirse usando marcos múltiples solapados.[3] Coñécese un exemplo de marcos de lectura solapados no ADN mitocondrial de mamíferos: porcións codificantes de xenes para dúas subunidades da ATPase que se solapan.
Código xenético
[editar | editar a fonte]- Artigo principal: Código xenético.
O ADN codifica as secuencias das proteínas por medio de series de tres nucleótidos codóns. Calquera secuencia de ADN podería lerse de seis mneiras: tres marcos de lectura nunha dirección (empezando en diferentes nucleótidos) e tres na dirección oposta. Durante a transcrición, a ARN polimerase le o molde da febra de ADN na dirección 3′→5′, pero o ARNm fórmase en dirección 5′→3′.[4] O ARNm é monoctenario e, por tanto, só contén tres marcos de lectura posibles, dos cales só un é traducido. Os codóns do marco de lectura do ARNm son traducidos na dirección 5′→3′ a aminoácidos por un ribosoma para producir unha cadea polipeptídica.
Marco de lectura aberto
[editar | editar a fonte]- Artigo principal: Marco de lectura aberto.
Un marco de lectura aberto ou ORF polas súas siglas en inglés (de open reading frame) é un marco de lectura que ten o potencial de ser transcrito a ARN e traducido a proteína. Require unha secuencia continua de ADN desde o codón de inicio, ao longo da rexión que está a continuación que xeralmente ten unha lonxitude de núcleótidos que é múliplo de 3, ata o codón de parada ou terminación do marco de lectura.[5]
Cando unha suposta secuencia de aminoácidos resultaría da tradución dun ORF que permanece descoñecido nos xenomas de mitocondrias e cloroplastos, o correspondente marco de lectura aberto denomínase marco de lectura non identificado ou URF (do inglés unidentified reading frame). Por exemplo, o xene MT-ATP8 foi descrito primeiro como o URF A6L cando se completou a secuenciación do xenoma mitocondrial humano.[6]
Marcos de lectura múltiple
[editar | editar a fonte]
O uso de marcos de lectura múltiple ofrece a posibilidade de que existan xenes solapados; hai moitos deles nos xenomas virais, procariotas e mitocondriais.[7] Algúns virus, por exemplo o virus da hepatite B e o BYDV, usan varios xenes solapados en diferentes marcos de lectura.
En raros casos, un ribosoma pode saltar dun marco a outro durante a tradución dun ARNm (desprazamento de marco traducional). Isto causa que a primeira parte do ARNm se traduza dun marco de lectura e a parte final se traduza dun marco de lectura diferente. Isto é diferente dunha mutación de cambio de pauta de lectura, xa que nela a secuencia de nucleótidos (ADN ou ARN) non se altera e só se altera o marco en que esta será lida.
Notas
[editar | editar a fonte]- ↑ Rainey S, Repka J. "Quantitative sequence and open reading frame analysis based on codon bias" (PDF). Systemics, Cybernetics and Informatics 4 (1): 65–72.
- ↑ Badger JH, Olsen GJ (April 1999). "CRITICA: Coding Region Identification Tool Invoking Comparative Analysis". Mol Biol Evol 16 (4): 512–24. PMID 10331277. doi:10.1093/oxfordjournals.molbev.a026133.
- ↑ Lander, Eric. "MITx 7.00x Biology". Arquivado dende o orixinal o 07 de decembro de 2019. Consultado o 17 de abril de 2020.
- ↑ Lodish (2007). Molecular Cell Biology (6th ed.). W. H. Freeman. p. 121. ISBN 978-1429203142.
- ↑ Benjamin C. Pierce (2012). Genetics: a conceptual approach. W. H. Freeman. ISBN 9781429232500.
- ↑ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). "Sequence and organization of the human mitochondrial genome". Nature 290 (5806): 457–65. PMID 7219534. doi:10.1038/290457a0.
- ↑ Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res 14 (11): 2268–72. PMC 525685. PMID 15520290. doi:10.1101/gr.2433104.