Tiorredoxina redutase
| Identificadores | |
|---|---|
| Símbolo | TrxR |
| InterPro | IPR005982 |
| PROSITE | PS00573 |
| SCOPe | 1zof / SUPFAM |
As tiorredoxina redutases (TR, TrxR, ThxR, TXNRD) (EC 1.8.1.9) son encimas que reducen a tiorredoxina (Trx).[1] Identificáronse dúas clases de tiorredoxina redutases: unha clase en bacterias e algúns eucariotas e unha en animais. En bacterias as TrxR tamén catalizan a redución das proteínas similares á glutarredoxina chamadas NrdH.[2][3][4] Ambas as clases son flavoproteínas que funcionan como homodímeros. Cada monómero contén un grupo prostético FAD, un dominio de unión ao NADPH, e un sitio activo que contén un enlace disulfuro con actividade redox.[5]
Función celular
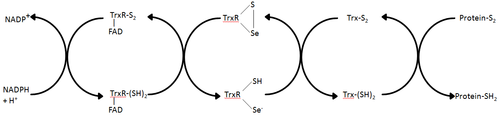
[editar | editar a fonte]As tiorredoxina redutases (TrxR) son encimas que catalizan a redución da tiorredoxina (Trx)[1] e, por tanto, son un compoñente central do sistema da tiorredoxina. Xunto coa tiorredoxina (Trx) e o NADPH a descrición máis xeral deste sistema é o dun sistema que reduce os enlaces disulfuro nas células. Tómanse electróns do NADPH por medio da TrxR e transfírense ao sitio activo da Trx, a cal reduce disulfuros en poteínas ou outros substratos.[6] O sistema Trx existe en todas as células vivas e ten unha historia evolutiva ligada ao ADN como material xenético, defensa contra danos oxidativos debido ao metabolismo do oxíxeno, e sinalización redox usando moléculas como o peróxido de hidróxeno e o óxido nítrico.[7][8]
Diversidade
[editar | editar a fonte]Evolucionaron independentemente dúas clases de tiorredoxina redutases:
- Un tipo de alto peso molecular (MW = ~55,000) que contén un residuo de selenocisteína no seu sitio activo identifiacado en eucariotss superiores incluíndo humanos. Esta TxR está relacionada coa glutatión redutase, tripanotión redutase, mercurio(II) redutase e lipoamida deshidroxenase.[5]
- Un tipo de baixo peso molecular (MW = ~ 35,000) identificado en arqueas, bacterias e algúns eucariotas.[5]
Estas dúas clases de TrxR teñen só un ~20% de identidade de secuencia na sección de secuencia primaria na que poden ser aliñadas fiablemente.[5] A reacción neta de ambas as clases de TrxR é idéntica pero o mecanismo de acción de cada unha é distinto.[9]
Os humanos expresan tres isocimas tiorredoxina redutases, que son: tiorredoxina redutase 1 (TrxR1, citosólica), tiorredoxina redutase 2 (TrxR2, mitocondrial), tiorredoxina redutase 3 (TrxR3, específica do testículo).[10] Cada encima está codificado por un xene separado, cada un nun cromosoma distinto:
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Estrutura
[editar | editar a fonte]E. coli
[editar | editar a fonte]En Escherichia coli a tiorredoxina redutase (ThxR) ten dous dominios de unión, un para o FAD e outro para o NADPH. A conexión entre estes dous dominios é unha folla β de dúas febras antiparalelas.[11] Cada dominio individualmente é moi similar a dominios análogos da glutatión redutase e lipoamida deshidroxenase pero a a orientación relativa destes dominios na ThxR está rotado 66 graos.[11] Isto é significativo no mecanismo de acción do encima que se describe máis abaixo. A ThxR homodimerízase coa interface entre os dous monómeros formados por tres hélices alfa e dous bucles.[11] Cada monómero pode unirse separadamente a unha molécula de tiorredoxina.
-
Estrutura da ThxR dímera de E. coli unida á tiorredoxina.
-
Estrutura da ThxR de E. coli cos grupos prostéticos FAD e NADPH etiquetados.
Mamíferos
[editar | editar a fonte]A estrutura da tiorredoxina redutase de mamíferos é similar á de E. coli. Contén un dominio de unión ao FAD e ao NADPH e unha interface entre dúas subunidades monómeras. Na tiorredoxina redutase de mamíferos hai unha inserción no dominio de unión ao FAD entre dúas hélices alfa que forman un pequeno par de febras beta.[12] O disulfuro activo do encima está localizado nunha destas hélices e así o enlace disulfuro activo está localizado no dominio FAD e non no dominio NADPH como en E. coli e outros procariotas.[12]
-
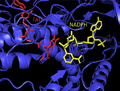
Estrutura dos grupos prostéticos FAD e NADPH da ThxR humana.
Mecanismo
[editar | editar a fonte]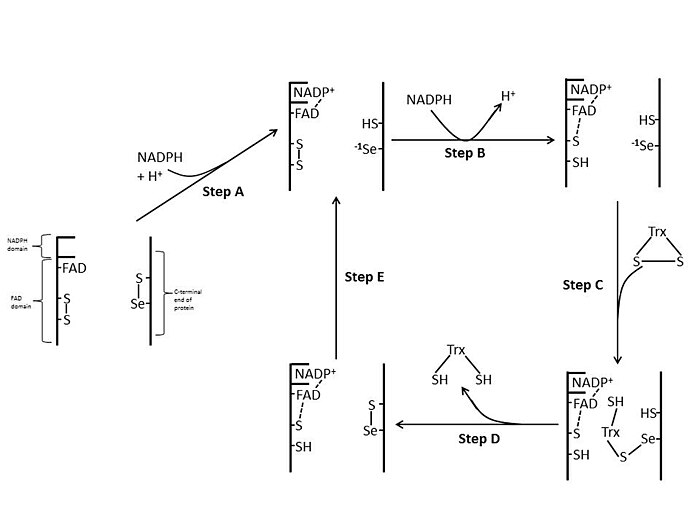
E. coli
[editar | editar a fonte]En E. coli a orientación espacial da ThxR dos dominios FAD e NADPH fai que os aneis con actividade redox do FAD e NADPH non están en estreita proximidade.[1] Cando o dominio FAD de E. coli está rotado 66 graos e o dominio NADPH permanece fixado, os dous grupos prostéticos móvense en contacto próximo permitindo que os electróns pasen do NADPH ao FAD e despois ao enlace disulfuro do sitio activo.[1][15] Os residuos do sitio activo conservados en E. coli son -Cys-Ala-Thr-Cys-.[1]
Mamíferos
[editar | editar a fonte]As TrxRs de mamíferos teñen unha homoloxía de secuencias moito maior coa glutatión redutase que E. coli.[1] Os residuos de Cys do sitio activo no dominio FAD e e do dominio NADPH unido están en estreita proximidade polo que non hai necesidade dunha rotación de 66 graos para a transferencia de electróns como a atopada en E. coli. Unha caracteística adicional do mecanismo dos mamíferos é a presenza dun residuo de selenocisteína no extremo C-terminal da proteína que cómpre para a actividade catalítica. Os residuos conservados no sitio activo de mamíferos son -Cys-Val-Asn-Val-Gly-Cys-.[1]
Métodos de detección
[editar | editar a fonte]A tiorredoxina redutase pode ser cuantificada por varios métodos como o ensaio DTNB que usa o reactivo de Ellman. A serie de sondas fluorescentes TRFS baseadas en disulfuros mostraron pode facer unha detección selectiva da TrxR.[16] [17][18][19] Mafireyi sintetizou a primeira sonda diselenuro que foi aplicada na detección da TrxR.[20] [21] Outros métodos de detección son técnicas inmunolóxicas e o ensaio de selenocistina-tiorredoxina (ensaio SC-TR).
Importancia clínica
[editar | editar a fonte]Tratamento do cancro
[editar | editar a fonte]Como a actividade deste encima é esencial para o crecemento e supervivencia celular, é unha boa diana para a terapia antitumoral. Ademais, o encima é regulado á alza en varios tipos de cancro, incluíndo o mesotelioma maligno.[22][23] Por exemplo, o motexafín gadolinio (MGd) é un novo axente quimioterapéutico que actúa selectivamente en células tumorais, orixinando a morte celular e a apoptose por inhibición da tiorredoxina redutase e a ribonucleótido redutase.
Cardiomiopatía
[editar | editar a fonte]A cardiomiopatía dilatada é un diagnóstico común en casos de insuficiencia cardíaca conxestiva. As tiorredoxina redutases son proteínas esenciais para regular o balance redox celular e mitigando o dano causado polas especies reactivas do oxíxeno xeradas pola fosforilación oxidativa nas mitocondrias. A inactivación da TrxR2 mitocondrial en ratos ten como resultado o adelgazamento das paredes ventriculares do corazón e a morte neonatal.[10] Ademais, atópanse dúas mutacións no xene TrxR2 en pacientes diagnosticados de cardiomiopatía dilatada, pero non nunha poboación de control. Hipotetízase que o impacto patolóxico destas mutacións é unha capacidade alterada para controlar os danos oxidativos en miocitos cardíacos.[24]
Antibióticos
[editar | editar a fonte]Recentemente algunhas investigacións mostraron que o baixo peso molecular da tiorredoxina redutase podería ser unha diana para novos antibióticos (como o auranofín ou o Ebselen.[25]) Isto é especialmente certo en Mycobacterium haemophilum e podería usarse contra bacterias resistentes a antibióticos.[26]
Notas
[editar | editar a fonte]- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 1,6 Mustacich D, Powis G (febreiro de 2000). "Thioredoxin reductase". The Biochemical Journal. 346 Pt 1 (1): 1–8. PMC 1220815. PMID 10657232. doi:10.1042/0264-6021:3460001.
- ↑ Jordan A, Aslund F, Pontis E, Reichard P, Holmgren A (xullo de 1997). "Characterization of Escherichia coli NrdH. A glutaredoxin-like protein with a thioredoxin-like activity profile". The Journal of Biological Chemistry 272 (29): 18044–50. PMID 9218434. doi:10.1074/jbc.272.29.18044.
- ↑ Phulera S, Mande SC (xuño de 2013). "The crystal structure of Mycobacterium tuberculosis NrdH at 0.87 Å suggests a possible mode of its activity". Biochemistry 52 (23): 4056–65. PMID 23675692. doi:10.1021/bi400191z.
- ↑ Phulera S, Akif M, Sardesai AA, Mande SC (2014-01-01). "Redox Proteins of Mycobacterium tuberculosis". Journal of the Indian Institute of Science (en inglés) 94 (1): 127–138. ISSN 0970-4140.
- ↑ 5,0 5,1 5,2 5,3 Hirt RP, Müller S, Embley TM, Coombs GH (xullo de 2002). "The diversity and evolution of thioredoxin reductase: new perspectives". Trends in Parasitology 18 (7): 302–8. PMID 12379950. doi:10.1016/S1471-4922(02)02293-6.
- ↑ 6,0 6,1 Holmgren A, Lu J (maio de 2010). "Thioredoxin and thioredoxin reductase: current research with special reference to human disease". Biochemical and Biophysical Research Communications 396 (1): 120–4. PMID 20494123. doi:10.1016/j.bbrc.2010.03.083.
- ↑ Meyer Y, Buchanan BB, Vignols F, Reichheld JP (2009). "Thioredoxins and glutaredoxins: unifying elements in redox biology". Annual Review of Genetics 43: 335–67. PMID 19691428. doi:10.1146/annurev-genet-102108-134201.
- ↑ Lillig CH, Holmgren A (xaneiro de 2007). "Thioredoxin and related molecules--from biology to health and disease". Antioxidants & Redox Signaling 9 (1): 25–47. PMID 17115886. doi:10.1089/ars.2007.9.25.
- ↑ Arscott LD, Gromer S, Schirmer RH, Becker K, Williams CH (abril de 1997). "The mechanism of thioredoxin reductase from human placenta is similar to the mechanisms of lipoamide dehydrogenase and glutathione reductase and is distinct from the mechanism of thioredoxin reductase from Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America 94 (8): 3621–6. Bibcode:1997PNAS...94.3621A. PMC 20490. PMID 9108027. doi:10.1073/pnas.94.8.3621.
- ↑ 10,0 10,1 Conrad M, Jakupoglu C, Moreno SG, Lippl S, Banjac A, Schneider M, Beck H, Hatzopoulos AK, Just U, Sinowatz F, Schmahl W, Chien KR, Wurst W, Bornkamm GW, Brielmeier M (novembro de 2004). "Essential role for mitochondrial thioredoxin reductase in hematopoiesis, heart development, and heart function". Molecular and Cellular Biology 24 (21): 9414–23. PMC 522221. PMID 15485910. doi:10.1128/MCB.24.21.9414-9423.2004.
- ↑ 11,0 11,1 11,2 Williams CH (outubro de 1995). "Mechanism and structure of thioredoxin reductase from Escherichia coli". FASEB Journal 9 (13): 1267–76. PMID 7557016. doi:10.1096/fasebj.9.13.7557016. hdl:2027.42/154540.
- ↑ 12,0 12,1 Sandalova T, Zhong L, Lindqvist Y, Holmgren A, Schneider G (agosto de 2001). "Three-dimensional structure of a mammalian thioredoxin reductase: implications for mechanism and evolution of a selenocysteine-dependent enzyme". Proceedings of the National Academy of Sciences of the United States of America 98 (17): 9533–8. Bibcode:2001PNAS...98.9533S. PMC 55487. PMID 11481439. doi:10.1073/pnas.171178698.
- ↑ Zhong L, Arnér ES, Holmgren A (maio de 2000). "Structure and mechanism of mammalian thioredoxin reductase: the active site is a redox-active selenolthiol/selenenylsulfide formed from the conserved cysteine-selenocysteine sequence". Proceedings of the National Academy of Sciences of the United States of America 97 (11): 5854–9. Bibcode:2000PNAS...97.5854Z. PMC 18523. PMID 10801974. doi:10.1073/pnas.100114897.
- ↑ Becker K, Herold-Mende C, Park JJ, Lowe G, Schirmer RH (agosto de 2001). "Human thioredoxin reductase is efficiently inhibited by (2,2':6',2' '-terpyridine)platinum(II) complexes. Possible implications for a novel antitumor strategy". Journal of Medicinal Chemistry 44 (17): 2784–92. PMID 11495589. doi:10.1021/jm001014i.
- ↑ Lennon BW, Williams CH (agosto de 1997). "Reductive half-reaction of thioredoxin reductase from Escherichia coli". Biochemistry 36 (31): 9464–77. PMID 9235991. doi:10.1021/bi970307j.
- ↑ Li X, Zhang B, Yan C, Li J, Wang S, Wei X, et al. (xuño de 2019). "A fast and specific fluorescent probe for thioredoxin reductase that works via disulphide bond cleavage". Nature Communications 10 (1): 2745. Bibcode:2019NatCo..10.2745L. PMC 6588570. PMID 31227705. doi:10.1038/s41467-019-10807-8.
- ↑ Ma H, Zhang J, Zhang Z, Liu Y, Fang J (outubro de 2016). "A fast response and red emission probe for mammalian thioredoxin reductase". Chemical Communications 52 (81): 12060–12063. PMID 27709154. doi:10.1039/C6CC04984B.
- ↑ Zhao J, Qu Y, Gao H, Zhong M, Li X, Zhang F, et al. (novembro de 2020). "Loss of thioredoxin reductase function in a mouse stroke model disclosed by a two-photon fluorescent probe". Chemical Communications 56 (90): 14075–14078. PMID 33107534. doi:10.1039/D0CC05900E.
- ↑ Liu Y, Ma H, Zhang L, Cui Y, Liu X, Fang J (febreiro de 2016). "A small molecule probe reveals declined mitochondrial thioredoxin reductase activity in a Parkinson's disease model". Chemical Communications 52 (11): 2296–9. PMID 26725656. doi:10.1039/c5cc09998f.
- ↑ Mafireyi TJ, Laws M, Bassett JW, Cassidy PB, Escobedo JO, Strongin RM (agosto de 2020). "A Diselenide Turn-On Fluorescent Probe for the Detection of Thioredoxin Reductase". Angewandte Chemie 59 (35): 15147–15151. PMC 9438933. PMID 32449244. doi:10.1002/ange.202004094.
- ↑ Mafireyi TJ, Escobedo JO, Strongin RM (2021-03-29). "Fluorogenic probes for thioredoxin reductase activity". Results in Chemistry (en inglés) 3: 100127. ISSN 2211-7156. doi:10.1016/j.rechem.2021.100127.
- ↑ Nilsonne G, Sun X, Nyström C, Rundlöf AK, Potamitou Fernandes A, Björnstedt M, Dobra K (setembro de 2006). "Selenite induces apoptosis in sarcomatoid malignant mesothelioma cells through oxidative stress". Free Radical Biology & Medicine 41 (6): 874–85. PMID 16934670. doi:10.1016/j.freeradbiomed.2006.04.031. hdl:10616/47514.
- ↑ Kahlos K, Soini Y, Säily M, Koistinen P, Kakko S, Pääkkö P, Holmgren A, Kinnula VL (maio de 2001). "Up-regulation of thioredoxin and thioredoxin reductase in human malignant pleural mesothelioma". International Journal of Cancer 95 (3): 198–204. PMID 11307155. doi:10.1002/1097-0215(20010520)95:3<198::AID-IJC1034>3.0.CO;2-F.
- ↑ Sibbing D, Pfeufer A, Perisic T, Mannes AM, Fritz-Wolf K, Unwin S, Sinner MF, Gieger C, Gloeckner CJ, Wichmann HE, Kremmer E, Schäfer Z, Walch A, Hinterseer M, Näbauer M, Kääb S, Kastrati A, Schömig A, Meitinger T, Bornkamm GW, Conrad M, von Beckerath N (maio de 2011). "Mutations in the mitochondrial thioredoxin reductase gene TXNRD2 cause dilated cardiomyopathy". European Heart Journal 32 (9): 1121–33. PMID 21247928. doi:10.1093/eurheartj/ehq507.
- ↑ Marshall AC, Kidd SE, Lamont-Friedrich SJ, Arentz G, Hoffmann P, Coad BR, Bruning JB (marzo de 2019). "Aspergillus fumigatus Thioredoxin Reductase". Antimicrobial Agents and Chemotherapy 63 (3). PMC 6395915. PMID 30642940. doi:10.1128/AAC.02281-18.
- ↑ Harbut MB, Vilchèze C, Luo X, Hensler ME, Guo H, Yang B, et al. (abril de 2015). "Auranofin exerts broad-spectrum bactericidal activities by targeting thiol-redox homeostasis". Proceedings of the National Academy of Sciences of the United States of America 112 (14): 4453–8. Bibcode:2015PNAS..112.4453H. PMC 4394260. PMID 25831516. doi:10.1073/pnas.1504022112.
Véxase tamén
[editar | editar a fonte]Ligazóns externas
[editar | editar a fonte]- Thioredoxin Reductase (NADPH) Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.