ERCC4
ERCC4(excision repair cross-complementation group 4)もしくはXPF(xeroderma pigmentosum complementation group F)は、ヒトではERCC4遺伝子にコードされるタンパク質である。ERCC4はERCC1とともにERCC1-XPF複合体を形成し、DNA修復やDNA組換えに関与する[5][6]。
ERCC1-XPFはDNAの特定の構造を切断するヌクレアーゼである。ERCC1とERCC4はDNA修復時のパートナーであるため、これらの因子は多くの面に関して共に記載がなされている。ERCC1-XPF複合体は、DNAのヌクレオチド除去修復経路において必須となるヌクレアーゼである。ERCC1-XPFヌクレアーゼはDNA二本鎖切断の修復や、2本のDNA鎖を連結する有害な架橋損傷の修復経路においても機能する。
ERCC4に不活化変異を有する細胞は、紫外線(UV)照射やDNA鎖間の架橋を行う化学物質など、特定のDNA損傷因子に対する感受性が正常細胞よりも高くなる。Ercc4に不活化変異を有する遺伝子改変マウスはDNA修復に欠陥がみられ、また代謝ストレス誘発性の生理的変化も伴うことで早老が引き起こされる[7]。Ercc4を完全に(ホモ接合型で)欠失したマウスは生存することができず、またヒトでもERCC4のホモ接合型欠失は見つかっていない。ヒト集団では、稀にERCC4の機能不全をもたらす遺伝的変異を有する個体がみられる。正常な遺伝子が存在しない場合、こうした変異は色素性乾皮症、コケイン症候群やファンコニ貧血などの疾患の原因となる場合がある。
遺伝子
[編集]ERCC4遺伝子の導入によって、CHO細胞由来の特定の紫外線感受性変異株のDNA修復の欠陥を修正することができた[8]。CHO細胞では複数の独立した相補群が単離されており[9]、この遺伝子はcomplementation group 4の細胞で紫外線耐性を回復した。この種間での遺伝的相補性を反映して、遺伝子は"Excision repair cross-complementing 4"(ERCC4)と命名された[10]。
ヒトのERCC4遺伝子は、916アミノ酸からなる約104 kDaのタンパク質をコードする[11]。
ERCC4と同等の機能を有する類似した遺伝子(オルソログ)は他の真核生物のゲノムにも存在する。最もよく研究されている遺伝子としては、出芽酵母Saccharomyces cerevisiaeのRAD1遺伝子、分裂酵母Schizosaccharomyces pombeのrad16+遺伝子がある[11]。
タンパク質
[編集]
1分子のERCC1と1分子のERCC4(XPF)が結合することで、活性型ヌクレアーゼであるERCC1-XPFヘテロ二量体が形成される。ERCC1-XPFヘテロ二量体においては、ERCC1がDNA-タンパク質間相互作用、タンパク質-タンパク質間相互作用を媒介している。XPFはエンドヌクレアーゼ活性部位を持ち、またDNA結合やタンパク質-タンパク質間相互作用に関与している[8]。
ERCC4/XPFには2つの保存されたドメインが存在し、その間は低保存性領域によって隔てられている。N末端領域はDNAヘリカーゼスーパーファミリーIIの保存されたドメインのいくつかとの相同性がみられるが、XPFはDNAヘリカーゼではない[12]。C末端領域にはヌクレアーゼ活性の活性部位残基が位置している[13]。ERCC1の大部分はXPFのC末端領域と配列レベルで関連しているが[14]、ヌクレアーゼドメインの残基は存在していない。ERCC1とXPFにはどちらもC末端に、DNAを結合するヘリックス-ヘアピン-ヘリックス(HhH)ドメインが存在する。
構造特異的ヌクレアーゼ
[編集]
ERCC1-XPF複合体は構造特異的エンドヌクレアーゼである。ERCC1-XPFは一本鎖のみまたは二本鎖のみからなるDNAに対する切断活性は持たないが、二本鎖DNAと一本鎖DNAの接合部のDNAホスホジエステル骨格を特異的に切断する。切断が導入されるのは、こうした接合部の5'側、約2ヌクレオチド離れた位置の二本鎖DNAである[15]。この構造特異性は、ERCC1-XPFの酵母オルソログであるRAD10-RAD1で初めて示された[16]。
ERCC1とXPFのC末端領域に位置する疎水的なHhHモチーフは、互いに相互作用して両者の二量体化を促進する[17][18]。二量体化していない場合、触媒活性を示すことはない。触媒ドメインはXPF内に存在し、ERCC1自体が触媒活性を持つわけではないが、ERCC1は複合体の活性に必要不可欠である。
ERCC1-XPFのDNAへの結合に関しては、関連するタンパク質断片の原子分解能構造に基づいて、いくつかのモデルが提唱されている[17]。DNAへの結合はERCC1とXPFのHhHドメインによって媒介され、これらのドメインによってヘテロ二量体は接合部に配置されていると考えられている。
ヌクレオチド除去修復
[編集]ヌクレオチド除去修復(NER)時には、いくつかのタンパク質複合体が協調的に損傷DNAを認識し、損傷部位の両側の短い範囲のDNAらせんを局所的に開く。ERCC-XPFヌクレアーゼは損傷DNA鎖の損傷部位の5'側に切り込みを入れる[15]。ERCC1はNERの過程ではXPAと相互作用し、DNAやタンパク質との協調的な相互作用を行う。
二本鎖切断修復
[編集]ERCC1-XPFに変異を有する哺乳類細胞は正常細胞と比較して、DNA二本鎖切断を引き起こす因子(電離放射線など)に対する感受性がある程度高くなる[19][20]。相同組換え修復と非相同末端結合の双方がERCC-XPF1の機能に依存しており[21][22]、どちらの二本鎖切断修復経路においてもERCC1-XPFの活性は再結合前のDNA末端からの非相同な3'一本鎖テールの除去と関係している。相同組換えにおいては、この活性は一本鎖アニーリング(single-strand annealing、SSA)経路で必要とされる。3'末端の一本鎖テールのトリミングは非相同末端結合経路においても必要であり、この経路においてはKuタンパク質の活性に依存している[23][24]。相同組換えを利用したDNAの組み込みは遺伝子操作の重要な技術であるが、ホスト細胞のERCC-XPFの機能に依存している[25]。
鎖間架橋の修復
[編集]ERCC1もしくはXPFに変異を有する哺乳類細胞は、DNA鎖間の架橋を引き起こす因子に対する感受性が特に高い[26]。鎖間架橋はDNA複製の進行を遮断し、遮断されたDNA複製フォーク部分の構造がERCC-XPFによる切断の基質となる[27][28]。DNAの一方の鎖の架橋の両側に切り込みを入れて架橋を外すことで、修復が開始されている可能性がある。他に、鎖間架橋付近のDNAに二本鎖切断が導入され、その後の相同組換え修復にERCC1-XPFが関与している可能性もある。ERCC1-XPFは鎖間架橋の修復に関与する唯一のヌクレアーゼではないものの、細胞周期のいくつかの段階で鎖間架橋の修復に必要とされる[29][30]。
臨床的意義
[編集]色素性乾皮症
[編集]希少遺伝疾患である色素性乾皮症(XP)の患者の一部は、ERCC4に変異を抱えている。これらの患者はグループF(XP-F)に分類される。XPの診断基準となる特徴は、乾燥した鱗状の皮膚、露光部の皮膚への異常な色素沈着、重度の光感受性であり、紫外線による皮膚がんの発症リスクが1000倍以上高くなる[5]。
コケイン症候群
[編集]XP-Fの患者の大部分ではXPの症状は中等度であるが、その中のわずかな割合でさらにコケイン症候群(CS)の症状がみられることがある[31]。コケイン症候群の患者は光感受性を示し、さらに発生の欠陥や神経症状を示す[5][7]。
ERCC4遺伝子の変異は、非常に稀にXFE早老症候群(XPF-ERCC1 progeroid syndrome)を引き起こす場合がある[32]。患者はXPとCSの特徴に加え、神経、肝胆道、筋骨格、造血系に症状がみられる。
ファンコニ貧血
[編集]ファンコニ貧血(FA)の症状を示す患者の中には、ERCC4遺伝子に原因変異が存在する場合がある。ファンコニ貧血は、主に造血系の症状を伴う複合的な疾患である。FAの特徴は、DNA鎖間の架橋を引き起こす因子に対する過敏性である。ERCC4遺伝子に変異を有するFAの患者はグループQ(FANCQ)に分類される[31][33]。
正常な結腸におけるERCC4
[編集]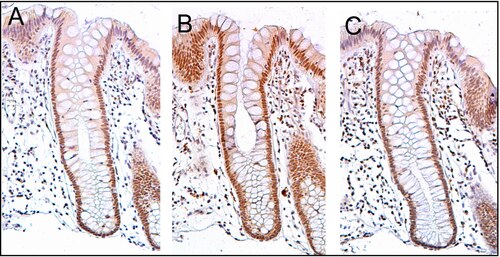
通常、ERCC4(XPF)は結腸の内壁の細胞の細胞核で高レベルに発現している。結腸の内壁は陥入を伴う単層円柱上皮で覆われている。陥入部は腸腺もしくは腸陰窩と呼ばれる。結腸陰窩は厚壁の試験管のような微視的形状をしており、管の長さ方向に中心に穴が開いている。陰窩の深さは細胞75個から110個分程度である。正常な非腫瘍性の結腸上皮ではDNA修復が非常に活発なようであり、ERCC4、PMS2、ERCC1の高発現を伴う。
細胞は陰窩の底部で生み出され、陰窩の軸に沿って上方に移動し、数日後には結腸管腔へ脱落する[35]。陰窩の底部には5個から6個の幹細胞が存在する。底部の幹細胞がERCC4を発現している場合、一般的にその陰窩の数千の細胞全てでERCC4が発現している。このことはこの節の図のパネルCにおいて、陰窩内のほぼ全ての腸細胞がERCC4に対する免疫染色で褐色に染色されていることからも示される。正常な結腸上皮の各陰窩の数千の腸細胞では、PMS2やERCC1でも同様の発現がみられる。この図ではヘマトキシリンによるDNAの対比染色も行われており、核が青灰色で示されている。粘膜固有層の細胞(陰窩の下部や周囲の細胞)の核の大部分はヘマトキシリンの青灰色を呈しており、PMS2、ERCC1、ERCC4をほとんど発現していない。また、陰窩の頂上部に位置する細胞ではPMS2やERCC4の発現は低下しており、同様に青灰色を呈している[34]。
結腸がんと隣接領域におけるERCC4の欠損
[編集]
大腸がんの約55%、そしてがんの周囲10 cm以内(発がん可能性の高い「発がん素地」(field defect)に位置する)結腸陰窩の約40%でERCC4(XPF)の発現は欠損している[34]。発がん素地においてERCC4が減少している場合には、DNA修復酵素ERCC1やPMSの発現も同様に低下していることが多い。結腸上皮におけるERCC1の欠乏は、エピジェネティックな抑制によるものであるようである[34]。ERCC4の欠乏はDNA損傷修復の減少をもたらすと考えられる。多くの種類のがんの根底には、DNA損傷に対して適切に応答して修復を行う能力の欠如がある[36]。がんやその周囲の発がん素地におけるERCC4のエピジェネティックな抑制は(ERCC1やPMS2のエピジェネティックな抑制とともに)、こうした変化が多くの結腸がんへの進行に中心的な役割を果たしている可能性を示している。
ERCC4の発現のエピジェネティックな低下はヒトの結腸がんで高頻度でみられるが、ERCC4に変異が生じていることは稀である[37]。ERCC4の変異は皮膚がんのリスクを高める[37]。また、ERCC4の遺伝的多型は乳がんにおいても重要であるようである[38]。こうした稀な変異もまた、ERCC4の欠乏ががんへの進行に関与している可能性が高いことを強調している。
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000175595 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000022545 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ a b c Friedberg, EC; Walker, GC; Siede, W; Wood, RD; Schultz, RA; Ellenberger, T (2006). DNA Repair and Mutagenesis. ASM Press. ISBN 978-1555813192
- ^ “Entrez Gene: ERCC4 excision repair cross-complementing rodent repair deficiency, complementation group 4”. 2023年1月28日閲覧。
- ^ a b “Physiological consequences of defects in ERCC1-XPF DNA repair endonuclease”. DNA Repair 10 (7): 781–91. (Jul 2011). doi:10.1016/j.dnarep.2011.04.026. PMC 3139823. PMID 21612988.
- ^ a b “Molecular cloning of a human DNA repair gene”. Nature 310 (5976): 425–9. (1984). Bibcode: 1984Natur.310..425W. doi:10.1038/310425a0. PMID 6462228.
- ^ “Summary of complementation groups of UV-sensitive CHO cell mutants isolated by large-scale screening”. Mutagenesis 4 (5): 349–54. (Sep 1989). doi:10.1093/mutage/4.5.349. PMID 2687628.
- ^ “ERCC4 (XPF) encodes a human nucleotide excision repair protein with eukaryotic recombination homologs”. Molecular and Cellular Biology 16 (11): 6553–62. (Nov 1996). doi:10.1128/mcb.16.11.6553. PMC 231657. PMID 8887684.
- ^ a b Manandhar, Mandira; Boulware, Karen S.; Wood, Richard D. (2015-09-15). “The ERCC1 and ERCC4 (XPF) genes and gene products”. Gene 569 (2): 153–161. doi:10.1016/j.gene.2015.06.026. ISSN 1879-0038. PMC 4536074. PMID 26074087.
- ^ “A relationship between a DNA-repair/recombination nuclease family and archaeal helicases”. Trends in Biochemical Sciences 24 (3): 95–7. (Mar 1999). doi:10.1016/s0968-0004(99)01355-9. PMID 10203755.
- ^ “The active site of the DNA repair endonuclease XPF-ERCC1 forms a highly conserved nuclease motif”. The EMBO Journal 21 (8): 2045–53. (Apr 2002). doi:10.1093/emboj/21.8.2045. PMC 125967. PMID 11953324.
- ^ “Activity of individual ERCC1 and XPF subunits in DNA nucleotide excision repair”. Nucleic Acids Research 29 (4): 872–9. (Feb 2001). doi:10.1093/nar/29.4.872. PMC 29621. PMID 11160918.
- ^ a b “Xeroderma pigmentosum group F caused by a defect in a structure-specific DNA repair endonuclease”. Cell 86 (5): 811–22. (Sep 1996). doi:10.1016/s0092-8674(00)80155-5. hdl:1765/3110. PMID 8797827.
- ^ “Specific cleavage of model recombination and repair intermediates by the yeast Rad1-Rad10 DNA endonuclease”. Science 265 (5181): 2082–5. (Sep 1994). Bibcode: 1994Sci...265.2082B. doi:10.1126/science.8091230. PMID 8091230.
- ^ a b “The structure of the human ERCC1/XPF interaction domains reveals a complementary role for the two proteins in nucleotide excision repair.”. Structure 13 (12): 1849–1858. (December 2005). doi:10.1016/j.str.2005.08.014. hdl:1874/14818. ISSN 1633-8413. PMID 16338413.
- ^ “The Cerebro-oculo-facio-skeletal Syndrome Point Mutation F231L in the ERCC1 DNA Repair Protein Causes Dissociation of the ERCC1-XPF Complex.”. J Biol Chem 290 (33): 20541–20555. (August 2015). doi:10.1074/jbc.M114.635169. ISSN 1083-351X. PMC 4536458. PMID 26085086.
- ^ “Radiation-induced lethality and mutation in a repair-deficient CHO cell line”. International Journal of Radiation Biology and Related Studies in Physics, Chemistry, and Medicine 43 (2): 207–13. (Feb 1983). doi:10.1080/09553008314550241. PMID 6600735.
- ^ “ERCC1-XPF endonuclease facilitates DNA double-strand break repair”. Molecular and Cellular Biology 28 (16): 5082–92. (Aug 2008). doi:10.1128/MCB.00293-08. PMC 2519706. PMID 18541667.
- ^ “Recombination-dependent deletion formation in mammalian cells deficient in the nucleotide excision repair gene ERCC1”. Proceedings of the National Academy of Sciences of the United States of America 94 (24): 13122–7. (Nov 1997). Bibcode: 1997PNAS...9413122S. doi:10.1073/pnas.94.24.13122. PMC 24273. PMID 9371810.
- ^ “The ERCC1/XPF endonuclease is required for efficient single-strand annealing and gene conversion in mammalian cells”. Nucleic Acids Research 36 (1): 1–9. (Jan 2008). doi:10.1093/nar/gkm888. PMC 2248766. PMID 17962301.
- ^ “Alternative-NHEJ is a mechanistically distinct pathway of mammalian chromosome break repair”. PLOS Genetics 4 (6): e1000110. (Jun 2008). doi:10.1371/journal.pgen.1000110. PMC 2430616. PMID 18584027.
- ^ “ERCC1-XPF endonuclease facilitates DNA double-strand break repair”. Molecular and Cellular Biology 28 (16): 5082–92. (Aug 2008). doi:10.1128/MCB.00293-08. PMC 2519706. PMID 18541667.
- ^ “The structure-specific endonuclease Ercc1-Xpf is required for targeted gene replacement in embryonic stem cells”. The EMBO Journal 20 (22): 6540–9. (Nov 2001). doi:10.1093/emboj/20.22.6540. PMC 125716. PMID 11707424.
- ^ “Mammalian nucleotide excision repair proteins and interstrand crosslink repair”. Environmental and Molecular Mutagenesis 51 (6): 520–6. (Jul 2010). doi:10.1002/em.20569. PMC 3017513. PMID 20658645.
- ^ “Recombination-dependent deletion formation in mammalian cells deficient in the nucleotide excision repair gene ERCC1”. Proceedings of the National Academy of Sciences of the United States of America 94 (24): 13122–7. (Nov 1997). Bibcode: 1997PNAS...9413122S. doi:10.1073/pnas.94.24.13122. PMC 24273. PMID 9371810.
- ^ “XPF-ERCC1 acts in Unhooking DNA interstrand crosslinks in cooperation with FANCD2 and FANCP/SLX4”. Molecular Cell 54 (3): 460–71. (May 2014). doi:10.1016/j.molcel.2014.03.015. PMC 5067070. PMID 24726325.
- ^ “Multiple roles of ERCC1-XPF in mammalian interstrand crosslink repair”. Environmental and Molecular Mutagenesis 51 (6): 567–81. (Jul 2010). doi:10.1002/em.20583. PMID 20658648.
- ^ “Advances in understanding the complex mechanisms of DNA interstrand cross-link repair”. Cold Spring Harbor Perspectives in Biology 5 (10): a012732. (Oct 2013). doi:10.1101/cshperspect.a012732. PMC 4123742. PMID 24086043.
- ^ a b “Malfunction of nuclease ERCC1-XPF results in diverse clinical manifestations and causes Cockayne syndrome, xeroderma pigmentosum, and Fanconi anemia”. American Journal of Human Genetics 92 (5): 807–19. (May 2013). doi:10.1016/j.ajhg.2013.04.007. PMC 3644632. PMID 23623389.
- ^ “A new progeroid syndrome reveals that genotoxic stress suppresses the somatotroph axis”. Nature 444 (7122): 1038–43. (Dec 2006). Bibcode: 2006Natur.444.1038N. doi:10.1038/nature05456. PMID 17183314.
- ^ “Mutations in ERCC4, encoding the DNA-repair endonuclease XPF, cause Fanconi anemia”. American Journal of Human Genetics 92 (5): 800–6. (May 2013). doi:10.1016/j.ajhg.2013.04.002. PMC 3644630. PMID 23623386.
- ^ a b c d e “Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer”. Genome Integr 3 (1): 3. (2012). doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ “Quantification of crypt and stem cell evolution in the normal and neoplastic human colon”. Cell Rep 8 (4): 940–7. (2014). doi:10.1016/j.celrep.2014.07.019. PMC 4471679. PMID 25127143.
- ^ “The DNA damage response: ten years after”. Mol. Cell 28 (5): 739–45. (2007). doi:10.1016/j.molcel.2007.11.015. PMID 18082599.
- ^ a b “Physiological consequences of defects in ERCC1-XPF DNA repair endonuclease”. DNA Repair (Amst.) 10 (7): 781–91. (2011). doi:10.1016/j.dnarep.2011.04.026. PMC 3139823. PMID 21612988.
- ^ “Combined genetic and nutritional risk models of triple negative breast cancer”. Nutr Cancer 66 (6): 955–63. (2014). doi:10.1080/01635581.2014.932397. PMID 25023197.





