リーディングフレーム
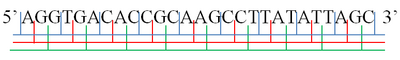
A·GGT·GAC·ACC·GCA·AGC·CTT·ATA·TTA·GC
AG·GTG·ACA·CCG·CAA·GCC·TTA·TAT·TAG·C
分子生物学では、リーディングフレーム(英: reading frame)とは、核酸(DNAまたはRNA)分子内のヌクレオチド配列を、重なり合わない連続したトリプレット(三連符)の集まりに分割する方法である。これらのトリプレットが、アミノ酸、あるいは翻訳時の終止信号に相当する場合、それらをコドンと呼ぶ。
核酸分子の一本の鎖には、5′末端と呼ばれるホスホリル端と、3′末端またはヒドロキシル端がある。これらは5′→3′方向を定義する。この5′→3′方向に読み取れるリーディングフレームは3つあり、それぞれはトリプレットの異なるヌクレオチドから始まる。二本鎖の核酸では、もう一方の相補的な鎖からこの鎖に沿って5′→3′方向に、さらに3つのリーディングフレームを読み取ることができる。二本鎖核酸分子の2本の鎖は逆平行であるため、第2鎖の5′→3′の方向は、第1鎖に沿った3′→5′の方向に対応する[1][2]。
一般的には、核酸の一部分の中で、生物学的に意味を持つのはせいぜい1つのリーディングフレーム(オープンリーディングフレームと呼ぶ)である。一部のウイルス転写産物は、複数のオーバーラップ(重畳)したリーディングフレームを使って翻訳できる。哺乳類のミトコンドリアDNAで、リーディングフレームがオーバーラップした例が知られている。それはATPaseの2つのサブユニットの遺伝子のコード部分である。
遺伝情報
[編集]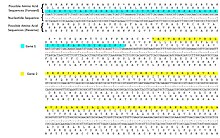
DNAは、一連の3つのヌクレオチドコドンによってタンパク質配列をコード化する。したがって、任意のDNA配列は6つの異なる方法で読み取ることができ、1つの方向に3つのリーディングフレーム(異なるヌクレオチドから始まる)と、逆方向に3つのリーディングフレームである。転写の際、RNAポリメラーゼは鋳型DNA鎖を3′→5′方向に読み取るが、mRNAは5′→3′方向に形成される[3]。mRNAは一本鎖であるため3つの可能なリーディングフレームしかなく、そのうちの1つだけが翻訳される。mRNAのリーディングフレームのコドンは、リボソームによって5′→3′方向にアミノ酸へ翻訳され、ポリペプチド鎖が生成される。
オープンリーディングフレーム
[編集]オープンリーディングフレーム(open reading frame、ORF)とは、RNAに転写されて、タンパク質に翻訳される可能性のあるリーディングフレームのことである。これには開始コドンから、通常3ヌクレオチドの倍数の長さを持つ後続領域を経て、同一リーディングフレーム内の終止コドンまでの連続したDNA配列を必要とする[4]。
ミトコンドリアや葉緑体のゲノムでは、あるORFが翻訳された結果、推定されるアミノ酸配列が不明のままの場合、対応するオープンリーディングフレームは「未確認のリーディングフレーム(URF)」と呼ばれていた。たとえば、MT-ATP8遺伝子は、ヒトのミトコンドリアゲノムの配列が完全に決定されたときに、初めてURF A6Lとして記載された[5]。
複数リーディングフレーム
[編集]
複数リーディングフレーム(multiple reading frames)を使用すると、オーバラップ遺伝子(overlapping gene)が生じる可能性がある。このようなものはウイルス、原核生物、およびミトコンドリアのゲノムに多数存在する可能性がある[6]。B型肝炎ウイルスやBYDVなどの一部のウイルスは、異なるリーディングフレームの中でいくつかのオーバーラップ遺伝子を使用している。オーバーラップ遺伝子を遺伝子重複(gene duplication)と混同しないよう注意すること。
まれに、mRNAの翻訳中にリボソームがあるフレームから別のフレームに移動することがあり、これを翻訳フレームシフト(translational frameshift)と言う。これにより、mRNAの最初の部分は1つのリーディングフレームで翻訳され、後の部分は異なるリーディングフレームで翻訳される。これは、フレームシフト変異とは異なるもので、ヌクレオチド配列(DNAまたはRNA)は変更されず、読み取られるフレームのみが変更される。
参照項目
[編集]脚注
[編集]- ^ Rainey S, Repka J. “Quantitative sequence and open reading frame analysis based on codon bias”. Systemics, Cybernetics and Informatics 4 (1): 65–72.
- ^ Badger JH, Olsen GJ (April 1999). “CRITICA: Coding Region Identification Tool Invoking Comparative Analysis”. Mol Biol Evol 16 (4): 512–24. doi:10.1093/oxfordjournals.molbev.a026133. PMID 10331277.
- ^ Lodish (2007). Molecular Cell Biology (6th ed.). W. H. Freeman. p. 121. ISBN 978-1429203142
- ^ Benjamin C. Pierce (2012). Genetics: a conceptual approach. W. H. Freeman. ISBN 9781429232500
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). “Sequence and organization of the human mitochondrial genome”. Nature 290 (5806): 457–65. Bibcode: 1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534.
- ^ Johnson Z, Chisholm S (2004). “Properties of overlapping genes are conserved across microbial genomes”. Genome Res 14 (11): 2268–72. doi:10.1101/gr.2433104. PMC 525685. PMID 15520290.