Termolisin
| Termolisin | |||||||||
|---|---|---|---|---|---|---|---|---|---|
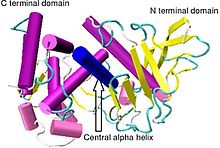 Struktur hablur termolisin Bacillus thermoproteolyticus.[1] | |||||||||
| Pengenal pasti | |||||||||
| Nombor EC | 3.4.24.27 | ||||||||
| Nombor CAS | 9073-78-3 | ||||||||
| Pangkalan data | |||||||||
| IntEnz | Lihat IntEnz | ||||||||
| BRENDA | Entri BRENDA | ||||||||
| ExPASy | Lihat NiceZyme | ||||||||
| KEGG | Entri KEGG | ||||||||
| MetaCyc | Laluan metabolik | ||||||||
| PRIAM | Profil | ||||||||
| Struktur PDB | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
Termolisin (EC 3.4.24.27, proteinase neutral Bacillus thermoproteolyticus, termoase, termoase Y10, TLN) ialah enzim metaloproteinase neutral termostabil yang dihasilkan oleh bakteria positif Gram, Bacillus thermoproteolyticus.[2] Ia memerlukan satu ion zink bagi menjamin aktiviti enzim, dan empat ion kalsium bagi kestabilan strukturnya.[3] Termolisin secara khusus memangkinkan hidrolisis ikatan peptida yang mengandungi asid amino hidrofobik. Walau bagaimanapun, termolisin juga digunakan secara meluas bagi pembentukan ikatan peptida melalui tindak balas hidrolisis terbalik.[4] Termolisin ialah ahli yang paling stabil dalam keluarga metaloproteinase yang dihasilkan oleh pelbagai spesies Bacillus. Enzim ini juga dipanggil proteinase "neutral" atau proteinase seperti termolisin (TLPs).
Sintesis
[sunting | sunting sumber]Seperti semua protease luar sel bakteria, termolisin mula-mulanya dihasilkan bakteria sebagai praproenzim.[5] Termolisin disintesis sebagai praproenzim yang terdiri daripada peptida isyarat 28 asid amino panjang, propeptida 204 asid amino panjang dan enzim matang itu sendiri dengan 316 asid amino panjangnya. Peptida isyarat bertindak sebagai isyarat untuk translokasi praprotermolisin ke membran sitoplasma bakteria. Di periplasma, praprotermolisin kemudiannya diproses menjadi protermolisin oleh peptidase isyarat. Untaian itu kemudiannya bertindak sebagai caperon molekul, dan menyebabkan swapemutusan ikatan peptida yang menghubungkan jujukan proprotein dan matang. Protein matang kemudian dirembeskan ke luar sel.[6]
Struktur
[sunting | sunting sumber]Termolisin mempunyai berat molekul 34,600 Da. Struktur keseluruhannya terdiri daripada dua domain sfera dengan celahan dalam melintasi bahagian tengah molekul yang memisahkan kedua-dua domain. Struktur sekunder setiap domain agak berbeza, dengan domain terminal N kebanyakannya terdiri daripada helaian beta, manakala domain terminal C kebanyakannya memiliki struktur heliks alfa. Kedua-dua domain ini disambungkan oleh heliks alfa pusat yang merangkumi asid amino 137-151.[7]
Berbeza dengan kebanyakan protein yang mengalami perubahan konformasi dan penyahaslian apabila dipanaskan, termolisin tidak mengalami sebarang perubahan konformasi ketara sehingga sekurang-kurangnya 70 °C.[8] Kestabilan haba ahli keluarga TLP diukur dari segi suhu T50. Pada suhu ini, pengeraman selama 30 minit mengurangkan aktiviti enzim sebanyak separuh. Termolisin mempunyai nilai T50 86.9 °C, menjadikannya ahli paling stabil haba dalam keluarga TLP.[9] Kajian terhadap sumbangan kalsium terhadap kestabilan termolisin menunjukkan bahawa penyahaktifan haba berlaku dengan pembuangan satu molekul kalsium.[10] Menghalang kalsium ini daripada mula mengikat molekul dengan mutasi tapak pengikatnya mengurangkan kestabilan termolisin sebanyak 7 °C. Walau bagaimanapun, sementara pengikatan kalsium memberi sumbangan besar kepada kestabilan termolisin, apa yang lebih penting untuk kestabilan ialah sekumpulan kecil asid amino domain terminal N yang terletak di permukaan protein.[9] Khususnya, satu fenilalanina (F) di kedudukan asid amino 63 dan prolina (P) di kedudukan asid amino 69 menyumbang ketara terhadap kestabilan termolisin. Penukaran asid amino ini kepada treonina (T) dan alanina (A) masing-masing di proteinase seperti termolisin yang kurang stabil yang dihasilkan oleh Bacillus stearothermophillus (TLP-ste), mengakibatkan pengurangan kestabilan 7 °C (F63→T) dan 6.3 °C (P69→A), dan apabila digabungkan, kestabilan berkurang sebanyak 12.3 °C.[9]
Aplikasi
[sunting | sunting sumber]- Dalam sintesis aspartam, lebih sedikit bahan sampingan pahit terbentuk ketika tindak balas dimangkinkan termolisin.[11]
- Termolisin digunakan dalam penentuan kestabilan protein dalam lisat sel dengan ujian proteolisis selari pantas (FASTpp).[12]
Rujukan
[sunting | sunting sumber]- ^ PDB: 3TMN; Holden HM, Matthews BW (March 1988). "The binding of L-valyl-L-tryptophan to crystalline thermolysin illustrates the mode of interaction of a product of peptide hydrolysis". J. Biol. Chem. 263 (7): 3256–60. doi:10.2210/pdb3tmn/pdb. PMID 3343246.
- ^ Endo, S. (1962). "Studies on protease produced by thermophilic bacteria". J. Ferment. Technol. 40: 346–353.
- ^ Tajima M, Urabe I, dll. (1976). "Role of calcium ions in the thermostability of thermolysin and Bacillus subtilis var. amylosacchariticus neutral protease". Eur. J. Biochem. 64 (1): 243–247. doi:10.1111/j.1432-1033.1976.tb10293.x. PMID 819262.
- ^ Trusek-Holownia A. (2003). "Synthesis of ZAlaPheOMe, the precursor of bitter dipeptide in the two-phase ethyl acetate-water system catalysed by thermolysin". J. Biotechnol. 102 (2): 153–163. doi:10.1016/S0168-1656(03)00024-5. PMID 12697393.
- ^ Yasukawa K, Kusano M, Inouye K (2007). "A new method for the extracellular production of recombinant thermolysin by co-expressing the mature sequence and pro-sequence in Escherichia coli". Protein Eng. Des. Sel. 20 (8): 375–383. doi:10.1093/protein/gzm031. PMID 17616558.
- ^ Inouye K, Kusano M, dll. (2007). Engineering, expression, purification, and production of recombinant thermolysin. Biotechnol. Annu. Rev. Biotechnology Annual Review. 13. m/s. 43–64. doi:10.1016/S1387-2656(07)13003-9. ISBN 978-0-444-53032-5. PMID 17875473.
- ^ Holmes MA, Matthews BW (1982). "Structure of thermolysin refined at 1.6 A resolution". J. Mol. Biol. 160 (4): 623–639. doi:10.1016/0022-2836(82)90319-9. PMID 7175940.
- ^ Matthews BW, Weaver LH, Kester WR (1974). "The conformation of thermolysin". J. Biol. Chem. 249 (24): 8030–8044. doi:10.1016/S0021-9258(19)42067-X. PMID 4214815.
- ^ a b c Eijsink VG, Veltman OR, dll. (1995). "Structural determinants of the stability of thermolysin-like proteinases". Nat. Struct. Biol. 2 (5): 374–379. doi:10.1038/nsb0595-374. PMID 7664094. S2CID 37785818.
- ^ Dahlquist FW, Long JW, Bigbee WL (1976). "Role of Calcium in the thermal stability of thermolysin". Biochemistry. 15 (5): 1103–1111. doi:10.1021/bi00650a024. PMID 814920.
- ^ Yagasaki, Makoto; Hashimoto, Shin-ichi (November 2008). "Synthesis and application of dipeptides; current status and perspectives". Applied Microbiology and Biotechnology. 81 (1): 13–22. doi:10.1007/s00253-008-1590-3. PMID 18795289. S2CID 10200090.
- ^ Minde, David P.; Maurice, Madelon M.; Rüdiger, Stefan G. D. (2012). "Determining Biophysical Protein Stability in Lysates by a Fast Proteolysis Assay, FASTpp". PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
Pautan luar
[sunting | sunting sumber]- Pangkalan data dalam talian MEROPS bagi peptidase dan perencatnya: M04.001
- Thermolysin dalam Tajuk Subjek Perubatan (MeSH) di Perpustakaan Perubatan Negara AS