Grupowanie hierarchiczne
Grupowanie hierarchiczne, hierarchiczna analiza skupień, klasteryzacja hierarchiczna, klastrowanie hierarchiczne – w eksploracji danych i statystyce: metoda analizy skupień, która ma na celu zbudowanie hierarchii klastrów. Służy do dzielenia obserwacji na grupy (klastry) bazując na podobieństwach między nimi. W przeciwieństwie do wielu algorytmów służących do klastrowania w tym wypadku nie jest konieczne wstępne określenie liczby tworzonych klastrów[1]. Strategie tworzenia klastrów hierarchicznych dzielą się zasadniczo na dwa typy[2]:
- metody aglomeracyjne (ang. agglomerative) – każda obserwacja tworzy na początku jednoelementowy klaster. Następnie pary klastrów są scalane, w każdej iteracji algorytmu łączone są z sobą dwa najbardziej zbliżone klastry. Tworzone są tak zwane aglomeracje. W tym typie podczas tworzenia klastrów idzie się w górę hierarchii.
- metody deglomeracyjne (ang. divisive) – początkowo wszystkie obserwacje znajdują się w jednym klastrze. W następnych krokach klastry dzielone są na mniejsze i bardziej jednorodne. Podziały wykonywane są rekursywnie. W czasie tworzenia klastrów idzie się w dół hierarchii.
Algorytmy grupowania hierarchicznego charakteryzują się złożonością obliczeniową O(n³) oraz wymagają O(n²) pamięci, co czyni je mało efektywnymi. Wyniki hierarchicznego grupowania stanowią zestaw zagnieżdżonych klastrów, które są zwykle prezentowane w dendrogramie. Dendrogram jest wielopoziomową hierarchią, w której klastry z jednego poziomu są połączone i tworzą większe klastry na kolejnych poziomach. Umożliwia on określenie poziomu, na który należy wyciąć drzewo w celu wygenerowania odpowiedniej ilości klastrów.
Stosując algorytmy grupowania hierarchicznego, konieczne jest dokonanie pomiaru odległości między punktami. Głównym celem jest to, aby odległości między obserwacjami tego samego klastra były możliwie jak najmniejsze, natomiast odległości między klastrami były jak największe. W hierarchicznym grupowaniu istnieją dwa bardzo ważne parametry: metryka odległości i metoda połączenia.
Metryka
[edytuj | edytuj kod]Zdefiniowanie sposobu określania odległości między obserwacjami jest jednym z najważniejszych aspektów tego algorytmu. Może zdarzyć się, że niedostosowanie odpowiedniej metryki do danych spowoduje otrzymanie bezsensownych wyników klastrowania[3]. Główne miary odległości są następujące:
| Nazwa | Wzór |
|---|---|
| Odległość Euklidesowa | |
| Kwadratowa Odległość Euklidesowa | |
| Odległość Manhattan | |
| Maksymalna Odległość |
Metody połączenia
[edytuj | edytuj kod]Metody połączenia określają, jak definiowana jest odległość między dwoma klastrami. Ważne jest, aby w danym eksperymencie wypróbować kilka metod łączenia oraz porównać ich wyniki. W zależności od zbioru danych, niektóre metody mogą działać lepiej. Poniżej znajduje się lista najczęściej występujących metod połączenia[3]:
| Nazwa | Opis |
|---|---|
| Pojedyncze połączenie | Odległość między dwoma klastrami jest minimalną odległością między obserwacją w jednym klastrze a obserwacją w innym klastrze. Sprawdza się, gdy klastry są wyraźnie oddzielone. |
| Kompletne połączenie | Odległość między dwoma klastrami jest maksymalną odległością między obserwacją w jednym klastrze a obserwacją w innym klastrze. Może być wrażliwy na występowanie outlier’ów. |
| Średnie połączenie | Odległość między dwoma klastrami jest średnią odległością między obserwacją w jednym klastrze a obserwacją w innym klastrze. |
| Połączenie centroidalne | Odległość między dwoma klastrami jest odległością pomiędzy centroidami klastra. |
| Połączenie medianowe | Odległość między dwoma klastrami jest medianą odległości między obserwacją w jednym klastrze a obserwacją w innym klastrze. Zmniejsza wpływ outlier’ów. |
| Połączenie Ward’a | Odległość między dwoma klastrami jest sumą kwadratów odchyleń od punktów do centroidów. Ten sposób dąży do zminimalizowania sumy kwadratów wewnątrz klastra. |
| Połączenie McQuitty’ego | Gdy dwa klastry A i B zostaną połączone, odległość do nowego klastra C jest średnią odległości A i B do C. W ten sposób odległość zależy od kombinacji klastrów zamiast indywidualnych obserwacji w klastrach. |
Metody aglomeracyjne
[edytuj | edytuj kod]Metody aglomeracyjne są najbardziej popularną metodą grupowania hierarchicznego[1]. W literaturze można spotkać się również z nazwą AGNES (z ang. agglomerative nesting). Potocznie mówi się, że aglomeracyjne grupowanie działa w sposób „oddolny” (ang. bottom-up). Na początku algorytmu każda obserwacja jest traktowana jako pojedynczy klaster. Następnie pary klastrów są sukcesywnie łączone, aż do momentu gdy wszystkie klastry zostaną scalone w jeden duży klaster zawierający wszystkie obiekty.
Przebieg algorytmu metody aglomeracyjnej można przedstawić następująco:
- Wyszukiwane są dwa najbliższe punkty w zbiorze danych
- Znalezione punkty są łączone – od tego momentu będą traktowane jako jeden punkt
- Proces rozpoczyna się od nowa. Od teraz wykorzystywany jest nowy zbiór obserwacji utworzony w poprzednich krokach
Decyzja o połączeniu dwóch klastrów w jedne jest nieodwracalna – tego klastra nie można rozdzielić już w następnej iteracji algorytmu.
Na grafikach przedstawiono przebieg procesu grupowania aglomeracyjnego. Na pierwszym zdjęciu widać zbiór danych, który zostanie poddany klasteryzacji. W poniższym przykładzie mamy sześć elementów {a} {b} {c} {d} {e} i {f}. Pierwszym krokiem działania algorytmu jest określenie, które elementy należy scalić w klastrze. Zwykle chcemy wziąć dwa najbliższe sobie elementy, zgodnie z wybraną odległością. W przykładzie wybór podejmowany jest na podstawie odległości Euklidesowej, najbardziej intuicyjnej. Jedną z możliwości porównywania odległości między sobą jest zbudowanie macierzy odległości na tym etapie, gdzie liczba w i-tym wierszu j-tej kolumny jest odległością między i-tym i j-tym elementem. Następnie, w miarę rozwoju klastrów, wiersze i kolumny macierzy są scalane, w momencie gdy scalane są klastry. Odległości powinny być wtedy aktualizowane.
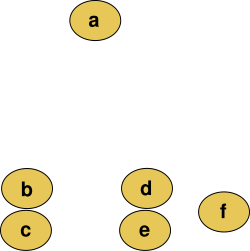
Łatwo zauważyć, że najbliżej siebie znajdują się elementy {b} i {c} oraz {d} i {e}. W pierwszej fazie działania algorytmu połączyliśmy dwa najbliższe elementy {b} i {c}. Od teraz mamy następujące klastry {a}, {b, c}, {d}, {e} i {f}. Naszym celem jest ich dalsze scalanie. Aby to zrobić, musimy określić odległość między klastrem {b c}, a pozostałymi klastrami (które stanowią na razie pojedyncze obserwacje). Następnym etapem będzie połączenie elementów {d} i {e}. Kolejne iteracje będą wykonywane aż do momentu gdy wszystkie sześć elementów znajdzie się w jednym klastrze.
Poniżej przedstawiono dendrogram, który przedstawia rezultat działania algorytmu grupowania hierarchicznego.
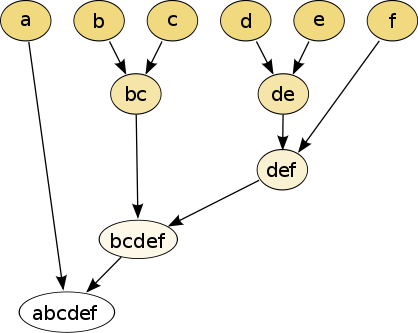
Metody deglomeracyjne
[edytuj | edytuj kod]Metody deglomeracyjne są przeciwnością metod aglomeracyjnych. Znane są również jako DIANA (od ang. divise analysis) i działa w sposób „z góry na dół” (ang. top-down). Działanie algorytmu zaczyna się w momencie gdy wszystkie obserwacje znajdują się w jednym klastrze. W każdej kolejnej iteracji najbardziej niejednorodny klaster jest dzielony na dwa klastry. Proces ten trwa aż do momentu gdy wszystkie obserwacje znajdują się we własnym klastrze (co oznacza, że liczba klastrów jest równa liczbie obserwacji). Należy zauważyć, że klastrowanie aglomeracyjne dobrze sprawdza się w dzieleniu małych klastrów. Natomiast klastrowanie deglomeracyjne jest dobre w identyfikowaniu dużych klastrów.
Przykład grupowania hierarchicznego w języku R
[edytuj | edytuj kod]W niniejszym paragrafie przedstawione zostanie działanie grupowania hierarchicznego w języku R. Jak już wspomniano, algorytm ten nie działa dobrze na dużych zbiorach danych. Zostanie on przetestowany na bardzo małym zbiorze „animals” dostępnego w pakiecie „clusters”. Ten zestaw danych zawiera opis 20 różnych zwierząt przy pomocy 6 charakterystyk.
Pierwszym etapem jest wczytanie danych oraz ich uporządkowanie. Dane, na których działa algorytm grupowania hierarchicznego powinny być przedstawione w postaci macierzy numeryczną, której wiersze reprezentują poszczególne obserwacje natomiast kolumny reprezentują zmienne (czyli w przypadku zestawu „animals” będziemy mieli do czynienia z macierzą 20x6).
Zalecana jest standaryzacja zmiennych przed kolejnymi etapami analizy. Standaryzacja sprawia, że zmienne są porównywalne. W tym celu została zastosowana funkcja scale() w języku R.
# Load the data
animals <- cluster::animals
colnames(animals) <- c("warm-blooded",
"can fly",
"vertebrate",
"endangered",
"live in groups",
"have hair")
Poniżej przedstawiono fragment zestawu danych.
> head(animals)
warm-blooded can fly vertebrate endangered live in groups have hair
ant 1 1 1 1 2 1
bee 1 2 1 1 2 2
cat 2 1 2 1 1 2
cpl 1 1 1 1 1 2
chi 2 1 2 2 2 2
cow 2 1 2 1 2 2
# Standardize the data
df<-scale(animals)
head(df, nrow = 6)
Po ustandaryzowaniu danych, prezentują się one następująco:
> head(df, nrow = 6)
warm-blooded can fly vertebrate endangered live in groups have hair
ant -0.9746794 -0.4873397 -1.4888474 -0.6871843 0.7164977 -0.8816307
bee -0.9746794 1.9493589 -1.4888474 -0.6871843 0.7164977 1.0775487
cat 0.9746794 -0.4873397 0.6380775 -0.6871843 -1.3135792 1.0775487
cpl -0.9746794 -0.4873397 -1.4888474 -0.6871843 -1.3135792 1.0775487
chi 0.9746794 -0.4873397 0.6380775 1.3743685 0.7164977 1.0775487
cow 0.9746794 -0.4873397 0.6380775 -0.6871843 0.7164977 1.0775487
Aby zdecydować, które obserwacje powinny być połączone lub podzielone, konieczny jest wybór metody pomiaru podobieństwa między obiektami. Tak jak już wspomniano, istnieje wiele metod obliczania informacji o podobieństwie, w tym odległości euklidesowej. W języku R można użyć funkcji dist() do obliczenia odległości między każdą parą obiektu w zbiorze danych. Wynikiem tej funkcji jest matryca odległości. Domyślnie funkcja dist() oblicza odległość euklidesową między obiektami; jednak możliwe jest wskazanie innych danych za pomocą metody argumentu. Aby łatwo zobaczyć informacje o odległości między obiektami, ponownie sformatowano wyniki w postać macierzową za pomocą funkcji as.matrix(). W tej macierzy wartość w komórce utworzonej przez wiersz i oraz kolumnę j, reprezentuje odległość między obiektem i oraz obiektem j w oryginalnym zbiorze danych. Na przykład element 1,1 reprezentuje odległość między obiektem 1 a samym sobą (który wynosi zero). Element 1,2 reprezentuje odległość między obiektem 1 a obiektem 2 i tak dalej.
# Compute the dissimilarity matrix
# df = the standardized data
res.dist <- dist(df, method = "euclidean")
> as.matrix(res.dist)[1:6, 1:6]
ant bee cat cpl chi cow
ant 0.000000 3.126641 4.035270 2.821276 4.051197 3.487434
bee 3.126641 0.000000 4.287484 3.171547 4.302477 3.776415
cat 4.035270 4.287484 0.000000 2.885101 2.893305 2.030077
cpl 2.821276 3.171547 2.885101 0.000000 4.085954 3.527750
chi 4.051197 4.302477 2.893305 4.085954 0.000000 2.061553
cow 3.487434 3.776415 2.030077 3.527750 2.061553 0.000000
Mając obliczoną odległość między klastrami, należy teraz połączyć te znajdujące się najbliżej siebie. Funkcja łączenia pobiera informacje o odległości, zwrócone przez funkcję dist() – w postaci macierzy odległości res.dist – i grupuje pary obiektów w klastry na podstawie ich podobieństwa. Następnie te nowo utworzone klastry są łączone ze sobą, aby utworzyć większe klastry. Ten proces jest powtarzany aż do momentu, gdy wszystkie obiekty w oryginalnym zestawie danych zostaną ze sobą połączone hierarchicznie drzewo. W niniejszym przykładzie, do obliczenia odległości między klastrami zastosowano odległość Ward’a.
Funkcja hclust() pobiera następujące parametry:
- d: macierz odległości – zwrócona przez funkcję dist().
- method: Metoda połączenia używana do obliczania odległości między klastrami. Dozwolone wartości to: „Ward.D”, „Ward.D2”, „Single”, „Complete”, „average”, „mcquitty”, „median” lub „centroid”.
res.hc <- hclust(d = res.dist, method = "ward.D2")
# cex: label size
library("factoextra")
fviz_dend(res.hc, cex = 0.5)
res.hc2 <- hclust(res.dist, method = "average")
# Cut tree into 4 groups
grp <- cutree(res.hc, k = 4)
Dendrogram prezentujący podział tego zbioru danych na klastry może być wygenerowany w R przy użyciu funkcji fviz.dend() z pakietu „factoextra”, gdzie res.hc jest wynikiem funkcji hclust().

W dendrogramie pokazanym powyżej każdy liść odpowiada jednej obserwacji. Wysokość połączenia, podana na osi pionowej, wskazuje podobieństwo / dystans (dis) między dwoma obiektami / klastrami. Im większa wysokość połączenia między obserwacjami, tym mniej podobne są do siebie obiekty. Nie można użyć bliskości między dwoma obserwacjami wzdłuż osi poziomej jako kryterium ich podobieństwa.
Jednym z problemów związanych z hierarchicznym grupowaniem jest to, że wynikiem nie jest informacja na ile klastrów należy podzielić obserwacje lub gdzie można przeciąć dendrogram w celu utworzenia klastrów. Możliwe jest przecięcie drzewa na określonej wysokości służy do tego funkcja cutree(). Zwraca ona wektor zawierający numer klastra każdej z obserwacji.
Drzewo hierarchiczne, które jest wynikiem działania algorytmu następnie podzielono na 4. Oznacza to, że w ostatecznym rozwiązaniu otrzymamy obserwacje podzielone na 4 klastry. Poniżej przedstawiono wybrane gatunki zwierząt wraz z ich przynależnością do konkretnego klastra. Możemy zauważyć na przykład, że kurczak (chi) oraz krowa (cow) trafiły do tego samego klastra. Przedstawiono też liczebność poszczególnych klastrów. Najwięcej obserwacji przyporządkowano do trzeciego klastra (6).
> head(grp, n = 8)
ant bee cat cpl chi cow duc eag
1 2 3 2 3 3 4 4
# Number of members in each cluster
> table(grp)
grp
1 2 3 4
5 4 6 5
fviz_dend(res.hc, k = 4, # Cut in four groups
cex = 0.5, # label size
k_colors = c("#2E9FDF", "#00AFBB", "#E7B800", "#FC4E07"),
color_labels_by_k = TRUE, # color labels by groups
rect = TRUE # Add rectangle around groups
)
Poniżej przedstawiono dendrogram podzielony na 4 klastry:

Przypisy
[edytuj | edytuj kod]- ↑ a b Alboukadel Kassambara: Practical Guide To Cluster Analysis in R. STHDA, 2017.
- ↑ Rokach, Lior, and Oded Maimon: „Clustering methods.” Data mining and knowledge discovery handbook. Springer, 2005, s. 321–352.
- ↑ a b Erik Rodríguez Pacheco: Unsupervised Learning with R. Packt Publishing Ltd., 2015.



