Partículas interferentes defeituosas
Partículas interferentes defeituosas (PIDs), também conhecidas como vírus interferentes defeituosos, são mutantes de vírus gerados espontaneamente nos quais uma porção crítica do genoma da partícula foi perdida devido à replicação defeituosa ou recombinação não homóloga . [2] [3] Presume-se que o mecanismo de sua formação seja resultado da troca de moldes durante a replicação do genoma viral, embora mecanismos não replicativos envolvendo a ligação direta de fragmentos de RNA genômicos também tenham sido propostas. [4] [5] As partículas interferentes defeituosas são derivadas e associadas ao seu vírus parental, e as partículas são classificadas como PIDs se forem tornadas não infecciosas devido à perda ou dano grave de pelo menos um gene essencial do vírus como resultado da defecção. [6] Uma partícula interferentes defeituosas geralmente ainda pode penetrar nas células hospedeiras, mas requer outra partícula de vírus totalmente funcional (o vírus 'auxiliar') para co-infectar uma célula com ela, a fim de fornecer os fatores perdidos. [7] [8]
As partículas interferentes defeituosas foram observadas pela primeira vez na década de 1950 por Von Magnus e Schlesinger, ambos trabalhando com vírus da gripe. [9] No entanto, a evidência direta de PIDs só foi encontrada na década de 1960 por Hackett que notou a presença de partículas 'atarracadas' do vírus da estomatite vesicular em micrografias eletrônicas [10] e a formalização da terminologia de partículas interferentes defeituosas PIDs foi em 1970 por Huang e Baltimore. [11] PIDs podem ocorrer em quase todas as classes de vírus de DNA e RNA, tanto em ambientes clínicos quanto laboratoriais, incluindo poliovírus, coronavírus SARS, sarampo, alfavírus, vírus sincicial respiratório e vírus influenza . [12] [13] [14] [15] [16] [17] [18] [19]
Deserção
[editar | editar código-fonte]Partículas interferentes defeituosas (PIDs) são um fenômeno natural que pode ser recriado em condições experimentais no laboratório e também pode ser sintetizado para uso experimental. Eles são produzidos espontaneamente por replicação viral propensa a erros, algo particularmente prevalente em vírus de RNA sobre vírus de DNA devido à enzima usada (replicase ou RNA polimerase dependente de RNA) [6] [20] Os genomas de interferentes defeituosas normalmente retêm as sequências terminais necessárias para o reconhecimento pelas polimerases virais e as sequências para o empacotamento de seu genoma em novas partículas, mas pouco mais. [21] [22] O tamanho do evento de deleção genômica pode variar muito, com um exemplo em um PIDs derivado do vírus da raiva exibindo uma deleção de 6,1 kb. [23] Em outro exemplo, o tamanho de vários genomas de vírus de plantas Interferentes Defeituosas/DNA variou de um décimo do tamanho do genoma original a metade. [24]
Interferência
[editar | editar código-fonte]As partículas são consideradas interferentes quando afetam a função do vírus parental através da inibição competitiva [6] durante a coinfecção. Em outras palavras, vírus defeituosos e não defeituosos se replicam simultaneamente, mas quando as partículas defeituosas aumentam, a quantidade de vírus não defeituosos replicados diminui. A extensão da interferência depende do tipo e tamanho da defecção no genoma; grandes deleções de dados genômicos permitem a rápida replicação do genoma defeituoso. [21] Durante a coinfecção de uma célula hospedeira, uma razão crítica será eventualmente alcançada em que mais fatores virais estão sendo usados para produzir as PIDs não infecciosas do que partículas infecciosas. [21] Partículas defeituosas e genomas defeituosos também demonstraram estimular as respostas imunes inatas do hospedeiro e sua presença durante uma infecção viral se correlaciona com a força da resposta antiviral. [12] No entanto, em alguns vírus, como o SARS-CoV-2, o efeito da inibição competitiva por partículas interferentes reduz as respostas imunes inatas mediadas por vírus e a inflamação produzindo um efeito terapêutico. [25]
Essa natureza interferente está se tornando cada vez mais importante para pesquisas sobre terapias virais. [26] [27] Pensa-se que, devido à sua especificidade, as PIDs serão direcionados para os locais de infecção. Em um exemplo, os cientistas usaram PIDs para criar "vírus protetores", que atenuaram a patogenicidade de uma infecção por influenza A em camundongos, induzindo uma resposta de interferon, a um ponto que não era mais letal. [28] Para SARS-CoV-2, o efeito de interferência foi usado para gerar partículas de interferência terapêutica (TIPs) que reduziram a patogênese e protegeram os hamsters de doenças graves. [29]
Patogênese
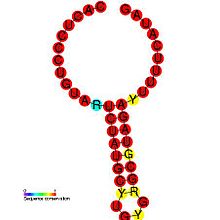
[editar | editar código-fonte]Partículas interferentes defeituosas (PIDs) demonstraram desempenhar um papel na patogênese de certos vírus. Um estudo demonstra a relação entre um patógeno e sua variante defeituosa, mostrando como a regulação da produção de interferentes defeituosas permitiu que o vírus atenuasse sua própria replicação infecciosa, diminuindo a carga viral e, assim, aumentando sua eficiência parasitária, evitando que o hospedeiro morresse muito rápido. [30] Isso também fornece ao vírus mais tempo para se espalhar e infectar novos hospedeiros. A geração de PIDs é regulada dentro de vírus: o elemento de replicação de ação cis Coronavirus SL-III (mostrado na imagem) é uma estrutura genômica de ordem superior implicada na mediação da produção de PIDs em coronavírus bovino, com homólogos aparentes detectados em outros grupos de coronavírus . [31] Uma introdução mais aprofundada pode ser encontrada no trabalho de Alice Huang e David Baltimore de 1970. [32]
Tipos de genomas de RNA defeituosos
[editar | editar código-fonte]- As deserções de deleções ocorrem quando um fragmento do modelo é ignorado. Exemplos deste tipo de defecção podem ser encontrados no vírus da murcha do tomateiro e no vírus Flock House. [33] [22]
- As deserções de snapbacks ocorrem quando a replicase transcreve parte de uma fita e usa essa nova fita como modelo. O resultado disso pode produzir um grampo de cabelo. Defecções de snapback foram observadas no vírus da estomatite vesicular . [34]
- As deserções de enclave são quando a polimerase carrega uma fita parcialmente feita e depois volta para transcrever a extremidade 5', formando a forma de enclave. As defecções de enclave são encontradas em vírus da gripe. [35]
- As deserções compostas ocorrem quando uma exclusão e uma deserção de snapback acontecem juntas.
- Genoma em mosaico ou interferentes defeituosos complexos, em que as várias regiões podem vir do mesmo genoma do vírus auxiliar, mas na ordem errada; de diferentes segmentos do genoma auxiliar, ou pode incluir segmentos de RNA hospedeiro. Duplicações também podem ocorrer. [3]
Pesquisar
[editar | editar código-fonte]Pesquisas foram conduzidas por virologistas para aprender mais sobre a interferência na infecção de células hospedeiras e como os genomas de DI poderiam funcionar como agentes antivirais imunoestimuladores . [3] Outro ramo de pesquisa buscou o conceito de engenharia de DIPs em partículas de interferência terapêutica antiviral (TIPs), [36] um conceito puramente teórico até recentemente. [37] Um artigo de 2014 descreve o trabalho pré-clínico para testar a eficácia imunoestimuladora de um PIDs contra os vírus da gripe induzindo as respostas imunes antivirais inatas (ou seja, interferon). [38] Trabalhos subsequentes testaram a eficácia pré-clínica de terapia usando PIDs contra o HIV [39] e SARS-CoV-2 . [25] Descobriu-se também que os DI-RNAs auxiliam pela primeira vez na infecção de fungos através de vírus da família Partitiviridae, o que abre espaço para um trabalho mais interdisciplinar. [20]
Várias ferramentas como viremia [40] e genomas de interferente viral defectivo [41] foram desenvolvidas para ajudar a detectar genomas virais defeituosos em dados de sequenciamento de próxima geração e abordagens de alto rendimento, como sequenciamento de biblioteca de exclusão aleatória (RanDeL-Seq), [42] permitem o mapeamento racional dos elementos genéticos virais que são necessários para a propagação de partículas interferente defectitvo.
Referências
[editar | editar código-fonte]- ↑ Raman S, Bouma P, Williams GD, Brian DA (junho de 2003). «Stem-loop III in the 5' untranslated region is a cis-acting element in bovine coronavirus defective interfering RNA replication». Journal of Virology. 77 (12): 6720–6730. PMC 156170Acessível livremente. PMID 12767992. doi:10.1128/JVI.77.12.6720-6730.2003
- ↑ White KA, Morris TJ (janeiro de 1994). «Nonhomologous RNA recombination in tombusviruses: generation and evolution of defective interfering RNAs by stepwise deletions». Journal of Virology. 68 (1): 14–24. PMC 236259
 . PMID 8254723. doi:10.1128/JVI.68.1.14-24.1994
. PMID 8254723. doi:10.1128/JVI.68.1.14-24.1994
- ↑ Pathak KB, Nagy PD (dezembro de 2009). «Defective Interfering RNAs: Foes of Viruses and Friends of Virologists». Viruses. 1 (3): 895–919. PMC 3185524
 . PMID 21994575. doi:10.3390/v1030895
. PMID 21994575. doi:10.3390/v1030895
- ↑ Gmyl AP, Belousov EV, Maslova SV, Khitrina EV, Chetverin AB, Agol VI (novembro de 1999). «Nonreplicative RNA recombination in poliovirus». Journal of Virology. 73 (11): 8958–8965. PMC 112927
 . PMID 10516001. doi:10.1128/JVI.73.11.8958-8965.1999
. PMID 10516001. doi:10.1128/JVI.73.11.8958-8965.1999
- ↑ a b c Pathak KB, Nagy PD (dezembro de 2009). «Defective Interfering RNAs: Foes of Viruses and Friends of Virologists». Viruses. 1 (3): 895–919. PMC 3185524
 . PMID 21994575. doi:10.3390/v1030895
. PMID 21994575. doi:10.3390/v1030895
- ↑ Makino S, Shieh CK, Soe LH, Baker SC, Lai MM (outubro de 1988). «Primary structure and translation of a defective interfering RNA of murine coronavirus». Virology. 166 (2): 550–560. PMC 7131284
 . PMID 2845661. doi:10.1016/0042-6822(88)90526-0
. PMID 2845661. doi:10.1016/0042-6822(88)90526-0
- ↑ Oxford Textbook of Zoonoses: Biology, Clinical Practice, and Public Health Control. [S.l.]: OUP Oxford. 2011. pp. 399–400. ISBN 978-0-19-857002-8. doi:10.1093/med/9780198570028.001.0001
- ↑ Gard S, Von Magnus P, Svedmyr A, Birch-Andersen A (outubro de 1952). «Studies on the sedimentation of influenza virus». Archiv für die Gesamte Virusforschung. 4 (5): 591–611. PMID 14953289. doi:10.1007/BF01242026
- ↑ Hackett AJ (setembro de 1964). «A possible morphologic basis for the autointerference phenomenon in vesicular stomatitis virus». Virology. 24 (1): 51–59. PMID 14208902. doi:10.1016/0042-6822(64)90147-3
- ↑ Huang AS, Baltimore D (abril de 1970). «Defective viral particles and viral disease processes». Nature. 226 (5243): 325–327. Bibcode:1970Natur.226..325H. PMID 5439728. doi:10.1038/226325a0
- ↑ a b Sun Y, Jain D, Koziol-White CJ, Genoyer E, Gilbert M, Tapia K, et al. (setembro de 2015). «Immunostimulatory Defective Viral Genomes from Respiratory Syncytial Virus Promote a Strong Innate Antiviral Response during Infection in Mice and Humans». PLOS Pathogens. 11 (9): e1005122. PMC 4559413Acessível livremente. PMID 26336095. doi:10.1371/journal.ppat.1005122
- ↑ Dimmock NJ, Dove BK, Scott PD, Meng B, Taylor I, Cheung L, et al. (2012). «Cloned defective interfering influenza virus protects ferrets from pandemic 2009 influenza A virus and allows protective immunity to be established». PLOS ONE. 7 (12): e49394. Bibcode:2012PLoSO...749394D. PMC 3521014
 . PMID 23251341. doi:10.1371/journal.pone.0049394
. PMID 23251341. doi:10.1371/journal.pone.0049394
- ↑ Saira K, Lin X, DePasse JV, Halpin R, Twaddle A, Stockwell T, et al. (julho de 2013). «Sequence analysis of in vivo defective interfering-like RNA of influenza A H1N1 pandemic virus». Journal of Virology. 87 (14): 8064–8074. PMC 3700204
 . PMID 23678180. doi:10.1128/JVI.00240-13
. PMID 23678180. doi:10.1128/JVI.00240-13
- ↑ Petterson E, Guo TC, Evensen Ø, Mikalsen AB (novembro de 2016). «Experimental piscine alphavirus RNA recombination in vivo yields both viable virus and defective viral RNA». Scientific Reports. 6. 36317 páginas. Bibcode:2016NatSR...636317P. PMC 5090867
 . PMID 27805034. doi:10.1038/srep36317
. PMID 27805034. doi:10.1038/srep36317
- ↑ Cattaneo R, Schmid A, Eschle D, Baczko K, ter Meulen V, Billeter MA (outubro de 1988). «Biased hypermutation and other genetic changes in defective measles viruses in human brain infections». Cell. 55 (2): 255–265. PMC 7126660
 . PMID 3167982. doi:10.1016/0092-8674(88)90048-7
. PMID 3167982. doi:10.1016/0092-8674(88)90048-7
- ↑ Makino S, Yokomori K, Lai MM (dezembro de 1990). «Analysis of efficiently packaged defective interfering RNAs of murine coronavirus: localization of a possible RNA-packaging signal». Journal of Virology. 64 (12): 6045–6053. PMC 248778
 . PMID 2243386. doi:10.1128/JVI.64.12.6045-6053.1990
. PMID 2243386. doi:10.1128/JVI.64.12.6045-6053.1990
- ↑ Lundquist RE, Sullivan M, Maizel JV (novembro de 1979). «Characterization of a new isolate of poliovirus defective interfering particles». Cell. 18 (3): 759–769. PMID 229964. doi:10.1016/0092-8674(79)90129-6
- ↑ Stauffer Thompson KA, Rempala GA, Yin J (abril de 2009). «Multiple-hit inhibition of infection by defective interfering particles». The Journal of General Virology. 90 (Pt 4): 888–899. PMC 2889439
 . PMID 19264636. doi:10.1099/vir.0.005249-0
. PMID 19264636. doi:10.1099/vir.0.005249-0
- ↑ a b Chiba S, Lin YH, Kondo H, Kanematsu S, Suzuki N (fevereiro de 2013). «Effects of defective interfering RNA on symptom induction by, and replication of, a novel partitivirus from a phytopathogenic fungus, Rosellinia necatrix». Journal of Virology. 87 (4): 2330–2341. PMC 3571465
 . PMID 23236074. doi:10.1128/JVI.02835-12
. PMID 23236074. doi:10.1128/JVI.02835-12
- ↑ a b c «Innate and intrinsic immunity». Introduction to Modern Virology. [S.l.]: John Wiley & Sons. 2015. pp. 201–217. ISBN 978-1-119-09453-1
- ↑ a b Resende R, de Haan P, van de Vossen E, de Avila AC, Goldbach R, Peters D (outubro de 1992). «Defective interfering L RNA segments of tomato spotted wilt virus retain both virus genome termini and have extensive internal deletions». The Journal of General Virology. 73 (10): 2509–2516. PMID 1402797. doi:10.1099/0022-1317-73-10-2509

- ↑ Conzelmann KK, Cox JH, Thiel HJ (outubro de 1991). «An L (polymerase)-deficient rabies virus defective interfering particle RNA is replicated and transcribed by heterologous helper virus L proteins». Virology. 184 (2): 655–663. PMID 1887588. doi:10.1016/0042-6822(91)90435-e
- ↑ Patil BL, Dasgupta I (2006). «Defective Interfering Dnas of Plant Viruses.». Critical Reviews in Plant Sciences. 25 (1): 47–64. doi:10.1080/07352680500391295
- ↑ a b Chaturvedi S, Vasen G, Pablo M, Chen X, Beutler N, Kumar A, et al. (dezembro de 2021). «Identification of a therapeutic interfering particle-A single-dose SARS-CoV-2 antiviral intervention with a high barrier to resistance». Cell. 184 (25): 6022–6036.e18. doi:10.1016/j.cell.2021.11.004
- ↑ Weinberger LS, Schaffer DV, Arkin AP (setembro de 2003). «Theoretical design of a gene therapy to prevent AIDS but not human immunodeficiency virus type 1 infection». Journal of Virology. 77 (18): 10028–10036. PMC 224590
 . PMID 12941913. doi:10.1128/jvi.77.18.10028-10036.2003
. PMID 12941913. doi:10.1128/jvi.77.18.10028-10036.2003
- ↑ Thompson KA, Yin J (setembro de 2010). «Population dynamics of an RNA virus and its defective interfering particles in passage cultures». Virology Journal. 7. 257 páginas. PMC 2955718
 . PMID 20920247. doi:10.1186/1743-422X-7-257
. PMID 20920247. doi:10.1186/1743-422X-7-257
- ↑ Easton AJ, Scott PD, Edworthy NL, Meng B, Marriott AC, Dimmock NJ (março de 2011). «A novel broad-spectrum treatment for respiratory virus infections: influenza-based defective interfering virus provides protection against pneumovirus infection in vivo» (PDF). Vaccine. 29 (15): 2777–2784. PMID 21320545. doi:10.1016/j.vaccine.2011.01.102
- ↑ Villanueva MT (dezembro de 2021). «Interfering viral-like particles inhibit SARS-CoV-2 replication». Nature Reviews. Drug Discovery. 21 (1): d41573–021–00205-5. doi:10.1038/d41573-021-00205-5
- ↑ Lukhovitskaya NI, Thaduri S, Garushyants SK, Torrance L, Savenkov EI (junho de 2013). «Deciphering the mechanism of defective interfering RNA (DI RNA) biogenesis reveals that a viral protein and the DI RNA act antagonistically in virus infection». Journal of Virology. 87 (11): 6091–6103. PMC 3648117
 . PMID 23514891. doi:10.1128/JVI.03322-12
. PMID 23514891. doi:10.1128/JVI.03322-12
- ↑ Raman S, Bouma P, Williams GD, Brian DA (junho de 2003). «Stem-loop III in the 5' untranslated region is a cis-acting element in bovine coronavirus defective interfering RNA replication». Journal of Virology. 77 (12): 6720–6730. PMC 156170
 . PMID 12767992. doi:10.1128/JVI.77.12.6720-6730.2003
. PMID 12767992. doi:10.1128/JVI.77.12.6720-6730.2003
- ↑ Huang AS, Baltimore D (abril de 1970). «Defective viral particles and viral disease processes». Nature. 226 (5243): 325–327. Bibcode:1970Natur.226..325H. PMID 5439728. doi:10.1038/226325a0
- ↑ Jaworski E, Routh A (maio de 2017). «Parallel ClickSeq and Nanopore sequencing elucidates the rapid evolution of defective-interfering RNAs in Flock House virus». PLOS Pathogens. 13 (5): e1006365. PMC 5435362
 . PMID 28475646. doi:10.1371/journal.ppat.1006365
. PMID 28475646. doi:10.1371/journal.ppat.1006365
- ↑ Schubert M, Lazzarini RA (fevereiro de 1981). «Structure and origin of a snapback defective interfering particle RNA of vesicular stomatitis virus». Journal of Virology. 37 (2): 661–672. PMC 171054
 . PMID 6261012. doi:10.1128/JVI.37.2.661-672.1981
. PMID 6261012. doi:10.1128/JVI.37.2.661-672.1981
- ↑ Fodor E, Pritlove DC, Brownlee GG (junho de 1994). «The influenza virus panhandle is involved in the initiation of transcription». Journal of Virology. 68 (6): 4092–4096. PMC 236924
 . PMID 8189550. doi:10.1128/JVI.68.6.4092-4096.1994
. PMID 8189550. doi:10.1128/JVI.68.6.4092-4096.1994
- ↑ Metzger VT, Lloyd-Smith JO, Weinberger LS (março de 2011). «Autonomous targeting of infectious superspreaders using engineered transmissible therapies». PLOS Computational Biology. 7 (3): e1002015. Bibcode:2011PLSCB...7E2015M. PMC 3060167
 . PMID 21483468. doi:10.1371/journal.pcbi.1002015
. PMID 21483468. doi:10.1371/journal.pcbi.1002015
- ↑ Gladstone Institutes. «A New Class of Antiviral Therapy Could Treat COVID-19». www.prnewswire.com (em inglês). Consultado em 28 de dezembro de 2021
- ↑ Dimmock NJ, Easton AJ (maio de 2014). «Defective interfering influenza virus RNAs: time to reevaluate their clinical potential as broad-spectrum antivirals?». Journal of Virology. 88 (10): 5217–5227. PMC 4019098
 . PMID 24574404. doi:10.1128/JVI.03193-13
. PMID 24574404. doi:10.1128/JVI.03193-13
- ↑ Tanner EJ, Jung SY, Glazier J, Thompson C, Zhou Y, Martin B, Son HI, Riley JL, Weinberger LS (30 de outubro de 2019). «Discovery and Engineering of a Therapeutic Interfering Particle (TIP): a combination self-renewing antiviral» (em inglês). doi:10.1101/820456
- ↑ Routh A, Johnson JE (janeiro de 2014). «Discovery of functional genomic motifs in viruses with ViReMa-a Virus Recombination Mapper-for analysis of next-generation sequencing data». Nucleic Acids Research. 42 (2): e11. PMC 3902915
 . PMID 24137010. doi:10.1093/nar/gkt916
. PMID 24137010. doi:10.1093/nar/gkt916
- ↑ Beauclair G, Mura M, Combredet C, Tangy F, Jouvenet N, Komarova AV (outubro de 2018). «DI-tector: defective interfering viral genomes' detector for next-generation sequencing data». RNA. 24 (10): 1285–1296. PMC 6140465
 . PMID 30012569. doi:10.1261/rna.066910.118
. PMID 30012569. doi:10.1261/rna.066910.118
- ↑ Notton T, Glazier JJ, Saykally VR, Thompson CE, Weinberger LS (janeiro de 2021). «RanDeL-Seq: a High-Throughput Method to Map Viral cis- and trans-Acting Elements». mBio. 12 (1): e01724–20. PMC 7845639
 . doi:10.1128/mBio.01724-20
. doi:10.1128/mBio.01724-20