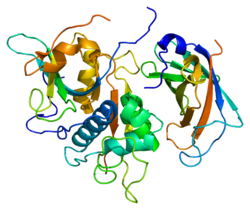
组织蛋白酶C
| 组织蛋白酶C排斥域 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 重新测定人二肽基肽酶i(组织蛋白酶 c)的天然结构 | |||||||||
| 鑑定 | |||||||||
| 標誌 | CathepsinC_exc | ||||||||
| Pfam | PF08773(旧版) | ||||||||
| InterPro | IPR014882 | ||||||||
| SCOP | 1k3b / SUPFAM | ||||||||
| |||||||||
组织蛋白酶C(EC 3.4.14.1)也称为二肽基肽酶I(DPP-I,酶学委员会接受的名字),是一种溶酶体外切半胱氨酸蛋白酶,属于肽酶C1蛋白质家族,是半胱氨酸组织蛋白酶的一个亚组。在人类中,它由CTSC基因编码。[6][7]
功能
[编辑]组织蛋白酶C似乎是激活免疫/炎症细胞中许多丝氨酸蛋白酶的中心协调因子。
组织蛋白酶C催化从蛋白质和肽底物的N端切除二肽,除非
结构
[编辑]编码大鼠、人、鼠类、牛、狗和两种血吸虫组织蛋白酶C的cDNA已被克隆和测序,表明该酶是高度保守的。[8]人和大鼠组织蛋白酶C的cDNA编码前体(前组织蛋白酶C),包括24个残基的信号肽、205个(大鼠组织蛋白酶C)或206个(人组织蛋白酶C)残基的前区和233个残基的催化结构域,其中含有催化残基,与木瓜蛋白酶和许多其他组织蛋白酶的成熟氨基酸序列有30至40%的相同性,包括组织蛋白酶B、H、K、L和S。[9]
通过多肽链的至少四次切割,将已翻译的前组织蛋白酶原C加工成成熟形式。信号肽在酶原(组织蛋白酶原C)的易位或分泌过程中被去除,并且保留在成熟酶中的大的N端前区片段(也称为排斥域)[10]与催化分离通过切除前区的一个较小的C端部分,称为激活肽。通过催化结构域的切割产生约164个残基的重链和约69个残基的轻链。
与木瓜蛋白酶家族的其他成员不同,成熟的组织蛋白酶C由四个亚基组成,每个亚基由N端前区片段、重链和轻链组成。前区片段和重链都被糖基化。
临床意义
[编辑]编码蛋白质的缺陷已被证明是导致掌跖角化-牙周破坏综合征的原因,[11][12]这是一种以掌跖角化和牙周炎为特征的常染色体隐性遗传病。
组织蛋白酶C是激活炎症细胞中颗粒丝氨酸肽酶的关键酶,例如中性粒细胞中的弹性蛋白酶和组织蛋白酶G,以及肥大细胞中的糜酶和类胰蛋白酶。在许多炎性疾病中,例如类风湿性关节炎、慢性阻塞性肺病、炎症性肠病、哮喘、败血症和囊性纤维化,发病机制的很大一部分是由这些炎性蛋白酶中的一些活性增加引起的。一旦被组织蛋白酶C激活,蛋白酶就能够降解各种细胞外基质成分,从而导致组织损伤和慢性炎症。
参考文献
[编辑]- ^ 與组织蛋白酶C相關的疾病;在維基數據上查看/編輯參考.
- ^ 2.0 2.1 2.2 GRCh38: Ensembl release 89: ENSG00000109861 - Ensembl, May 2017
- ^ 3.0 3.1 3.2 GRCm38: Ensembl release 89: ENSMUSG00000030560 - Ensembl, May 2017
- ^ Human PubMed Reference:. National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Mouse PubMed Reference:. National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Entrez Gene: CTSC cathepsin C.
- ^ Paris A, Strukelj B, Pungercar J, Renko M, Dolenc I, Turk V. Molecular cloning and sequence analysis of human preprocathepsin C. FEBS Letters. Aug 1995, 369 (2–3): 326–30. PMID 7649281. S2CID 45737414. doi:10.1016/0014-5793(95)00777-7
 .
.
- ^ Hola-Jamriska, Lubomira; Tort, Jose F.; Dalton, John P.; Day, Sharon R.; Fan, Jinjiang; Aaskov, John; Brindley, Paul J. Cathepsin C from Schistosoma japonicum . cDNA encoding the preproenzyme and its phylogenetic relationships. European Journal of Biochemistry. 1998-08, 255 (3). ISSN 0014-2956. doi:10.1046/j.1432-1327.1998.2550527.x (英语).
- ^ Kominami E, Ishido K, Muno D, Sato N. The primary structure and tissue distribution of cathepsin C. Biological Chemistry Hoppe-Seyler. Jul 1992, 373 (7): 367–73. PMID 1515062. doi:10.1515/bchm3.1992.373.2.367.
- ^ Turk, Dušan; Janjić, Vojko; Štern, Igor; Podobnik, Marjetka; Lamba, Doriano; Weis Dahl, Søren; Lauritzen, Connie; Pedersen, John; Turk, Vito; Turk, Boris. Structure of human dipeptidyl peptidase I (cathepsin C): exclusion domain added to an endopeptidase framework creates the machine for activation of granular serine proteases. The EMBO Journal. 2001-12-03, 20 (23) [2022-09-17]. ISSN 0261-4189. PMID 11726493. doi:10.1093/emboj/20.23.6570. (原始内容存档于2022-09-22).
- ^ Wani AA, Devkar N, Patole MS, Shouche YS. Description of two new cathepsin C gene mutations in patients with Papillon-Lefèvre syndrome. Journal of Periodontology. Feb 2006, 77 (2): 233–7. PMID 16460249. doi:10.1902/jop.2006.050124.
- ^ Meade JL, de Wynter EA, Brett P, Sharif SM, Woods CG, Markham AF, Cook GP. A family with Papillon-Lefevre syndrome reveals a requirement for cathepsin C in granzyme B activation and NK cell cytolytic activity. Blood. May 2006, 107 (9): 3665–8. PMID 16410452. doi:10.1182/blood-2005-03-1140
 .
.