ADNA

aDNA (von englisch ancient DNA ‚alte DNA‘) bezeichnet (meist über 100 Jahre) alte DNA. Ein typischer Fall sind Reste von Erbgutmolekülen aus toten Organismen.
Die aDNA-Forschung ist in Zielen und Methoden eng mit der genetischen Rechtsmedizin und der forensischen Anthropologie verwandt und verwendet Methoden der Genanalyse wie die Polymerasekettenreaktion.
Geschichte
[Bearbeiten | Quelltext bearbeiten]Die Geschichte der aDNA ist eng mit der Entwicklung der Polymerase-Kettenreaktion (engl. Polymerase Chain Reaction, PCR), einer speziellen molekularbiologischen Technik verwoben, die es ermöglicht, auch geringe Mengen Erbgut zu vervielfältigen und zu untersuchen. In der Anfangszeit der aDNA kam es zu einer Reihe von Berichten über aDNA, die sich später als falsch herausstellten und Material verwendeten, das aus heutiger Sicht keine Aussicht auf Erhaltung von amplifizierbarer DNA hat. Den Ergebnissen der Untersuchung von aDNA wird daher auch heute noch mit Vorbehalten begegnet.
Die Methodik ist auf die Gewinnung möglichst reiner, vieler und großer Oligonukleotid-Ketten ausgerichtet. Je nach Überlieferungsbedingungen stehen verschiedene Gewebe zur Verfügung, jedoch sind aufgrund der Abgeschlossenheit Hartgewebe (Knochen, Dentin) vorzuziehen, in denen die DNA von zum Beispiel Osteoklasten am besten vor Umwelteinflüssen geschützt erhalten bleibt.
Nach der Gewinnung und Reinigung der DNA-Reste aus dem Gewebe werden aus diesen zuerst die zu untersuchenden Sequenzen in einer PCR bis zur Nachweisgrenze vervielfältigt. Die PCR-Amplifikate können dann durch Fragmentgrößenbestimmung oder Sequenzierung untersucht werden. Vor der Sequenzierung wird, um Kontamination oder bestimmte PCR-Artefakte aufzudecken, häufig das PCR Amplifikat kloniert und selektiert.
Anwendungsgebiete
[Bearbeiten | Quelltext bearbeiten]Der Erkenntnisgewinn anhand von aDNA ist für die genetische Biologie, die Paläozoologie, die Paläobotanik sowie die Anthropologie (besonders Paläoanthropologie) und mit letzterer im Schulterschluss auch für die Archäologie von Bedeutung. Aus letzterer entwickelte sich ein eigener Wissenschaftszweig, die Paläogenetik.
Wurde aDNA zunächst ausschließlich aus fossilen Knochen und Zähnen gewonnen, werden seit den 2010er-Jahren und verstärkt seit 2020 zunehmend auch DNA-Reste aus Sedimenten, die zum Beispiel in Höhlen abgelagert wurden oder im Permafrost liegen, genutzt. Auf diese Weise kann deren Besiedelung rekonstruiert und anhand des Alters der Sedimente datiert werden, obwohl sichtbare Spuren fehlen.[1][2][3] Im Dezember 2022 wurde auf diese Weise ein rund 2 Millionen Jahre altes Ökosystem in Grönland rekonstruiert.[4]
Artbestimmung
[Bearbeiten | Quelltext bearbeiten]Jede biologische Art ist durch ein spezifisches genetisches Merkmalsmuster gekennzeichnet, deswegen ermöglicht die Untersuchung des Erbgutes schon einer einzigen Zelle die eindeutige artspezifische Zuweisung. aDNA nutzt man zur Artbestimmung, wenn die Erhaltungsbedingungen keine anderen eindeutigen Identifikationsmöglichkeiten mehr zulassen oder die Trennschärfe anderer Methoden zu ungenau ist. So lassen sich zum Beispiel Schafe und Ziegen aufgrund hoher Ähnlichkeit im Knochenbau allein anhand der Skelettmerkmale nicht auseinanderhalten.
Phylogenese
[Bearbeiten | Quelltext bearbeiten]Die Evolution der Arten und ihre verwandtschaftlichen Beziehungen untereinander lassen sich statistisch anhand ihrer verschiedenen genetischen Merkmalsmuster darstellen; als Maßzahl dient hier der genetische Abstand. Zur Einordnung bereits ausgestorbener Arten behilft sich die phylogenetische Forschung der aDNA.
Individuelle Verwandtschaftsanalyse
[Bearbeiten | Quelltext bearbeiten]Zwei Individuen sind im biologischen Sinne miteinander verwandt, wenn sie mindestens einen gemeinsamen Vorfahren aufweisen. Der Grad der biologischen Verwandtschaft zweier Individuen lässt sich ebenfalls anhand ihres Erbgutes ablesen. Verfahren ähnlich dem genetischen Vaterschaftstest finden auch in der aDNA-Analyse Anwendung. Einschränkend wirkt sich hier allerdings die sehr bruchstückhafte Erhaltung der DNA-Moleküle aus, die die Untersuchung jeweils nur kleiner Abschnitte ermöglicht. Besonderes Augenmerk gilt hier den Bereichen hoher Variabilität, d. h. Stellen, an denen häufig Mutationen auftreten. Im Speziellen sind das STRs (Short Tandem Repeats) und in zunehmendem Maße auch SNPs (Single Nucleotide Polymorphisms).
Vielversprechend ist hier vor allem die Auswertung mitochondrialer DNA, da diese im Vergleich zur DNA des Zellkerns in wesentlich größerer Kopienzahl vorliegt (etwa 1000 mitochondriale Kopien, aber nur 2 nukleare pro Zelle) und so Erhaltungsprobleme gelindert werden. Allerdings werden Mitochondrien nur von der Mutter, nicht aber vom Vater an die Kinder weitergegeben, d. h., es lässt sich nur die – im biologischen Sinne – matrilineare Abstammungslinie verfolgen.
Geschlechtsdiagnostik
[Bearbeiten | Quelltext bearbeiten]Das genetische Geschlecht eines Individuums ist bei günstigen Erhaltungsbedingungen, d. h. bei der Überlieferung chromosomaler DNA, bestimmbar. Bei Arten, die wie der Mensch nur ein geschlechtsspezifisches Chromosom (Y-Chromosom) besitzen, sind sichere Differenzierungsmöglichkeiten jedoch eingeschränkt. Mit dem Nachweis von aDNA-Bruchstücken, die vom Y-Chromosom stammen (zum Beispiel aus der SRY-Region) ist das genetische Geschlecht eindeutig als männlich belegt, dagegen kann aus dem Fehlen Y-spezifischer Merkmale nicht sicher auf weibliches Geschlecht geschlossen werden, denn unwägbare Erhaltungsbedingungen könnten ebenso der Grund hierfür sein.
Trotz dieser Unsicherheit wird die molekulare Geschlechtsdiagnostik v. a. bei menschlichen Skelettresten angewendet. Insbesondere bei Individuen, an denen aufgrund des jungen Sterbealters oder unspezifischer Skelettmerkmale nur eine unsichere Einordnung mittels biologisch-anthropologischer Methoden möglich ist, hilft die aDNA weiter.
Identifizierung von Individuen und Gegenständen
[Bearbeiten | Quelltext bearbeiten]Als Ausnahmen, die allerdings meist unter großer Anteilnahme der Öffentlichkeit stattfinden, können die Identifizierungen historisch bedeutender Personen mit sterblichen Überresten gelten. Der Wissenschaftler extrahiert dazu zunächst aDNA aus Gewebeproben des fraglichen Individuums (Knochen, Haare, Kleidung), vor allem um daraus die mt-Haplogruppe bzw. bei männlichen Individuen auch den Y-Haplotyp zu bestimmen. Anschließend vergleicht er diesen mit „authentischer“ DNA, die bestenfalls von noch lebenden mutmaßlichen Verwandten stammt. In manchen Fällen hilft auch die Rekonstruktion des genetischen Fingerabdrucks der Person, um diesen mit dem von eng verwandten Personen aus gesicherten Bestattungen zu vergleichen. Eine Extraktion aus persönlichen Gegenständen des fraglichen Individuums ist ebenfalls möglich, allerdings können hier aus Zweifeln an der Authentizität Probleme in der Beweisführung folgen. Bei signifikanter Übereinstimmung zwischen fraglichen und authentischen Proben gilt das Individuum als identifiziert.
Paläopathologie
[Bearbeiten | Quelltext bearbeiten]Die wenigsten Krankheiten lassen sich eindeutig an Skeletten diagnostizieren, deswegen versucht die aDNA-Forschung seit zirka Mitte der 1990er Jahre Erreger von Infektionskrankheiten in menschlichen Überresten nachzuweisen. Im Mittelpunkt stehen dabei zunächst neben der Methodenentwicklung unter anderem der Nachweis eventuell ausgestorbener infektiöser Bakterienstämme und die Gegenüberstellung geschichtlich überlieferter Krankheitsverläufe und -symptome mit den heutigen Erkenntnissen über die jeweilige Krankheit. Mit wachsender Datenzahl kann dieser Zweig in der Zukunft außerdem einen wichtigen Beitrag zur historischen Epidemiologie liefern.
Genetische Geschichte
[Bearbeiten | Quelltext bearbeiten]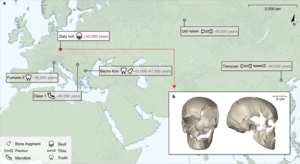
Zum Stand 2021 sind die ältesten vollständig rekonstruierten menschlichen Genome ~45.000 Jahre alt.[6][5] Solche genetischen Daten geben Aufschluss über die Migrations- und genetische Geschichte – unter anderem auch über Kreuzungen zwischen archaischen und modernen Menschen, wie zwischen ersten europäischen modernen Menschen und Neandertalern.[7][8]
Kritik und Probleme
[Bearbeiten | Quelltext bearbeiten]aDNA-Nachweise werden skeptisch aufgenommen, da sie sich häufig als nicht reproduzierbar erwiesen haben. Es besteht grundsätzlich das Risiko, dass die Untersuchungsergebnisse durch Kontaminationen mit fremder, rezenter DNA (auch der der Untersucher) verfälscht sind. Ein Review von 2004 fasste die Bedenken zusammen und berichtet z. B. von einer Untersuchung eines alten Bärenzahnes, aus welchem sich reproduzierbar menschliche DNA nachweisen ließ. Die Arbeit stammte nicht von notorischen Gegnern derartiger Untersuchungen, sondern sollte zu besonderen Vorsichtsmaßnahmen bei aDNA-Analysen auffordern.[9]
Biologische Halbwertszeit
[Bearbeiten | Quelltext bearbeiten]In trockener und kühler Umgebung kann DNA lange Zeit überdauern, allerdings lösen sich die empfindlichen Makromoleküle in kleine Kettenbruchstücke auf. Wärme, Feuchtigkeit, saure und basische pH-Werte begünstigen diese DNA-Schäden und einen Zerfall in immer kleinere Stückchen. aDNA-Fragmente können noch bei einer Nukleotid-Kettenlänge von unter 200 Basenpaaren verwertbar sein – das ist im Vergleich zur theoretischen Gesamtlänge zum Beispiel des menschlichen Genoms von 3×109 Basenpaaren sehr kurz.[10] Für gegebene Umweltparameter lassen sich „Halbwertszeiten“ berechnen, mit denen die zu erwartende Qualität der Ergebnisse abgeschätzt werden kann.[11]
Der Forscher Morten Allentoft von der Murdoch University in Perth (Australien) spricht für ein internationales Forscherteam im Fachmagazin „Proceedings of the Royal Society B“ von 5,5 DNA-Brüchen pro 1 Million Molekülen pro Jahr. Es wurde eine sich ändernde Halbwertszeit der DNA „je nach Temperatur und Umgebungsfaktoren“ ermittelt: „Bei minus 5 Grad (Celsius) beispielsweise betrage die Halbwertszeit für kleine DNA-Stücke im Knochen 158.000 Jahre, bei höheren Temperaturen sei sie kürzer.“ Das Forscherteam äußerte sich wie folgt: „Unsere Halbwertszeit-Berechnungen zeigen beispielsweise, dass es extrem unwahrscheinlich ist, aus 80 bis 85 Millionen Jahre alten Knochen noch intakte DNA-Fragmente isolieren zu können“. Möglich sei aber, dass „urzeitliches Erbgut mehrere Hunderttausend bis sogar eine Million Jahre überdauern könne“.[12] Dieser Ansicht widersprechen andere Wissenschaftler und belegen, dass es durchaus Erhaltungsmöglichkeiten für sehr alte aDNA gebe.[13][14] aDNA-Extraktionen und deren Analysen seien auch an sehr alten Fossilien möglich.[15]
Die Kinetik des DNA-Zerfalls wurde durch „beschleunigtes Altern“ abhängig von der Lagertemperatur und Lagerfeuchtigkeit im Labor gemessen. Die gemessene Aktivierungsenergie des DNA-Zerfalls von 155 kJ/mol zeigt die Grenzen der Langzeitstabilität von DNA bei tiefen Temperaturen auf.[16]
Postmortale Mutationen
[Bearbeiten | Quelltext bearbeiten]Es wurde auch festgestellt, dass bei sehr alter aDNA, etwa aus dem Miozän, gehäuft mit postmortalen Mutationen zu rechnen sei, da die ursprüngliche Base Cytosin dann als Uracil vorliegen könne, was die Interpretation erschwere.[17]
Ein weiteres, bisher kaum erforschtes Problem liegt in den so genannten „Hot Spots“, denn diese DNA-Stellen können nach dem Ableben des Organismus durch chemische Reaktionen derart verändert werden. So entstehen Pseudo-Mutationen, die es zu erkennen gilt.
Kontamination
[Bearbeiten | Quelltext bearbeiten]Da aDNA meist in sehr geringen Mengen überliefert ist, bedient sich die Forschung der PCR, um die erhaltenen Stücke zunächst zu vervielfältigen. Aufgrund der ausgesprochen hohen Sensibilität der PCR sind Fehlamplifikationen sehr häufig, d. h., es wird anstatt der originären alten Ziel-DNA das Erbgut anderer Organismen (häufig von Boden-Bakterien) oder moderne menschliche DNA vervielfältigt, die durch ungenügende Aufbereitung des Materials oder unsauberes Arbeiten in die Probe gelangt ist.
Andere Probleme
[Bearbeiten | Quelltext bearbeiten]Von geringerer Bedeutung sind sogenannte Inhibitoren, die aus dem Liegenmilieu der DNA, zum Beispiel dem Boden, stammen und die PCR durch Blockieren des Enzyms verhindern können. Häufig wird Bovines Serumalbumin, ein aus Rinderblut gewonnenes Eiweiß, zur Bindung von Eisen, dem häufigsten Inhibitor, dem Reaktionsgemisch zugefügt. Liegen dennoch Hinweise vor, dass PCR-Inhibition die Ursache für falsch negative Ergebnisse ist, hilft es, die aDNA-Probe verdünnt einzusetzen. Leider verringert sich durch die Verdünnung auch die DNA-Konzentration in der Probe, was die Chancen auf PCR-Erfolg wieder verringert.
Schließlich sagt die sichtbare Erhaltung eines Organismus wenig über den Zustand der enthaltenen aDNA aus. So ist zum Beispiel aus Moorleichen aufgrund des sauren Liegemilieus selten verwertbare DNA zu extrahieren. Auch Trockenmumien mit hervorragender Weichteilerhaltung enthalten oftmals nur noch sehr geringe aDNA-Spuren.
Beispiele (Auswahl)
[Bearbeiten | Quelltext bearbeiten]Die Auswahl gibt einen Überblick über die Spannweite der aDNA-Forschung. Da auch negative Ergebnisse von wissenschaftlicher Bedeutung sind, werden „berühmte und wichtige Misserfolge“ im Folgenden ebenfalls angeführt.
Bernstein-Inklusen
[Bearbeiten | Quelltext bearbeiten]Film und Roman des Titels „Jurassic Park“ haben Anfang der 1990er-Jahre stark zur öffentlichen und sogar wissenschaftlichen Euphorie in Sachen aDNA beigetragen. In der Geschichte wird aus Fossilien in Bernstein (aus so genannten Inklusen) altes Erbgut gewonnen, das zur Neuzüchtung bereits ausgestorbener Arten verwendet wird.
Tatsächlich wurde wiederholt publiziert, dass aus Bernstein nicht nur sequenzierbare aDNA isoliert werden kann,[18][19][20] auch aus Chloroplasten-DNA,[21][22] sondern auch Proteine[23] und sogar lebensfähige Organismen.[24][25] Diese Nachweise wurden kontrovers diskutiert.[26][27][28][29][30]
Viren
[Bearbeiten | Quelltext bearbeiten]- Influenza-A-Virus H1N1. – Aus erhaltenen Gewebeproben von Opfern der Spanischen Grippe, die im Winter 1918/19 verstorben waren, wurde die RNA des pandemischen Influenza-A-Virus H1N1 gewonnen.[31]
- „ancient caribou feces associated virus“ (aCFV). – Aus dem vor rund 700 Jahren am Rande der Arktis abgesetzten Kot eines Karibus wurde ein DNA-Virus isoliert und in den Zellen einer Tabakpflanze (Nicotiana benthamiana) reaktiviert.[32]
Prokaryoten
[Bearbeiten | Quelltext bearbeiten]- Mycobacterium leprae. – Lepra (auch „Aussatz“ genannt) war jahrhundertelang eine gefürchtete Infektionskrankheit. Deren Erreger, Mycobacterium leprae, ist heute noch so virulent wie im Hochmittelalter; dass Lepra inzwischen eine selten gewordene Erkrankung ist, kann daher auf die verbesserte Hygiene zurückgeführt werden.[33]
- Mycobacterium tuberculosis. – Eine offene Frage in der Paläopathologie, der Streit um die Herkunft des Syphilis-Erregers (Treponema pallidum), wurde unter anderem mittels aDNA-Analysen zu beantworten versucht. Die gezielte Suche nach allen bekannten Erregern der Gattung Treponema in einer 46 menschliche Skelette umfassenden Studie blieb jedoch erfolglos. Die Entdeckung von alten Tuberkulose-Bakterien in einigen dieser Skelette[34] bestätigte dagegen im Jahr 2005 grundsätzlich die Möglichkeit, Erreger bestimmter Krankheiten mittels aDNA nachzuweisen.[35]
- 2014 wurden aus drei jeweils tausend Jahre alten, präkolumbischen Skeletten das Genom von Mycobacterium tuberculosis isoliert und auf diese Weise belegt, dass der Erreger der Tuberkulose bereits lange vor dem Eindringen der Europäer in Südamerika verbreitet war.[36] Schon 2001 war DNA von Mycobacterium tuberculosis aus dem Skelett eines 17.000 Jahre alten, nordamerikanischen Bisons gewonnen worden.[37]
- Pestbakterium (Yersinia pestis). – 2011 wurde das Genom des Yersinia pestis-Stammes beschrieben, der von 1348 bis 1350 während der Zeit des „Schwarzen Todes“ Menschen in England infiziert hatte; so konnte erstmals belegt werden, dass die mittelalterliche Pest vom gleichen Erreger verursacht wurde wie die Pest-Erkrankungen in der Gegenwart.[38]
Pflanzen und Pilze
[Bearbeiten | Quelltext bearbeiten]- Baumwolle (Gossypium). – Vier Funde aus archäologischen Grabungen in Brasilien, Peru und Ägypten – die ältesten 3820 bis 3630 Jahre alt (cal BP) – trugen dazu bei, Veränderungen im Genom der Baumwolle im Verlauf der vergangenen rund 4000 Jahre nachzuvollziehen.[39]
- Einkorn (Triticum monococcum). – Aus rund 3300 Jahre alten Samen von Einkorn, die in Griechenland erhalten geblieben waren, konnten Teile des Genoms identifiziert werden.[40]
- Kartoffelmehltau (Phytophthora infestans). – Fünf DNA-Proben aus Herbarien trugen dazu bei, Veränderungen im Genom des Erregers der Kraut- und Knollenfäule in der Zeit seit 1845 nachzuvollziehen; Phytophthora infestans war Verursacher der Großen Hungersnot in Irland zwischen 1845 und 1852.[41]
- Mais (Zea mays). – Der Vergleich von bis zu 4700 Jahre altem Mais mit rezenten Mais-Varianten erbrachte Hinweise darauf, dass der heute angebaute Mais von unterschiedlichen Wild-Populationen abstammt.[42]
Tiere
[Bearbeiten | Quelltext bearbeiten]aDNA wurde in einer Vielzahl von Arbeiten unter anderem zur Klärung von Verwandtschaftsverhältnissen bei Tieren verwendet.
Vögel
[Bearbeiten | Quelltext bearbeiten]
- Moa-Nalos. – Anhand der Analyse von fossiler mtDNA wurde belegt, dass diese ausgestorbenen, einstmals auf Hawaii lebenden, großen, flugunfähigen Vögel zwar am nächsten verwandt waren mit einer Unterfamilie der Entenvögel, den Anatinae, jedoch keine engere Verwandtschaft zu einer der heute noch lebenden Entenarten besaßen.[43]
- Haastadler (Harpagornis moorei). – Mit einem Gewicht von 10 bis 15 Kilogramm und einer Spannweite der Flügel von 2 bis 3 Metern war er einer der Spitzenprädatoren Neuseelands. Gleichwohl konnte anhand erhalten gebliebener Gewebeproben dieser ausgestorbenen Art nachgewiesen werden, dass er unter den rezenten Vogelarten am ehesten verwandt war mit dem nur rund ein Zehntel so großen, in Australien lebenden Hieraaetus morphnoides („Little Eagle“).[44]
- Dodo (Raphus cucullatus). – Anhand der Analyse von fossiler rDNA wurde belegt, dass diese ausgestorbenen, einstmals auf Mauritius lebenden, großen, flugunfähigen Vögel am nächsten verwandt waren mit dem ebenfalls ausgerotteten Rodrigues-Solitär von der Insel Rodrigues. Unter den heute noch lebenden Vögeln stehen ihnen die Kragentauben am nächsten.[45]
Säugetiere
[Bearbeiten | Quelltext bearbeiten]- Beuteltiere
- Beutelwolf (auch: Tasmanischer Tiger, Thylacinus cynocephalus). – Aus 12 in Museen erhalten gebliebenen Exemplaren des seit 1936 ausgestorbenen Beutelwolfs wurde DNA gewonnen. Deren Vergleich ergab, dass die genetische Variabilität der 102 bis 159 Jahre alten DNA extrem gering war. Ursache hierfür ist vermutlich, dass Tasmanien seit rund 10.000 Jahren – nach dem Höhepunkt der letzten Eiszeit – von Australien abgeschnitten war.[46]
- Zahnarme
- Faultiere (Folivora). – Um die verwandtschaftlichen Beziehungen zu analysieren, wurde bereits im Jahr 2001 ribosomale DNA (rDNA) aus erhaltenen Zellen von einzelnen ausgestorbenen Riesenfaultieren (unter anderem Mylodon und Nothrotheriops) mit rDNA von Zweifinger-Faultieren und Dreifinger-Faultieren verglichen.[47] Im Jahr 2019 konnte durch weitere genetische Analysen in Verbindung mit Proteinuntersuchungen an ausgestorbenen, zusätzlich einbezogen waren Megatherium und Megalonyx, und rezenten Vertretern die gesamte systematische Gliederung der Gruppe neu überarbeitet werden.[48][49]
- Gepanzerte Nebengelenktiere
- Gürteltiere (Dasypoda) und Glyptodontidae. – Im Jahr 2015 ließ sich über genetische Analysen an den Gürteltieren und an Doedicurus aus der Gruppe der Glyptodonten feststellen, dass letztere lediglich einen Zweig innerhalb der ersteren darstellen.[50]
- Rüsseltiere
- Elefanten (Elephantidae). – Die Mammute (Mammuthus) standen schon sehr früh im Fokus paläogenetischer Forschung. Das Verwandtschaftsverhältnis des Wollhaarmammuts (Mammuthuis primigenius) zu den heutigen Elefanten konnte dadurch geklärt werden. Nach einer anfänglichen festgestellten näheren Verwandtschaft zum Afrikanischen Elefanten (Loxodonta africana),[51] ermittelt an einem rund 28.000 Jahren Mammutfund, zeigten spätere Analysen eine nähere Beziehung zum Asiatischen Elefanten (Elephas maximus). Die beiden Linien trennten sich vor rund 6,7 Millionen Jahren, der Afrikanische Elefant hatte sich schon vor etwa 7,6 Millionen Jahren abgespalten.[52][53] Des Weiteren weist der DNA-Code für das Hämoglobin eines 43.000 Jahre alten Wollhaarmammuts drei vom Hämoglobin eines Asiatischen Elefanten abweichende Sequenzen auf. Diese wurden 2010 in die DNA-Sequenz für das Hämoglobin eines Asiatischen Elefanten eingebaut, um Erkenntnisse über die Kälteanpassung der Mammuts zu gewinnen.[54] Außerdem zeigen genetische Studien an den letzten Vertretern des Wollhaarmammuts auf der Wrangelinsel zahlreiche Mutationen auf, die unter anderem zur Verminderung der Geruchswahrnehmung führten.[55][56]
- Für das Steppenmammut (Mammuthus trogontherii) liegt der derzeit (Stand Februar 2021) älteste Beleg für DNA vor, die rund 1,2 Millionen Jahre alt ist. Sie wurde aus dem Backenzahn eines in Sibirien entdeckten Steppenmammuts gewonnen. Mit ihrer Hilfe klärte sich einerseits die enge Verwandtschaft zum Wollhaarmammut, andererseits auch zum nordamerikanischen Präriemammut (Mammuthus columbi). Letzteres entstand durch einen deutlichen Genfluss seitens der Wollhaarmammut-Steppenmammut-Linie.[57] Eine deutliche Hybridisierung zwischen dem Wollhaarmammut und dem Präriemammut ist auch für die letzte Kaltzeit im Raum der Großen Seen feststellbar.[58] Zuvor sehr alte DNA entstammt dem Kreta-Zwergmammut (Mammuthus creticus), deren Alter rund 800.000 Jahre betrug.[59] Für den Europäischen Waldelefanten (Palaeoloxodon antiquus) aus der Gattung Palaeoloxodon konnte genetisch eine engere Beziehung zum heutigen Waldelefanten (Loxodonta cyclotis) herausgearbeitet werden.[60][61]
- Mammutidae. – Die urtümliche Rüsseltiergruppe der Mammutidae, nicht verwandt mit den Mammuten, spaltete sich genetischen Analysen zufolge bereits vor rund 26 Millionen Jahren von der Linie ab, die zu den Elefanten führte. Gewonnen wurden die Daten am Amerikanischen Mastodon (Mammut americanum), das bis in das späte Pleistozän in Nordamerika auftrat.[52][53]
- Tenrekartige
- Tenreks (Tenrecidae). – Das möglicherweise erst vor rund 500 Jahren ausgestorbene Plesiorycteropus auf Madagaskar, das lange Zeit als ein Verwandter des Erdferkels (Orycteropus) galt, erwies sich im Jahr 2013 durch genetische Untersuchungen als den Tenreks näherstehend.[62]
- Raubtiere
- Bären (Ursidae). – Die DNA aus den Zellen eines fossilen Höhlenbären (Ursus spelaeus) aus der Vindija-Höhle gehörte 1999 zu den ersten in Teilen rekonstruierten DNA-Fragmenten von Lebewesen, die schon vor tausenden Jahren ausgestorben waren.[63] Eine rund 360.000 Jahre alte aDNA-Probe aus einem Knochen eines Höhlebären der Kudaro-Höhle im Kaukasus wurde für einen Bericht im Jahr 2021 aufbereitet und gehört zu den ältesten derartigen Nachweisen für ein Lebewesen außerhalb des heutigen Permafrostgebietes.[64] Im Jahr 2005 wurde die Nähe der Verwandtschaft von Ursus spelaeus mit anderen Bärenarten analysiert mit dem Ergebnis, dass die engsten Beziehungen zum Braunbären (Ursus arctos) und zum Eisbären (Ursus maritimus) bestehen.[65] Darüber hinaus trug die Analyse der DNA eines rund 120.000 Jahre alten Exemplars des Eisbären dazu bei, eine wiederholte genetische Vermischung mit dem Braunbären aufzuspüren.[66]
- Hyänen (Hyaenidae). – Die enge Verwandtschaft der Höhlenhyäne (Crocuta spelaea) zur Tüpfelhyäne (Crocuta crocuta) bestätigte sich bereits im Jahr 2005 durch aDNA.[67] Sukzessive weitere genetische Analysen deckten ein komplexes Beziehungsgeflecht auf.[68]
- Katzen (Felidae). – Paläogenetische Untersuchungen bewiesen nicht nur das enge Verhältnis des Höhlenlöwen (Panthera spelaea) zum heutigen Löwen (Panthera leo) sowie zum Amerikanischen Löwen (Panthera atrox), sondern auch deren komplexe Verwandtschaftsverhältnisse. Auch wurde ermittelt, dass der Höhlenlöwe bis in das nördliche Nordamerika vorkam, ursprünglich galten diese Bereich als vom Amerikanischen Löwen besiedelt.[69][70][71] Die Gruppe der Säbelzahnkatzen erwies sich durch DNA-Untersuchungen an Homotherium und Smilodon als Schwestergruppe aller anderen Katzen. Ihre Trennung vollzog sich bereits vor rund 20 Millionen Jahren.[72] Weitere Studien an Homotherium deckten eine enge genetische Verzahnung zwischen nordamerikanischen und europäischen Vertretern der Gattung auf.[73]
- Hunde (Canidae). – Der ursprünglich als Verwandter des Wolfes (Canis lupus) angesehene dire wolf wurde aufgrund paläogenetischer Befunde im Jahr 2021 in die Gattung Aenocyon verschoben.[74]
- Paarhufer
- Hirsche (Cervidae). – Der Riesenhirsch (Megaloceros giganteus) starb nach dem Höhepunkt der letzten Kaltzeit aus, seine verwandtschaftliche Nähe zu anderen Arten der Echten Hirsche wurde 2006 anhand von erhalten gebliebener mtDNA analysiert, wobei die engste Bindung zum Damhirsch (Dama dama) besteht.[75][76] Für den Korsischen Rothirsch, auch Tyrrhenischer Rothirsch (Cervus elaphus corsicanu) wurde anhand von mitochondrialer aDNA aus rund 6.300 bis 15.600 (cal BP) alten Knochenfunden nachgewiesen, dass die in Sardinien beheimatete Population von einer Population aus Italien abstammt.[77]
- Hornträger (Bovidae). – Der Steppenbison (Bos priscus, auch: Bison priscus) war während der letzten Kaltzeit weit verbreitet in Europa und starb vor rund 9000 Jahren aus. Anhand von aDNA konnte nachvollzogen werden, dass – ohne intensive Einwirkung des Menschen – bereits vor 37.000 Jahren die genetische Vielfalt dieser Art dramatisch abnahm.[78] Eine Klärung seiner Verwandtschaftsverhältnisse zu anderen Rindern erfolgte im Jahr 2017. Demnach steht der Steppenbison in der Vorfahrenlinie zum Amerikanischen Bison (Bos bison), während eine andere ausgestorbene Form, Bos schoetensacki, einen Vorläufer des Wisents (Bos bonasus) bildet.[79] Die Domestizierung des Hausrindes (Bos taurus) aus dem Auerochsen (Bos primigenius) konnte mit Hilfe von aDNA nachvollzogen werden.[80]
- Anhand der DNA aus 6000 Jahre alten Knochen der Höhlenziege (Myotragus balearicus) konnte ermittelt werden, dass die einstmals auf Mallorca und Menorca heimische Art näher mit dem Hausschaf (Ovis gmelini aries) als mit der Hausziege (Capra aegagrus hircus) verwandt ist.[81] Aus Pergament des 17. und 18. Jahrhunderts gewonnene aDNA wies nach, dass es aus der Haut von Hausschafen hergestellt worden war.[82]
- Unpaarhufer
- Pferde (Equidae). – Das Quagga (Equus quagga quagga) gehörte zu den ersten Tieren, deren aDNA untersucht wurde. Aus Zellen aus Museumspräparaten gewannen Wissenschaftler im Jahr 1984 die ersten Nukleotidsequenzen.[83] Im Ergebnis der Untersuchungen steht das Quagga heute als Unterart des Steppenzebras fest.[84][85] Die bisher älteste untersuchte DNA eines Pferdes der Gattung Equus wurde auf ein Alter von 780.000 bis 560.000 Jahre datiert und stammt aus dem Permafrostgebiet von Kanada.[86] Weitere paläogenetische Daten sind von spätpleistozänen und frühholozänen Pferden entnommen worden, so von Equus lenensis und von Equus ovodovi, beide im nördlichen Asien verbreitet, sowie von Equus hydruntinus, das in weiten Bereichen Eurasiens auftrat.[87] Eine umfassende Studie an Hauspferden aus verschiedenen kulturgeschichtlichen Epochen und am Przewalski-Pferd (Equus przewalskii) kam im Jahr 2018 zu der Erkenntnis, dass letzteres nicht wie angenommen das einzige verbliebene Wildpferd repräsentiert, sondern wahrscheinlich ein Nachfahre wieder verwilderter Pferde ist, die vor rund 5500 Jahren in Zentralasien domestiziert worden waren.[88]
- Die stilt-legged horses, eine Gruppe schmalfüßiger Pferde aus dem Pleistozän Nordamerikas, waren in ihrer phylogenetischen Stellung lange Zeit umstritten. Eine paläogenetische Studie aus dem Jahr 2017 führte zur Aufstellung der eigenen Gattung Haringtonhippus.[89]
- Nashörner (Rhinocerotidae). – Die anatomisch vermutete Verwandtschaft des Wollnashorns (Coelodonta antiquitatis) aus der Gattung Coelodonta mit dem Sumatra-Nashorn (Dicerorhinus sumatrensis) bestätigte sich durch aDNA im Jahr 2003 nach der Untersuchung eines Fossils aus der Scladina-Höhle in Belgien,[90] das Ergebnis konnte nachfolgend mehrfach reproduziert werden.[91][92] Die Trennung der beiden Linien liegt fast 26 Millionen Jahre zurück. Im Jahr 2020 zeichnete eine Genanalyse das Verschwinden des Wollnashorns im ausgehenden Pleistozän nach.[93] In den engeren Verwandtschaftskreis aus Coelodonta-Dicerorhinus lässt sich nach genetischen Untersuchungen eines Schädels aus Sibirien im Jahr 2017 auch das Waldnashorn (Stephanorhinus kirchbergensis) einordnen, das innerhalb der Gattung Stephanorhinus steht.[94] Auch das war vorher aufgrund anatomischer Übereinstimmungen angenommen worden und fand im gleichen Jahr eine Absicherung durch Proteinsequenzierungen.[95] Noch vor dem Sumatra-Nashorn hatte sich genetischen Daten zufolge vor gut 31 Millionen Jahren die Linie von Elasmotherium abgespaltet, ein riesiges einhörniges Nashorn, das noch in der letzten Kaltzeit in Osteuropa sowie in Zentral- und Nordasien vorkam.[96]
- Südamerikanische Huftiere (Meridiungulata). – Auch wenn nicht direkt zu den Unpaarhufern gehörend, untermauerten im Jahr 2017 Genuntersuchungen an Macrauchenia die nähere Verwandtschaft zumindest der Litopterna als große Gruppe der ausgestorbenen Südamerikanischen Huftiere mit den Unpaarhufern.[97] Zuvor war dies bereits mit Proteinuntersuchungen festgestellt worden, wobei hierbei zusätzlich auch für Toxodon aus der Gruppe der Notoungulata ein vergleichbares Ergebnis vorgelegt werden konnte. Eine teils angenommene Verwandtschaft mit den Afrotheria ließ sich dadurch nicht belegen.[98][99]
- Nagetiere
- Biber (Castoridae). – Nach genetischen Befunden trennten sich die im ausgehenden Pleistozän ausgestorbenen Riesenbiber (Castoroides) von den heutigen Bibern (Castor) vor knapp 20 Millionen Jahren ab. Beide Gattungen gehören zu den semi-aquatisch lebenden Vertretern der Familie, so dass diese Verhaltensweise wahrscheinlich wenigstens seit dem Unteren Miozän besteht.[100]
Menschen
[Bearbeiten | Quelltext bearbeiten]Neandertaler
[Bearbeiten | Quelltext bearbeiten]- Teile der Neandertaler-DNA wurden 1997 erstmals in der Arbeitsgruppe von Svante Pääbo sequenziert;[101] 2008 wurde die vollständige mitochondriale DNA[102] und 2010 wurden mehr als drei Milliarden Basenpaare (= 60 Prozent) der DNA analysiert.[103]
Denisova-Mensch
[Bearbeiten | Quelltext bearbeiten]- Wie zuvor die Neandertaler-DNA wurde in der Arbeitsgruppe von Svante Pääbo auch die DNA der Denisova-Menschen weitgehend rekonstruiert.[104]
Anatomisch moderner Mensch
[Bearbeiten | Quelltext bearbeiten]- Das bislang älteste größtenteils sequenzierte menschliche Genom stammt aus dem Oberschenkelknochen eines Mannes aus einer ausgestorbenen, westsibirischen Population und ist etwa 43.000 bis 47.000 Jahre alt.[105]
- Ägyptische Mumien
- Der Evolutionsgenetiker Svante Pääbo publizierte 1985 als erster die Entdeckung alter DNA in Proben einer ägyptischen Mumie.[106] Aufgrund damals noch fehlender Kontrollmöglichkeiten musste er später jedoch hinzufügen, dass seine aDNA-Proben möglicherweise durch moderne DNA kontaminiert gewesen sein könnten.[107][108]
- Aufgrund hervorragender Weichteilerhaltung sind die ägyptischen Mumien inzwischen häufiger Gegenstand von aDNA-Untersuchung, denn man erhofft sich auch auf molekularer Ebene gute Überlieferungsbedingungen.[109]
- russische Zarenfamilie (siehe Nikolaus II.)
- Richard III. (England)
- Kaspar Hauser
- Martin Bormann
- Der Tod Bormanns (der sich später bei Untersuchungen des Skeletts als Freitod herausstellte) im Frühjahr 1945 ist mehrfach angezweifelt worden. 1972 wurden am Lehrter Bahnhof in Berlin zwei Skelette entdeckt, von denen eines laut gerichtsmedizinischem Gutachten aus der forensischen Odontologie (Gebissmerkmale) als Bormann identifiziert werden konnte. Erneute Zweifel führten schließlich zu einer DNA-Analyse Ende der 1990er – obwohl es sich der Definition nach noch nicht um eine aDNA-Untersuchung handelte (<75 Jahre), konnten nur noch verhältnismäßig geringe Reste DNA nachgewiesen werden. Die Übereinstimmung mitochondrialer Muster zwischen dem beprobten Skelett und einer noch lebenden Cousine Bormanns macht eine verwandtschaftliche Beziehung in mütterlicher Linie und damit die Identifizierung Bormanns wahrscheinlich.[110]
- Der Schwedenkönig fiel 1632 während der Schlacht bei Lützen. Seine Leiche wurde einbalsamiert nach Stockholm überführt und dort beigesetzt. Teile seiner Kleidung verblieben jedoch in Lützen und werden dort heute im Museum ausgestellt. Die Untersuchung von Blutresten im Stoff erbrachte ausreichende Mengen alter DNA, um diese mit dem Erbgut seiner Nachfahren im heutigen schwedischen Herrscherhaus vergleichen zu können. Die Echtheit wurde bestätigt.[111]
- Thomas Jefferson
- Friedrich Schiller
- Verwandtschaftsanalyse der spätbronzezeitlichen Skelette aus der Lichtensteinhöhle im Harz
- Verwandtschaftsanalyse der schnurkeramischen Skelette aus Eulau in Sachsen-Anhalt[112]
Literatur
[Bearbeiten | Quelltext bearbeiten]Zur Einführung
[Bearbeiten | Quelltext bearbeiten]- Michael Hofreiter: Spurensuche in alter DNA. In: Biologie in unserer Zeit. Band 39, Nr. 3, 2009, S. 176–184, doi:10.1002/biuz.200910392.
- Elizabeth D. Jones: Ancient DNA: The Making of a Celebrity Science. Yale University Press, 2022, ISBN 978-0-30024012-2.
- Martin Jones: The Molecule Hunt. How Archaeologists are Bringing the Past Back to Life. Penguin Books 2002, ISBN 1-55970-679-1.
- Jacob S. Sherkow, Henry T. Greenly: What if Extinction is not Forever? In: Science. Band 340, Nr. 6128 2013, S. 32–33, doi:10.1126/science.1236965.
Fachartikel
[Bearbeiten | Quelltext bearbeiten]- Jermey J. Austin et al.: Problems of reproducibility – does geologically ancient DNA survive in amber-preserved insects? In: Proceedings of the Royal Society London. Band 264, Nr. 1381, 1997. S. 467–474, doi:10.1098/rspb.1997.0067 (Volltext (PDF; 429 kB) )
- Jacob S. Sherko, Henry T. Greely: What If Extinction Is Not Forever? In: Science. Band 340, Nr. 6128, 2013, S. 32–33, doi:10.1126/science.1236965, Volltext (PDF; 289 kB)
- Beth Shapiro und Michael Hofreiter: A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA. In: Science. Band 343, Nr. 6165, 2014, doi:10.1126/science.1236573.
- Montgomery Slatkin und Fernando Racimo: Ancient DNA and human history. In: PNAS. Band 113, Nr. 23, 2016, S. 6380–6387, doi:10.1073/pnas.1524306113 (freier Volltext)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Eske Willerslev, John Davison, Mari Moora, Martin Zobel, Eric Coissac, Mary E. Edwards, Eline D. Lorenzen, Mette Vestergård, Galina Gussarova, James Haile, Joseph Craine, Ludovic Gielly, Sanne Boessenkool, Laura S. Epp, Peter B. Pearman, Rachid Cheddadi, David Murray, Kari Anne Bråthen, Nigel Yoccoz, Heather Binney, Corinne Cruaud, Patrick Wincker, Tomasz Goslar, Inger Greve Alsos, Eva Bellemain6, Anne Krag Brysting, Reidar Elven, Jørn Henrik Sønstebø, Julian Murton, Andrei Sher, Morten Rasmussen, Regin Rønn, Tobias Mourier, Alan Cooper, Jeremy Austin, Per Möller, Duane Froese, Grant Zazula, Francois Pompanon, Delphine Rioux, Vincent Niderkorn, Alexei Tikhonov, Grigoriy Savvinov, Richard G. Roberts, Ross D. E. MacPhee, M. Thomas P. Gilbert, Kurt H. Kjær, Ludovic Orlando, Christian Brochmann und Pierre Taberle: Fifty thousand years of Arctic vegetation and megafaunal diet. In: Nature. Band 506, 2014, S. 47–51, doi:10.1038/nature12921.
- ↑ Elena I. Zavala et al.: Pleistocene sediment DNA reveals hominin and faunal turnovers at Denisova Cave. In: Nature. Band 595, 2021, S. 399–403, doi:10.1038/s41586-021-03675-0.
Pleistocene sediment DNA from Denisova Cave. Auf: eurekalert.org vom 23. Juni 2021. - ↑ Pere Gelabert et al.: Genome-scale sequencing and analysis of human, wolf, and bison DNA from 25,000-year-old sediment. In: Current Biology. Online-Veröffentlichung vom 12. Juli 2021, doi:10.1016/j.cub.2021.06.023.
DNA from dirt can offer new view of ancient life. Auf: sciencemag.org vom 12. Juli 2021. - ↑ Kurt H. Kjær et al.: A 2-million-year-old ecosystem in Greenland uncovered by environmental DNA. In: Nature. Band 612, 2022, S. 283–291, doi:10.1038/s41586-022-05453-y.
Älteste DNA enthüllt „verlorene Welt“. Auf: scinexx.de vom 8. Dezember 2022. - ↑ a b Kay Prüfer, Cosimo Posth, He Yu, Alexander Stoessel, Maria A. Spyrou, Thibaut Deviese, Marco Mattonai, Erika Ribechini, Thomas Higham, Petr Velemínský, Jaroslav Brůžek, Johannes Krause: A genome sequence from a modern human skull over 45,000 years old from Zlatý kůň in Czechia. In: Nature Ecology & Evolution. 7. April 2021, S. 1–6, doi:10.1038/s41559-021-01443-x.
- ↑ Neanderthal ancestry identifies oldest modern human genome In: phys.org. Abgerufen am 10. Mai 2021 (englisch).
- ↑ Europe’s oldest known humans mated with Neandertals surprisingly often In: Science News, 7. April 2021. Abgerufen am 10. Mai 2021
- ↑ Mateja Hajdinjak et al.: Initial Upper Palaeolithic humans in Europe had recent Neanderthal ancestry. In: Nature. 592. Jahrgang, Nr. 7853, April 2021, S. 253–257, doi:10.1038/s41586-021-03335-3.
- ↑ Svante Pääbo, Hendrik Poinar, David Serre, Viviane Jaenicke-Després, Juliane Hebler, Nadin Rohland, Melanie Kuch, Johannes Krause, Linda Vigilant, Michael Hofreiter: Genetic analyses from ancient DNA. In: Annual Review of Genetics. Band 38, Dezember 2004, S. 645–679, doi:10.1146/annurev.genet.37.110801.143214.
- ↑ Susanne Hummel: Ancient DNA Typing. Methods, Strategies and Applications. Springer, 2003, ISBN 978-3-642-07705-0.
- ↑ Zum Beispiel: Isolina Marota et al.: DNA decay rate in papyri and human remains from Egyptian archaeological sites. In: American Journal of Physical Anthropology. Band 117, Nr. 4, 2002, S. 310–318, doi:10.1002/ajpa.10045.
- ↑ Forscher ermitteln die Halbwertszeit der DNA. In: Frankfurter Rundschau vom 9. Oktober 2012.
- ↑ S. P. Tiwari, D. K. Chauhan: Ancient DNA: the molecular evidence of the evolutionary past. In: Bioherald: International Journal of Biodiversity & Environment Band 2, Nr. 1, 2012, S. 19–24.
- ↑ Erika Hagelberg, Michael Hofreiter, Christine Keyser: Ancient DNA: the first three decades. In: Philosophical Transactions of the Royal Society B: Biological Sciences. Band 370, Nr. 1660, 2015, 20130371, doi:10.1098/rstb.2013.0371.
- ↑ Bernd Herrmann, Susanne Hummel (Hrsg.): Ancient DNA: Recovery and Analysis of Genetic Material from Paleontological, Archaeological, Museum, Medical, and Forensic Specimens. Springer Science & Business Media, Dezember 2012.
- ↑ Robert N. Grass et al.: Robust Chemical Preservation of Digital Information on DNA in Silica with Error-Correcting Codes In: Angewandte Chemie International Edition. Band 54, Nr. 8, 2015, S. 2552–2555, doi:10.1002/anie.201411378.
- ↑ Jesse Dabney, Matthias Meyer, Svante Pääbo: Ancient DNA damage. In: Cold Spring Harbor perspectives in biology. Band 5, Nr. 7, 2013, a012567.
- ↑ K. O. Walden, Hugh M. Robertson: Ancient DNA from amber fossil bees? In: Molecular Biology and Evolution. Band 14, Nr. 10, 1997, S. 1075–1077.
- ↑ Rob DeSalle, J. Gatesy, W. Wheeler, D. Grimaldi: DNA sequences from a fossil termite in Oligo-Miocene amber and their phylogenetic implications. In: Science. Band 257, Nr. 5078, 1992, S. 1933–1936, doi:10.1126/science.1411508.
- ↑ Raúl J. Cano, Heridrik N. Poinar, Norman J. Pieniazek, Aftim Acra, George O. Poinar Jr.: Amplification and sequencing of DNA from a 120–135-million-year-old weevil. In: Nature. Band 363, Nr. 6429, 1993, S. 536–538.
- ↑ Josep A. Rosselló: The never-ending story of geologically ancient DNA: was the model plant Arabidopsis the source of Miocene Dominican amber? In: Biological Journal of the Linnean Society. Band 111, Nr. 1, 2014, S. 234–240, doi:10.1111/bij.12192.
- ↑ Kathrin Feldberg, Jochen Heinrichs, Alexander R. Schmidt, Jiří Váňa, Harald Schneider: Exploring the impact of fossil constraints on the divergence time estimates of derived liverworts. In: Plant Systematics and Evolution. Band 299, Nr. 3, 2013, S. 585–601, doi:10.1007/s00606-012-0745-y.
- ↑ Jeffrey L. Bada, Xueyun S. Wang, Hendrik N. Poinar, Svante Pääbo, George O. Poinar: Amino acid racemization in amber-entombed insects: implications for DNA preservation. In: Geochimica et Cosmochimica Acta. Band 58, Nr. 14, 1994, S. 3131–3135, doi:10.1016/0016-7037(94)90185-6.
- ↑ C. L. Greenblatt, A. Davis, B. G. Clement, C. L. Kitts, T. Cox, R. J. Cano: Diversity of microorganisms isolated from amber. In: Microbial Ecology. Band 38, Nr. 1, 1999, S. 58–68, doi:10.1007/s002489900153.
- ↑ R. J. Cano, M. K. Borucki: Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber. In: Science. Band 268, Nr. 5213, S. 1060–1064, doi:10.1126/science.7538699.
- ↑ David A. Grimaldi: Amber: window to the past. Harry N. Abrams, Publishers, 1996.
- ↑ Jeremy J. Austin, Andrew J. Ross, Andrew B. Smith, Richard A. Fortey, Richard H. Thomas: Problems of reproducibility – does geologically ancient DNA survive in amber–preserved insects?. In: Proceedings of the Royal Society of London B: Biological Sciences. Band 264, Nr. 1381, 1997, S. 467–474, doi:10.1098/rspb.1997.0067.
- ↑ Gabriel Gutiérrez, A. Marin: The most ancient DNA recovered from an amber-preserved specimen may not be as ancient as it seems. In: Molecular Biology and Evolution. Band 15, Nr. 7, 1998, S. 926–929.
- ↑ Jeremy J. Austin, Andrew B. Smith, Richard H. Thomas: Palaeontology in a molecular world: the search for authentic ancient DNA. In: Trends in Ecology & Evolution. Band 12, Nr. 8, 1997, S. 303–306, doi:10.1016/S0169-5347(97)01102-6.
- ↑ Beth Shapiro, M. Hofreiter: Ancient DNA. (PDF; 456 kB) Humana Press, 2012, Kapitel V.15, S. 475–481.
- ↑ Ann H. Reid et al.: 1918 Influenza Pandemic and Highly Conserved Viruses with Two Receptor-Binding Variants. In: Emerging Infectious Diseases. [serial online]. Band 9, Nr. 10, 2003, doi:10.3201/eid0910.020789
- ↑ Terry Fei Fan Ng et al.: Preservation of viral genomes in 700-y-old caribou feces from a subarctic ice patch. In: PNAS. Band 111, Nr. 47, 2014, S. 16842–16847, doi:10.1073/pnas.1410429111, Volltext
- ↑ Verena J. Schuenemann et al.: Genome-Wide Comparison of Medieval and Modern Mycobacterium leprae. In: Science. Band 341, Nr. 6142, 2013, S. 179–183, doi:10.1126/science.1238286
- ↑ Abigail S. Bouwman, Terence A. Brown: The limits of biomolecular palaeopathology: ancient DNA cannot be used to study venereal syphilis. In: Journal of Archaeological Science. Band 32, Nr. 5, 2005, S. 703–713, doi:10.1016/j.jas.2004.11.014
- ↑ Abigail S. Bouwman et al.: Genotype of a historic strain of Mycobacterium tuberculosis. In: PNAS. Band 109, Nr. 45, 2012, S. 18511–18516, doi:10.1073/pnas.1209444109
- ↑ Kirsten I. Bos et al.: Pre-Columbian mycobacterial genomes reveal seals as a source of New World human tuberculosis. In: Nature. Band 514, 2014, S. 494–497, doi:10.1038/nature13591
- ↑ Bruce M. Rothschild et al.: Mycobacterium tuberculosis Complex DNA from an Extinct Bison Dated 17,000 Years before the Present. In: Clinical Infectious Diseases. Band 33, Nr. 3, 2001, S. 305–311, doi:10.1086/321886
- ↑ Kirsten I. Bos et al.: A draft genome of Yersinia pestis from victims of the Black Death. In: Nature. Band 478, Nr. 7370, 2011, S. 506–510, doi:10.1038/nature10549.
- ↑ Sarah A. Palmer et al.: Archaeogenomic Evidence of Punctuated Genome Evolution in Gossypium. In: Molecular Biology and Evolution. Band 29, Nr. 8, 2012, S. 2031–2038, doi:10.1093/molbev/mss070
- ↑ Sandra L. Bunning et al.: Next generation sequencing of DNA in 3300-year-old charred cereal grains. In: Journal of Archaeological Science. Band 39, Nr. 8, 2012, S. 2780–2784, doi:10.1016/j.jas.2012.04.012
- ↑ Michael D. Martin et al.: Reconstructing genome evolution in historic samples of the Irish potato famine pathogen. In: Nature Communications. Band 4, Artikel Nr. 2172, 2013, doi:10.1038/ncomms3172
- ↑ Pierre Goloubinoff et al.: Evolution of maize inferred from sequence diversity of an Adh2 gene segment from archaeological specimens. In: PNAS. Band 90, Nr. 5, 1993, S. 1997–2001, doi:10.1073/pnas.90.5.1997
- ↑ Michael D. Sorenson et al.: Relationships of the extinct moa-nalos, flightless Hawaiian waterfowl, based on ancient DNA. In: Proceedings of the Royal Society of London B: Biological Sciences. Band 266, Nr. 1434, 1999, S. 2187–2193, doi:10.1098/rspb.1999.0907
- ↑ Michael Bunce et al.: Ancient DNA provides new insights into the evolutionary history of New Zealand’s giant eagle. In: PLoS Biology. Band 3, Nr. 1, e9, 2005, doi:10.1371/journal.pbio.0030009
- ↑ Beth Shapiro et al.: Flight of the Dodo. In: Science. Band 295, Nr. 5560, 2002, S. 1683, doi:10.1126/science.295.5560.1683, Volltext (PDF; 237 kB)
- ↑ Brandon R. Menzies et al.: Limited Genetic Diversity Preceded Extinction of the Tasmanian Tiger. In: PLoS ONE. Band 7, Nr. 4, S. e35433, doi:10.1371/journal.pone.0035433
- ↑ Alex D. Greenwood, Jose Castresana, Gertraud Feldmaier-Fuchs und Svante Pääbo: A Molecular Phylogeny of Two Extinct Sloths. In: Molecular Phylogenetics and Evolution. Band 18, Nr. 1, 2001, S. 94–103, doi:10.1006/mpev.2000.0860
- ↑ Frédéric Delsuc, Melanie Kuch, Gillian C. Gibb, Emil Karpinski, Dirk Hackenberger, Paul Szpak, Jorge G. Martínez, Jim I. Mead, H. Gregory McDonald, Ross D. E. MacPhee, Guillaume Billet, Lionel Hautier und Hendrik N. Poinar: Ancient mitogenomes reveal the evolutionary history and biogeography of sloths. In: Current Biology. Band 29, Nr. 12, 2019, S. 2031–2042, doi:10.1016/j.cub.2019.05.043
- ↑ Samantha Presslee, Graham J. Slater, François Pujos, Analía M. Forasiepi, Roman Fischer, Kelly Molloy, Meaghan Mackie, Jesper V. Olsen, Alejandro Kramarz, Matías Taglioretti, Fernando Scaglia, Maximiliano Lezcano, José Luis Lanata, John Southon, Robert Feranec, Jonathan Bloch, Adam Hajduk, Fabiana M. Martin, Rodolfo Salas Gismondi, Marcelo Reguero, Christian de Muizon, Alex Greenwood, Brian T. Chait, Kirsty Penkman, Matthew Collins und Ross D. E. MacPhee: Palaeoproteomics resolves sloth relationships. In: Nature Ecology & Evolution. Band 3, 2019, S. 1121–1130, doi:10.1038/s41559-019-0909-z
- ↑ Frédéric Delsuc, Gillian C. Gibb, Melanie Kuch, Guillaume Billet, Lionel Hautier, John Southon, Jean-Marie Rouillard, Juan Carlos Fernicola, Sergio F. Vizcaíno, Ross D. E. MacPhee und Hendrik N. Poinar: The phylogenetic affinities of the extinct glyptodonts. In: Current Biology. Band 26, 2016, S. R155–R156, doi:10.1016/j.cub.2016.01.039
- ↑ Hendrik N. Poinar, Carsten Schwarz, Ji Qi, Beth Shapiro, Ross D. E. MacPhee, Bernard Buigues, Alexei Tikhonov, Daniel H. Huson, Lynn P. Tomsho, Alexander Auch, Markus Rampp, Webb Miller und Stephan C. Schuster: Metagenomics to Paleogenomics: Large-Scale Sequencing of Mammoth DNA. In: Science. Band 311, Nr. 5759, 2006, S. 392–394, doi:10.1126/science.1123360
- ↑ a b Nadin Rohland, Anna-Sapfo Malaspinas, Joshua L. Pollack, Montgomery Slatkin, Paul Matheus und Michael Hofreiter: Proboscidean Mitogenomics: Chronology and Mode of Elephant Evolution Using Mastodon as Outgroup. In: PLoS Biology Band 5, Nr. 8, 2007, S. e207, doi:10.1371/journal.pbio.0050207
- ↑ a b Nadin Rohland, David Reich, Swapan Mallick, Matthias Meyer, Richard E. Green, Nicholas J. Georgiadis, Alfred L. Roca und Michael Hofreiter: Genomic DNA Sequences from Mastodon and Woolly Mammoth Reveal Deep Speciation of Forest and Savanna Elephants. In: PLoS Biology Band 8, Nr. 12, 2010, S. e1000564, doi:10.1371/journal.pbio.1000564
- ↑ Kevin L. Campbell, Jason E. E. Roberts, Laura N, Watson, Jörg Stetefeld, Angela M. Sloan, Anthony V. Signore, Jesse W. Howatt, Jeremy R. H. Tame, Nadin Rohland, Tong-Jian Shen, Jeremy J. Austin, Michael Hofreiter, Chien Ho, Roy E Weber und Alan Cooper: Substitutions in woolly mammoth hemoglobin confer biochemical properties adaptive for cold tolerance. In: Nature Genetics. Band 42, 2010, S. 536–540, doi:10.1038/ng.574
- ↑ Eleftheria Palkopoulou, Swapan Mallick, Pontus Skoglund, Jacob Enk, Nadin Rohland, Heng Li, Ayça Omrak, Sergey Vartanyan, Hendrik Poinar, Anders Götherström, David Reich und Love Dalén: Complete Genomes Reveal Signatures of Demographic and Genetic Declines in the Woolly Mammoth. In: Current Biology. Band 25, 2015, S. 1395–1400, doi:10.1016/j.cub.2015.04.007
- ↑ Erin Fry, Sun K. Kim, Sravanthi Chigurapti, Katelyn M. Mika, Aakrosh Ratan, Alexander Dammermann, Brian J. Mitchell, Webb Miller und Vincent J Lynch: Functional Architecture of Deleterious Genetic Variants in the Genome of a Wrangel Island Mammoth. In: Genome Biology and Evolution. Band 12, Nr. 3, 2020, S. 48–58, doi:10.1093/gbe/evz279
- ↑ Tom van der Valk, Patrícia Pečnerová, David Díez-del-Molino, Anders Bergström, Jonas Oppenheimer, Stefanie Hartmann, Georgios Xenikoudakis, Jessica A. Thomas, Marianne Dehasque, Ekin Sağlıcan, Fatma Rabia Fidan, Ian Barnes, Shanlin Liu, Mehmet Somel, Peter D. Heintzman, Pavel Nikolskiy, Beth Shapiro, Pontus Skoglund, Michael Hofreiter, Adrian M. Lister, Anders Götherström und Love Dalén: Million-year-old DNA sheds light on the genomic history of mammoths. Nature, 2021, doi:10.1038/s41586-021-03224-9
- ↑ Jacob Enk, Alison Devault, Regis Debruyne, Christine E King, Todd Treangen, Dennis O’Rourke, Steven L Salzberg, Daniel Fisher, Ross MacPhee und Hendrik Poinar: Complete Columbian mammoth mitogenome suggests interbreeding with woolly mammoths. In: Genome Biology. Band 12, 2011, S. R51 (S. 1–29), doi:10.1186/gb-2011-12-5-r51
- ↑ Nikos Poulakakis, Aris Parmakelis, Petros Lymberakis, Moysis Mylonas, Eleftherios Zouros, David S. Reese, Scott Glaberman und Adalgisa Caccone: Ancient DNA forces reconsideration of evolutionary history of Mediterranean pygmy elephantids. In: Biology Letters. Band 2, Nr. 3, 2006, S. 451–454, doi:10.1098/rsbl.2006.0467
- ↑ Matthias Meyer, Eleftheria Palkopoulou, Sina Baleka, Mathias Stiller, Kirsty E. H. Penkman, Kurt W. Alt, Yasuko Ishida, Dietrich Mania, Swapan Mallick, Tom Meijer, Harald Meller, Sarah Nagel, Birgit Nickel, Sven Ostritz, Nadin Rohland, Karol Schauer, Tim Schüler, Alfred L Roca, David Reich, Beth Shapiro und Michael Hofreiter: Palaeogenomes of Eurasian straight-tusked elephants challenge the current view of elephant evolution. In: eLife. Band 6, 2017, S. e25413, doi:10.7554/eLife.25413
- ↑ Eleftheria Palkopoulou, Mark Lipson, Swapan Mallick, Svend Nielsen, Nadin Rohland, Sina Baleka, Emil Karpinski, Atma M. Ivancevic, Thu-Hien To, R. Daniel Kortschak, Joy M. Raison, Zhipeng Qu, Tat-Jun Chin, Kurt W. Alt, Stefan Claesson, Love Dalén, Ross D. E. MacPhee, Harald Meller, Alfred L. Roca, Oliver A. Ryder, David Heiman, Sarah Young, Matthew Breen, Christina Williams, Bronwen L. Aken, Magali Ruffier, Elinor Karlsson, Jeremy Johnson, Federica Di Palma, Jessica Alfoldi, David L. Adelson, Thomas Mailund, Kasper Munch, Kerstin Lindblad-Toh, Michael Hofreiter, Hendrik Poinar und David Reich: A comprehensive genomic history of extinct and living elephants. In: PNAS. Band 115, Nr. 11, 2018, S. E2566–E2574, doi:10.1073/pnas.1720554115
- ↑ Michael Buckley: A Molecular Phylogeny of Plesiorycteropus Reassigns the Extinct Mammalian Order ‘Bibymalagasia’. In: PlosOne. Band 8, Nr. 3, 2013, S. e59614, doi:10.1371/journal.pone.0059614
- ↑ Alex D. Greenwood, Cristian Capelli, Göran Possnert und Svante Pääbo: Nuclear DNA sequences from late Pleistocene megafauna. In: Molecular Biology and Evolution. Band 16, 1999, S. 1466–1473, Volltext (PDF; 153 kB)
- ↑ Axel Barlow, Johanna L. A. Paijmans, Federica Alberti, Boris Gasparyan, Guy Bar-Oz, Ron Pinhasi, Irina Foronova, Andrey Y. Puzachenko, Martina Pacher, Love Dalén, Gennady Baryshnikov und Michael Hofreiter: Middle Pleistocene genome calibrates a revised evolutionary history of extinct cave bears. In: Current Biology., 2021, doi:10.1016/j.cub.2021.01.073
- ↑ James P. Noonan, Michael Hofreiter, Doug Smith, James R. Priest, Nadin Rohland, Gernot Rabeder, Johannes Krause, J. Chris Detter, Svante Pääbo und Edward M. Rubin: Genomic Sequencing of Pleistocene Cave Bears. In: Science. Band 309, Nr. 5734, 2005, S. 597–599, doi:10.1126/science.1113485
- ↑ Webb Miller, Stephan C. Schuster, Andreanna J. Welch, Aakrosh Ratan, Oscar C. Bedoya-Reina, Fangqing Zhao, Hie Lim Kim, Richard C. Burhans, Daniela I. Drautz, Nicola E. Wittekindt, Lynn P. Tomsho, Enrique Ibarra-Laclette, Luis Herrera-Estrella, Elizabeth Peacock, Sean Farley, George K. Sage, Karyn Rode, Martyn Obbardi, Rafael Montiel, Lutz Bachmann, Ólafur Ingólfsson, Jon Aars, Thomas Mailund, Øystein Wiig, Sandra L. Talbot und Charlotte Lindqvist: Polar and brown bear genomes reveal ancient admixture and demographic footprints of past climate change. In: PNAS. Band 109, Nr. 36, 2012, S. E2382–E2390, doi:10.1073/pnas.1210506109
- ↑ Nadin Rohland, Joshua L. Pollack, Doris Nagel, Cédric Beauval, Jean Airvaux, Svante Pääbo und Michael Hofreiter: The Population History of Extant and Extinct Hyenas. In: Molecular Biology and Evolution. Band 22, Nr. 12, S. 2435–2443, doi:10.1093/molbev/msi244
- ↑ Michael V. Westbury, Stefanie Hartmann, Axel Barlow, Michaela Preick, Bogdan Ridush, Doris Nagel, Thomas Rathgeber, Reinhard Ziegler, Gennady Baryshnikov, Guilian Sheng, Arne Ludwig, Ingrid Wiesel, Love Dalen, Faysal Bibi, Lars Werdelin, Rasmus Heller und Michael Hofreiter: Hyena paleogenomes reveal a complex evolutionary history of cross-continental gene flow between spotted and cave hyena. In: Science Advances. Band 6, Nr. 11, 2020, S. eaay0456, doi:10.1126/sciadv.aay0456
- ↑ Joachim Burger, Wilfried Rosendahl, Odile Loreille, Helmut Hemmer, Torsten Eriksson, Anders Götherström, Jennifer Hiller, Matthew J. Collins, Timothy Wess und Kurt W. Alt: Molecular phylogeny of the extinct cave lion Panthera leo spelaea. In: Molecular Phylogenetics and Evolution. Band 30, Nr. 3, 2004, S. 841–849
- ↑ R. Barnett, B. Shapiro, I. Barnes, S. Y. Ho, J. Burger, N. Yamaguchi, T. F. Higham, H. T. Wheeler, W. Rosendahl, A. V. Sher, M. Sotnikova, T. Kuznetsova, G. F. Baryshnikov, L. D. Martin, C. R. Harington, J. A. Burns und A. Cooper: Phylogeography of lions (Panthera leo ssp.) reveals three distinct taxa and a late Pleistocene reduction in genetic diversity. In: Molecular ecology. Band 18, Nr. 8, 2009, S. 1668–1677, doi:10.1111/j.1365-294X.2009.04134.x
- ↑ David W. G. Stanton, Federica Alberti, Valery Plotnikov, Semyon Androsov, Semyon Grigoriev, Sergey Fedorov, Pavel Kosintsev, Doris Nagel, Sergey Vartanyan, Ian Barnes, Ross Barnett, Erik Ersmark, Doris Döppes, Mietje Germonpré, Michael Hofreiter, Wilfried Rosendahl, Pontus Skoglund und Love Dalén: Early Pleistocene origin and extensive intra‑species diversity of the extinct cave lion. In: Scientific Reports. Band 10, 2020, S. 12621, doi:10.1038/s41598-020-69474-1
- ↑ Johanna L. A. Paijmans, Ross Barnett, M. Thomas P. Gilbert, M. Lisandra Zepeta-Mendoza, Jelle W. F. Reumer, John de Voss, Grant Zazula, Doris Nagel, Gennady F. Baryshnikov, Jennifer A. Leonard, Nadine Rohland, Michael V. Westbury, Axel Barlow und Michael Hofreiter: Evolutionary history of sabre-toothed cats based on ancient mitogenomics. In: Current Biology. Band 27, 2017, S. 3330–3336, doi:10.1016/j.cub.2017.09.033
- ↑ Ross Barnett, Michael V. Westbury, Marcela Sandoval-Velasco, Filipe Garrett Vieira, Sungwon Jeon, Grant Zazula, Michael D. Martin, Simon Y. W. Ho, Niklas Mather, Shyam Gopalakrishnan, Jazmín Ramos-Madrigal, Marc de Manuel, M. Lisandra Zepeda-Mendoza, Agostinho Antunes, Aldo Carmona Baez, Binia De Cahsan, Greger Larson, Stephen J. O’Brien, Eduardo Eizirik, Warren E. Johnson, Klaus-Peter Koepfli, Andreas Wilting, Jörns Fickel, Love Dalén, Eline D. Lorenzen, Tomas Marques-Bonet, Anders J. Hansen, Guojie Zhang, Jong Bhak, Nobuyuki Yamaguchi, M. Thomas und P. Gilbert: Genomic Adaptations and Evolutionary History of the Extinct Scimitar-Toothed Cat, Homotherium latidens. In: Current Biology., 2020, doi:10.1016/j.cub.2020.09.051
- ↑ A. R. Perri, K. J. Mitchell, A. Mouton, S. Álvarez-Carretero, A. Hulme-Beaman, J. Haile, A. Jamieson, J. Meachen, A. T. Lin, B. W. Schubert, C. Ameen, E. E. Antipina, P. Bover, S. Brace, A. Carmagnini, C. Carøe, J. A. Samaniego Castruita, J. C. Chatters, K. Dobney, M. dos Reis, A. Evin, P. Gaubert, S. Gopalakrishnan, G. Gower, H. Heiniger, K. M. Helgen, J. Kapp, P. A. Kosintsev, A. Linderholm, A. T. Ozga, S. Presslee, A. T. Salis, N. F. Saremi, C. Shew, K. Skerry, D. E. Taranenko, M. Thompson, M. V. Sablin, Y. V. Kuzmin, M. J. Collins, M.-H. S, Sinding, M. T. P. Gilbert, A. C. Stone, B. Shapiro, B. Van Valkenburgh, R. K. Wayne, G. Larson, A. Cooper und L. A. F. Frantz: Dire wolves were the last of an ancient New World canid lineage. In: Nature., 2021, doi:10.1038/s41586-020-03082-x
- ↑ A. M. Lister, C. J. Edwards, D. A. W. Nock, M. Bunce, I. A. van Pijlen, D. G. Bradley, M. G. Thomas und I. Barnes: The phylogenetic position of the ‘giant deer’ Megaloceros giganteus. In: Nature. Band 438, 2005, S. 850–853
- ↑ Sandrine Hughes, Thomas J. Hayden, Christophe J. Douady, Christelle Tougard, Mietje Germonpré, Anthony Stuart, Lyudmila Lbova, Ruth F.Carden, Catherine Hänni und Ludovic Say: Molecular phylogeny of the extinct giant deer, Megaloceros giganteus. In: Molecular Phylogenetics and Evolution. Band 40, Nr. 1, 2006, S. 285–291, doi:10.1016/j.ympev.2006.02.004
- ↑ K. Doan, F. E. Zachos, B. Wilkens, J.-D. Vigne, N. Piotrowska, A. Stanković, B. Jędrzejewska, K. Stefaniak und M. Niedziałkowska: Phylogeography of the Tyrrhenian red deer (Cervus elaphus corsicanus) resolved using ancient DNA of radiocarbon-dated subfossils. In: Scientific Reports. Band 7, 2017, S. 2331, doi:10.1038/s41598-017-02359-y
- ↑ Beth Shapiro, Alexei J. Drummond, Andrew Rambaut, Michael C. Wilson, Paul E. Matheus, Andrei V. Sher, Oliver G. Pybus, M. Thomas P. Gilbert, Ian Barnes, Jonas Binladen, Eske Willerslev, Anders J. Hansen, Gennady F. Baryshnikov, James A. Burns, Sergei Davydov, 10 Jonathan C. Driver, Duane G. Froese, C. Richard Harington, Grant Keddie, Pavel Kosintsev, Michael L. Kunz, Larry D. Martin, Robert O. Stephenson, John Storer, Richard Tedford, Sergei Zimov und Alan Cooper: Rise and Fall of the Beringian Steppe Bison. In: Science. Band 306, Nr. 5701, 2004, S. 1561–1565, doi:10.1126/science.1101074
- ↑ Pauline Palacio, Véronique Berthonaud, Claude Guérin, Josie Lambourdière, Frédéric Maksud, Michel Philippe, Delphine Plaire, Thomas Stafford, Marie-Claude Marsolier-Kergoat und Jean-Marc Elalouf: Genome data on the extinct Bison schoetensacki establish it as a sister species of the extant European bison (Bison bonasus). In: BMC Evolutionary Biology. Band 17, 2017, S. 48 doi:10.1186/s12862-017-0894-2
- ↑ Albano Beja-Pereira, David Caramelli, Carles Lalueza-Fox, Cristiano Vernesi, Nuno Ferrand, Antonella Casoli, Felix Goyache, Luis J. Royo, Serena Conti, Martina Lari, Andrea Martini, Lahousine Ouragh, Ayed Magid, Abdulkarim Atash, Attila Zsolnai, Paolo Boscato, Costas Triantaphylidis, Konstantoula Ploumi, Luca Sineo, Francesco Mallegni, Pierre Taberlet, Georg Erhardt, Lourdes Sampietro, Jaume Bertranpetit, Guido Barbujani, Gordon Luikart und Giorgio Bertorelle: The origin of European cattle: Evidence from modern and ancient DNA. In: PNAS. Band 103, Nr. 21, 2006, S. 8113–8118, doi:10.1073/pnas.0509210103
- ↑ Oscar Ramírez, Elena Gigli, Pere Bover, Josep Antoni Alcover, Jaume Bertranpetit, Jose Castresana und Carles Lalueza-Fox: Paleogenomics in a Temperate Environment: Shotgun Sequencing from an Extinct Mediterranean Caprine. In: PLoS ONE. Band 4, Nr. 5, S. e5670, doi:10.1371/journal.pone.0005670
- ↑ M. D. Teasdale, N. L. van Doorn, S. Fiddyment, C.C.Webb, T. O’Connor, M. Hofreiter, M. J. Collins und D. G. Bradley: Paging through history: parchment as a reservoir of ancient DNA for next generation sequencing. In: Philosophical Transactions B. Band 370, 2017, S. 20130379, doi:10.1098/rstb.2013.0379
- ↑ Russell Higuchi et al.: DNA sequences from the quagga, an extinct member of the horse family. In: Nature. Band 312, 1984, S. 282–284, doi:10.1038/312282a0
- ↑ Jennifer A. Leonard, Nadin Rohland, Scott Glaberman, Robert C. Fleischer, Adalgisa Caccone und Michael Hofreiter: A rapid loss of stripes: the evolutionary history of the extinct quagga. In: Biological Letters. Band 1, 2005, S. 291–295
- ↑ Beth Shapiro und Michael Hofreiter: A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA. In: Science. Band 343, Nr. 6165, 2014, doi:10.1126/science.1236573
- ↑ Ludovic Orlando, Aurélien Ginolhac, Guojie Zhang, Duane Froese, Anders Albrechtsen, Mathias Stiller, Mikkel Schubert, Enrico Cappellini, Bent Petersen, Ida Moltke, Philip L. F. Johnson, Matteo Fumagalli, Julia T. Vilstrup, Maanasa Raghavan, Thorfinn Korneliussen, Anna-Sapfo Malaspinas, Josef Vogt, Damian Szklarczyk, Christian D. Kelstrup, Jakob Vinther, Andrei Dolocan, Jesper Stenderup, Amhed M. V. Velazquez, James Cahill, Morten Rasmussen, Xiaoli Wang, Jiumeng Min, Grant D. Zazula, Andaine Seguin-Orlando, Cecilie Mortensen, Kim Magnussen, John F. Thompson, Jacobo Weinstock, Kristian Gregersen, Knut H. Røed, Véra Eisenmann, Carl J. Rubin, Donald C. Miller, Douglas F. Antczak, Mads F. Bertelsen, Søren Brunak, Khaled A. S. Al-Rasheid, Oliver Ryder, Leif Andersson, John Mundy, Anders Krogh, M. Thomas P. Gilbert, Kurt Kjær, Thomas Sicheritz-Ponten, Lars Juhl Jensen, Jesper V. Olsen, Michael Hofreiter, Rasmus Nielsen, Beth Shapiro, Jun Wang und Eske Willerslev: Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse. In: Nature. Band 499, 2013, S. 74–78, doi:10.1038/nature12323
- ↑ Ludovic Orlando, Jessica L. Metcalf, Maria T. Alberdi, Miguel Telles-Antunes, Dominique Bonjean, Marcel Otte, Fabiana Martin, Véra Eisenmann, Marjan Mashkour, Flavia Morello, Jose L. Prado, Rodolfo Salas-Gismondi, Bruce J. Shockey, Patrick J. Wrinn, Sergei K. Vasil’ev, Nikolai D. Ovodov, Michael I. Cherry Blair Hopwood, Dean Male, Jeremy J. Austin, Catherine Hänni und Alan Cooper: Revising the recent evolutionary history of equids using ancient DNA. In: PNAS. Band 106, 2009, S. 21754–21759
- ↑ Charleen Gaunitz, Antoine Fages, Kristian Hanghøj, Anders Albrechtsen, Naveed Khan, Mikkel Schubert, Andaine Seguin-Orlando, Ivy J. Owens, Sabine Felkel, Olivier Bignon-Lau, Peter de Barros Damgaard, Alissa Mittnik, Azadeh F. Mohaseb, Hossein Davoudi, Saleh Alquraishi, Ahmed H. Alfarhan, Khaled A. S. Al-Rasheid, Eric Crubézy, Norbert Benecke, Sandra Olsen, Dorcas Brown, David Anthony, Ken Massy, Vladimir Pitulko, Aleksei Kasparov, Gottfried Brem, Michael Hofreiter, Gulmira Mukhtarova, Nurbol Baimukhanov, Lembi Lõugas, Vedat Onar, Philipp W. Stockhammer, Johannes Krause, Bazartseren Boldgiv, Sainbileg Undrakhbold, Diimaajav Erdenebaatar, Sébastien Lepetz, Marjan Mashkour, Arne Ludwig, Barbara Wallner, Victor Merz, Ilja Merz, Viktor Zaibert, Eske Willerslev, Pablo Librado, Alan K. Outram und Ludovic Orlando: Ancient genomes revisit the ancestry of domestic and Przewalski’s horses. In: Science. Band 360, Nr. 6384, 2018, S. 111–114, doi:10.1126/science.aao3297
- ↑ Peter D. Heintzman, Grant D. Zazula, Ross D. E. MacPhee, Eric Scott, James A. Cahill, Brianna K. McHorse, Joshua D. Kapp, Mathias Stiller, Matthew J. Wooller, Ludovic Orlando, John Southon, Duane G. Froese und Beth Shapiro: A new genus of horse from Pleistocene North America. In: eLife. Band 6, 2017, S. e29944, doi:10.7554/eLife.29944
- ↑ Ludovic Orlando, Jennifer A. Leonard, Aurélie Thenot, Vincent Laudet, Claude Guerin und Catherine Hänni: Ancient DNA analysis reveals woolly rhino evolutionary relationships. In: Molecular Phylogenetics and Evolution. Band 28, 2003, S. 485–499
- ↑ Eske Willerslev, M Thomas P. Gilbert, Jonas Binladen, Simon Y. W. Ho, Paula F. Campos, Aakrosh Ratan, Lynn P. Tomsho, Rute R. da Fonseca, Andrei Sher, Tatanya V. Kuznetsova, Malgosia Nowak-Kemp, Terri L. Roth, Webb Miller und Stephan C Schuster: Analysis of complete mitochondrial genomes from extinct and extant rhinoceroses reveals lack of phylogenetic resolution. In: BMC Evolutionary Biology. Band 9, 2009, S. 95, doi:10.1186/1471-2148-9-95
- ↑ Yuan JunXia, Sheng GuiLian, Hou XinDong, Shuang XiaoYan, Yi Jian, Yang Hong und Lai XuLong: Ancient DNA sequences from Coelodonta antiquitatisin China reveal its divergence and phylogeny. In: Science China: Earth Sciences. Band 57, Nr. 3, 2014, S. 388–396, doi:10.1007/s11430-013-4702-6
- ↑ Edana Lord, Nicolas Dussex, Marcin Kierczak, David Díez-del-Molino, Oliver A. Ryder, David W. G. Stanton, M. Thomas P. Gilbert, Fátima Sánchez-Barreiro, Guojie Zhang, Mikkel-Holger S. Sinding, Eline D. Lorenzen, Eske Willerslev, Albert Protopopov, Fedor Shidlovskiy, Sergey Fedorov, Hervé Bocherens, Senthilvel K. S. S. Nathan und Benoit Goossens: Pre-extinction Demographic Stability and Genomic Signatures of Adaptation in the Woolly Rhinoceros. In: Current Biology., 2020, doi:10.1016/j.cub.2020.07.046
- ↑ Irina V. Kirillova, Olga F. Chernova, Jan van der Made und Vladimir V. Kukarskih: Discovery of the skull of Stephanorhinus kirchbergensis (Jäger, 1839) above the Arctic Circle. In: Quaternary Research. Band 88, 2017, S. 537–550, doi:10.1017/qua.2017.53
- ↑ Frido Welker, Geoff M. Smith, Jarod M. Hutson, Lutz Kindler, Alejandro Garcia-Moreno, Aritza Villaluenga, Elaine Turner und Sabine Gaudzinski-Windheuser: Middle Pleistocene protein sequences from the rhinoceros genus Stephanorhinus and the phylogeny of extant and extinct Middle/Late Pleistocene Rhinocerotidae. In: PeerJ. Band 5, 2017, S. e3033, doi:10.7717/peerj.3033
- ↑ Ashot Margaryan, Mikkel-Holger S. Sinding, Shanlin Liu, Filipe Garrett Vieira, Yvonne L. Chan, Senthilvel K. S. S. Nathan, Yoshan Moodley, Michael W. Bruford und M. Thomas P. Gilbert: Recent mitochondrial lineage extinction in the critically endangered Javan rhinoceros. In: Zoological Journal of the Linnean Society. Band 190, Nr. 1, 2020, S. 372–383, doi:10.1093/zoolinnean/zlaa004.
- ↑ Michael Westbury, Sina Baleka, Axel Barlow, Stefanie Hartmann, Johanna L. A. Paijmans, Alejandro Kramarz, Analıá M. Forasiepi, Mariano Bond, Javier N. Gelfo, Marcelo A. Reguero, Patricio López-Mendoza, Matias Taglioretti, Fernando Scaglia, Andrés Rinderknecht, Washington Jones, Francisco Mena, Guillaume Billet, Christian de Muizon, José Luis Aguilar, Ross D. E. MacPhee und Michael Hofreiter: A mitogenomic timetree for Darwin’s enigmatic South American mammal Macrauchenia patachonica. In: Nature Communications. Band 8, 2017, S. 15951, doi:10.1038/ncomms15951.
- ↑ Frido Welker, Matthew J. Collins, Jessica A. Thomas, Marc Wadsley, Selina Brace, Enrico Cappellini, Samuel T. Turvey, Marcelo Reguero, Javier N. Gelfo, Alejandro Kramarz, Joachim Burger, Jane Thomas-Oates, David A. Ashford, Peter D. Ashton, Keri Rowsell, Duncan M. Porter, Benedikt Kessler, Roman Fischer, Carsten Baessmann, Stephanie Kaspar, Jesper V. Olsen, Patrick Kiley, James A. Elliott, Christian D. Kelstrup, Victoria Mullin, Michael Hofreiter, Eske Willerslev, Jean-Jacques Hublin, Ludovic Orlando, Ian Barnes und Ross D. E. MacPhee: Ancient proteins resolve the evolutionary history of Darwin’s South American ungulates. In: Nature. Band 522, 2015, S. 81–84, doi:10.1038/nature142499.
- ↑ Michael Buckley: Ancient collagen reveals evolutionary history of the endemic South American ‘ungulates’. In: Proceedings of the Royal Society B. Band 282, 2015, S. 20142671, doi:10.1098/rspb.2014.2671.
- ↑ Georgios Xenikoudakis, Mayeesha Ahmed, Jacob Colt Harris, Rachel Wadleigh, Johanna L. A. Paijmans, Stefanie Hartmann, Axel Barlow, Heather Lerner und Michael Hofreiter: Ancient DNA reveals twenty million years of aquatic life in beavers. In: Current Biology. Band 30, 2020, S. R110–R111, doi:10.1016/j.cub.2019.12.041.
- ↑ Matthias Krings u. a.: Neandertal DNA Sequences and the Origin of Modern Humans. In: Cell. Band 90, Nr. 1, 1997, S. 19–30, doi:10.1016/S0092-8674(00)80310-4, Volltext (PDF; 195 kB)
- ↑ Richard E. Green u. a.: A Complete Neandertal Mitochondrial Genome Sequence Determined by High-Throughput Sequencing. In: Cell. Band 134, Nr. 3, 2008, S. 416–426, doi:10.1016/j.cell.2008.06.021.
- ↑ Richard E. Green u. a.: A draft sequence of the Neandertal Genome. In: Science. Band 328, 2010, S. 710–722, doi:10.1126/science.1188021.
- ↑ David Reich et al.: Genetic history of an archaic hominin group from Denisova Cave in Siberia. In: Nature. Band 468, Nr. 7327, 2010, S. 1053–1060, doi:10.1038/nature09710.
- ↑ Qiaomei Fu, Heng Li, Priya Moorjani, Flora Jay, Sergey M. Slepchenko, Aleksei A. Bondarev, Philip L. F. Johnson, Ayinuer Aximu-Petri, Kay Prüfer, Cesare de Filippo, Matthias Meyer, Nicolas Zwyns, Domingo C. Salazar-Garcia, Yaroslav V. Kuzmin, Susan G. Keates, Pavel A. Kosintsev, Dmitry I. Razhev, Michael P. Richards, Nikolai V. Peristov, Michael Lachmann, Katerina Douka, Thomas F. G. Higham, Montgomery Slatkin, Jean-Jacques Hublin, David Reich, Janet Kelso, T. Bence Viola, Svante Pääbo: Genome sequence of a 45,000-year-old modern human from western Siberia. In: Nature. Band 514, Nr. 7523, 2014, S. 445–449, doi:10.1038/nature13810.
- ↑ Svante Pääbo: Molecular cloning of ancient Egyptian mummy DNA. In: Nature. Band 314, Nr. 6012, 1985, S. 644–645, doi:10.1038/314644a0
- ↑ Jane Gitschier: Imagine: An Interview with Svante Pääbo. In: PLOS Genetics. Band 4, Nr. 3, 2008, e1000035. doi:10.1371/journal.pgen.1000035
- ↑ Nick Zagorski: Profile of Svante Pääbo. In: PNAS. Band 103, Nr. 37, 2006, S. 13575–13577, doi:10.1073/pnas.0606596103.
- ↑ Verena J. Schuenemann et al.: Ancient Egyptian mummy genomes suggest an increase of Sub-Saharan African ancestry in post-Roman periods. In: Nature Communications. Band 8, Artikel-Nr. 15694, 2017, doi:10.1038/ncomms15694.
- ↑ K. Anslinger, G. Weichhold, W. Keil, B. Bayer, W. Eisenmenger: Identification of the skeletal remains of Martin Bormann by mtDNA analysis. In: International Journal of Legal Medicine. Band 114, Nr. 3, 2001, S. 194–196, doi:10.1007/s004140000176.
- ↑ H. Ellegren: Gamla gener guldgruva för historiker. In: Forskning & Framsteg. 6/1994, S. 21–26.
- ↑ Wolfgang Haak et al.: Ancient DNA, Strontium isotopes, and osteological analyses shed light on social and kinship organization of the Later Stone Age. In: PNAS. Band 105, Nr. 47, 2008, S. 18226–18231, doi:10.1073/pnas.0807592105.