Thermolysin
| Thermolysin | ||
|---|---|---|

| ||
| Bändermodell nach PDB 2A7G. Die Zink-Bindungsstellen sind gelb, andere katalytische Aminosäuren orange, Zink hellblau, Calcium lachsfarben. | ||
| Masse/Länge Primärstruktur | 316 Aminosäuren | |
| Kofaktor | Zn2+; 4 Ca2+ | |
| Bezeichner | ||
| Gen-Name(n) | npr | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.4.24.27, Metalloprotease | |
| MEROPS | M04.001 | |
| Substrat | Xaa-+-Leu > Xaa-+-Phe | |
| Vorkommen | ||
| Homologie-Familie | Thermolysin | |
| Übergeordnetes Taxon | Bakterien | |
Thermolysin ist ein thermostabiles Enzym, das zur Gruppe der Metallopeptidasen gehört. Es wird vom thermophilen Bakterium Bacillus thermoproteolyticus sezerniert.[1] Als Endoprotease katalysiert es die Spaltung von Peptidbindungen; dabei erfolgt die Hydrolyse der Peptidbindung vor großen, hydrophoben Resten (P1' Aktivität), (beispielsweise nach Leucin, Isoleucin oder Phenylalanin) innerhalb des Polypeptides.[2]
Das Enzym trägt die EC-Nummer EC 3.4.24.27.
Allgemeines
[Bearbeiten | Quelltext bearbeiten]Thermolysin gehört zu den Zink enthaltenden Metalloproteasen und ist eines der bestuntersuchten Enzyme dieser Superfamilie. Die dreidimensionale Struktur wurde bereits 1972 von Matthews und Mitarbeitern in einer Auflösung von 2,3 Å aufgeklärt, mittlerweile wurde eine Auflösung von 1,6 Å erreicht.[3][4]
Struktur
[Bearbeiten | Quelltext bearbeiten]Thermolysin hat eine molare Masse von 34,6 kDa. Das Enzym hat eine N-terminale und eine C-terminale Domäne, die eine Spalte für sein aktives Zentrum ausbilden. Die N-terminale Domäne besteht hauptsächlich aus β-Faltblättern, die stabilere C-terminale Domäne aus α-Helices.[5]
Thermolysin benötigt ein Zinkion und vier Calciumionen. Ersteres ist für die Enzymaktivität essentiell, trägt aber kaum zur Thermostabilität bei. Das Zinkion findet sich im aktiven Zentrum. Die Thermostabilität wird durch die Calciumionen vermittelt.[6] Die Calciumionen stabilisieren die strukturelle Integrität und schützen die aus der Oberfläche des Enzyms ragenden Schleifen (loops) vor Autoproteolyse („Selbstverdauung“).[7]
Das Protein besitzt keine Disulfidbrücken.
Chemische Eigenschaften
[Bearbeiten | Quelltext bearbeiten]Die Endoprotease ist thermostabil, ihr Funktionsoptimum liegt zwischen 65 und 70 °C bei pH 8,0.[8] Bei 80 °C liegt seine strukturelle Halbwertszeit bei einer Stunde.[9] Eine Hitzedenaturation von Thermolysin erfolgt jedoch schnell und irreversibel. Dies geschieht durch Entfaltung und Autoproteolyse exponierter Sequenzen.[5]
Katalysemechanismus
[Bearbeiten | Quelltext bearbeiten]
Obzwar viele Daten über Kristallstrukturanalysen, Mutageneseexperimente und Untersuchungen der Reaktionskinetik vorliegen, steht der exakte Katalysemechanismus noch zur Debatte. Mit Hilfe von Computersimulationen aus kristallographischen Daten gilt aber folgender Reaktionsmechanismus als der wahrscheinlichste.
Im nativen Zustand liegt das Zinkion tetraedrisch koordiniert vor. Dabei bildet das Metallion einen Komplex zu drei Aminosäureresten des Enzyms (Histidin 142 (His 142), Histidin 146 (His 146) und Glutaminsäure 166 (Glu 166)) und einem Wassermolekül (vgl. auch untenstehende Abbildung). Letzteres ist allen Zink-Metalloproteinen gemeinsam.
Nach Binden des zu hydrolisierenden Peptids (Schritt 1 in der Abbildung) liegt das Zinkion pentakoordiniert vor. Gleichzeitig wird das Wassermolekül in Nähe des Glutamats 143 (Glu 143) gebracht und von diesem und dem positiv geladenen Zinkatom stark polarisiert. Dies hat zur Folge, dass die Nukleophilie des Wassermoleküls groß genug wird, um die Peptidbindung anzugreifen.
Der nukleophile Angriff (Schritt 2) auf die Peptidbindung erfolgt, dabei wird das eine Proton des Wassers durch das Glu 143 zum Stickstoff übertragen. Das Kohlenstoffatom der Peptidbindung liegt nun tetraedrisch vor. His 231 und Tyrosin (Tyr) 157 helfen dabei, den Carbonylsauerstoff zu stabilisieren. Schließlich erfolgt die Spaltung der Peptidbindung, dabei wird das zweite vom Wasser stammende Proton via Glu 143 auf das Stickstoffatom übertragen.
Das Aminprodukt verlässt das aktive Zentrum als protonierte Form (Schritt 3). Das Carboxylprodukt wird durch ein neues Wassermolekül ersetzt (Schritt 4), so dass das Zinkatom wieder tetraedrisch koordiniert vorliegt. Ein neuer Reaktionsmechanismus kann beginnen.
Der Glu-143-Rest formt mit den beiden Histidinen His 142 und 146 ein sogenanntes übereinstimmendes Sequenzmotiv, das HEXXH-Motiv. Dieses Motiv ist in allen Vertretern der Zinkendoproteasen mit einem Zinkatom hochkonserviert.

Inhibitoren
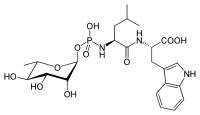
[Bearbeiten | Quelltext bearbeiten]Ein natürlicher Inhibitor von Thermolysin ist Phosphoramidon[10] aus Streptomyces tanashiensis. Der zurzeit wirksamste Hemmstoff ist das artifizielle ZFpLA (Carbobenzoxy-L-PheP-L-Leu-L-Ala).[11] In beiden Fällen liegt ein Phosphoramidat vor (eine kovalente Verbindung zwischen einem Stickstoff- und einem Phosphoratom).

Technische Anwendungen
[Bearbeiten | Quelltext bearbeiten]Das Enzym kann für die Synthese von Aspartam, einem Süßstoff, verwendet werden.[9] Dabei wird die Kondensation – also die Umkehrreaktion der Hydrolyse (reverse proteolysis) – von L--Aspartat und L-Phenylalanin zu L-Aspartyl-L-phenylalaninmethylester (=Aspartam) katalysiert.[12]
Literatur
[Bearbeiten | Quelltext bearbeiten]- Matthews, BW. (1988): Structural basis of the action of thermolysin and related zinc peptidases. In: Acc. Chem. Res. 21(9); 333–340; doi:10.1021/ar00153a003
- Pelmenschikov, V. et al. (2002): A theoretical study of the mechanism for peptide hydrolysis by thermolysin. J Biol Inorg Chem. 7(3); 284–98; PMID 11935352; doi:10.1007/s007750100295
Weblinks
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Endo, S. (1962). Studies on protease produced by thermophilic bacteria. In: J. Ferment. Technol. 40; 346–353.
- ↑ Morihara, K. und Tsuzuki, H. (1970): Thermolysin: kinetic study with oligopeptides. In: Eur J Biochem. 15(2); 374–80; PMID 4993757.
- ↑ Colman PM. et al. (1972): The structure of thermolysin: an electron density map at 2-3 Å resolution. In: J Mol Biol. 70(3): 701–24; PMID 5083153.
- ↑ Weaver LH. et al. (1976): The structure and stability of thermolysin. In: Experientia Suppl. 26; 31–9; PMID 820566.
- ↑ a b Roche, RS. und Voordouw, G. (1978): The structural and functional roles of metal ions in thermolysin. In: CRC Crit Rev Biochem. 5(1); 1–23; PMID 357082.
- ↑ Tajima, M. et al. (1976): Role of calcium ions in the thermostability of thermolysin and Bacillus subtilis var. amylosacchariticus neutral protease. In: Eur J Biochem. 64(1); 243–7; PMID 819262.
- ↑ W. Kaim/B. Schwederski, Bioanorganische Chemie, Teubner Studienbücher, 1995, ISBN 3-519-13505-1, S. 261.
- ↑ Datenblatt Thermolysin ( vom 19. Februar 2014 im Internet Archive) (PDF; 104 kB) der Firma Daiwa Kasei K.K.
- ↑ a b Rao, MB. et al. (1998): Molecular and biotechnological aspects of microbial proteases. In: Microbiol Mol Biol Rev. 62(3); 597–635; PMID 9729602; PDF (freier Volltextzugriff, engl.).
- ↑ Externe Identifikatoren von bzw. Datenbank-Links zu Phosphoramidon: CAS-Nr.: 36357-77-4, EG-Nr.: 252-996-3, ECHA-InfoCard: 100.048.164, PubChem: 445114, ChemSpider: 392848, DrugBank: DB02557, Wikidata: Q7187544.
- ↑ Externe Identifikatoren von bzw. Datenbank-Links zu ZFpLA, auch 4tmn: CAS-Nr.: 110786-00-0, PubChem: 5492587, ChemSpider: 4591060, Wikidata: Q27451311.
- ↑ Arnold Ulrich: Limitierte Proteolyse zur Analyse lokaler und globaler Änderungen der Proteinstruktur am Beispiel von Ribonucleasen. Martin-Luther-Universität Halle-Wittenberg, Habilitationsschrift, 2008, S. 35–36.