Mã di truyền

Mã di truyền (tiếng Anh: Genetic code) là bộ quy tắc mà các tế bào sống sử dụng để dịch mã thông tin được mã hóa trong vật liệu di truyền (những chuỗi bộ ba nucleotide hoặc codon của DNA hoặc RNA) thành protein. Dịch mã được ribosome tiến hành, liên kết amino acid tạo protein theo thứ tự mà RNA thông tin (mRNA) quy định, sử dụng các phân tử của RNA vận chuyển (tRNA) để mang amino acid và đọc ba nucleotide của mRNA cùng lúc. Mã di truyền rất giống nhau giữa mọi sinh vật và có thể trình bày trong bảng 64 ô.
Trong sinh tổng hợp protein, các codon có vai trò xác định amino acid nào sẽ được "lắp" vào vị trí kế tiếp trên trên chuỗi. Một bộ ba nucleotide trong trình tự acid nucleic chỉ mã hóa cho một amino acid duy nhất (trừ một vài trường hợp ngoại lệ).[1] Đa số gen được mã hóa bằng một sơ đồ duy nhất (xem bảng mã RNA). Sơ đồ đó thường được gọi là mã di truyền tiêu chuẩn, hay đơn giản là mã di truyền. Có tồn tại các mã biến thể (variant code), chẳng hạn như trong ty thể.
Lịch sử
[sửa | sửa mã nguồn]
Cấu trúc DNA được phát hiện vào năm 1953, từ thời điểm đó hàng loạt các nghiên cứu về mã hóa protein được tiến hành. Nhà sinh lý học người Anh Francis Crick và nhà sinh vật học người Mỹ James Watson làm việc cùng nhau tại Phòng thí nghiệm Cavendish của Đại học Cambridge, họ cùng đưa ra giả thuyết rằng thông tin truyền từ DNA và có mối liên hệ giữa DNA và protein.[2] Nhà vật lý người Mỹ gốc Liên Xô George Gamow là người đầu tiên tạo ra sơ đồ tổng hợp protein từ DNA.[3] Ông cho rằng bộ ba base phải được dùng để mã hóa 20 amino acid tiêu chuẩn mà các tế bào sống sử dụng để tạo nên protein, tức cho phép tối đa 43 = 64 amino acid.[4] Ông đặt tên tương tác của DNA-protein này (mã di truyền gốc) là "mã kim cương" (diamond code).[5]
Năm 1954, Gamow thành lập một tổ chức khoa học không chính thức là câu lạc bộ RNA Tie (theo đề xuất của Watson) dành cho các nhà khoa học có quan điểm khác nhau, cùng quan tâm đến cách thức protein được tổng hợp từ gen. Tuy nhiên, câu lạc bộ chỉ có 20 thành viên thường trực, mỗi thành viên đại diện cho một amino acid; và câu lạc bộ này có thêm 4 thành viên danh dự để đại diện cho bốn nucleotide của DNA.[6]
Đóng góp đầu tiên của câu lạc bộ cho khoa học về sau được ghi nhận là "một trong những bài báo chưa công bố quan trọng nhất trong lịch sử khoa học"[7] và "bài báo chưa công bố nổi tiếng nhất trong biên niên sử sinh học phân tử,"[8] do Crick tiến hành. Crick trình bày một bài báo đánh chữ có nhan đề "On Degenerate Templates and the Adaptor Hypothesis: A Note for the RNA Tie Club"[9] gửi cho các thành viên của câu lạc bộ vào tháng 1 năm 1955; bài báo này "thay đổi hoàn toàn cách mà chúng ta nghĩ về tổng hợp protein", như Watson kể lại.[10] Giả thuyết cho rằng mã bộ ba không được truyền cho các amino acid như Gamow nghĩ, mà được một phân tử khác cất giữ (một "tác nhân thích ứng" (adaptor) tương tác với các amino acid).[8] Sau này, "tác nhân thích ứng" được xác định là tRNA.[11]
Codon
[sửa | sửa mã nguồn]Thí nghiệm Crick, Brenner, Barnett và Watts-Tobin lần đầu chứng minh rằng codon bao gồm ba base DNA. Marshall Nirenberg và Heinrich J. Matthaei là những người đầu tiên tiết lộ bản chất của một mã vào năm 1961.[12]
Họ đã sử dụng một hệ thống phi tế bào (cell-free system) để dịch mã một trình tự RNA poly-uracil (ví dụ: UUUUU...) và phát hiện ra rằng polypeptide mà họ đã tổng hợp chỉ gồm có chỉ amino acid phenylalanin.[13] Do đó, họ suy luận rằng codon UUU quy định amino acid phenylalanin.[14]
Kế đến là các thí nghiệm trong phòng thí nghiệm của Severo Ochoa đã chứng minh rằng trình tự RNA poly-adenin (AAAAA...) được mã hóa cho polypeptide poly-lysin[15] và trình tự RNA poly-cytosine (CCCCC...) được mã hóa cho polypeptide poly-prolin.[16] Do đó, codon AAA quy định amino acid lysin còn codon CCC quy định amino acid prolin.[17] Nhờ sử dụng nhiều chất copolymer mà sau đó hầu hết các mã còn lại đã được xác định.[18]
Nghiên cứu của Har Gobind Khorana xác định các mã di truyền còn lại. Ngay sau đó, Robert W. Holley xác định cấu trúc của RNA vận chuyển (tRNA), phân tử thích ứng hỗ trợ quá trình dịch mã RNA thành protein. Nghiên cứu này dựa trên những nghiên cứu trước của Ochoa. Năm 1959, Ochoa nhận giải Nobel về Sinh lý học và Y học cho công trình nghiên cứu về enzym học trong tổng hợp RNA.[19]
Qua việc mở rộng nghiên cứu, Nirenberg và Philip Leder tiết lộ bản chất bộ ba của mã di truyền và giải mã các codon trong nó. Trong những thí nghiệm này, nhiều tổ hợp của mRNA được truyền qua một bộ lọc chứa ribosome, những thành phần của tế bào dịch RNA thành protein. Những bộ ba mã hóa đã thúc đẩy các tRNA cụ thể liên kết với ribosome. Leder và Nirenberg còn có thể xác định trình tự của 54 trong số 64 codon ở thí nghiệm của họ.[20] Khorana, Holley và Nirenberg cùng nhận giải Nobel năm 1968 cho công trình nghiên cứu của họ.[21]
Hai nhà khoa học khám phá ra bộ ba kết thúc là Richard Epstein và Charles Steinberg. Họ đặt tên bộ ba UAA là ochre, UAG là amber, còn UGA là opal. "Amber" được đặt theo tên của Harris Bernstein (người bạn của hai nhà khoa học trên), trong đó "bernstein" trong tiếng Đức nghĩa là hổ phách, tiếng Anh là "amber".[22] Hai codon kết thúc khác được đặt tên là "ochre" (màu thổ hoàng) và "opal" để giữ theo chủ đề màu sắc.[23]
Mã di truyền mở rộng (sinh học tổng hợp)
[sửa | sửa mã nguồn]Trong nhiều đối tượng học thuật, khái niệm về sự tiến hóa của mã di truyền từ mã di truyền gốc và mơ hồ sang mã rành mạch ("mã đông cứng"- "frozen") với danh mục 20 (+2) amino acid chính tắc được nhiều người chấp nhận.[24] Tuy nhiên lại có những ý kiến, khái niệm, cách tiếp cận và ý tưởng khác nhau, đây là cách tốt nhất để thay đổi nó bằng thực nghiệm. Thậm chí các mô hình được đề xuất để dự đoán "điểm vào" mà amino acid tổng hợp dùng để xâm nhập vào mã di truyền.[25]
Kể từ năm 2001, 40 amino acid phi tự nhiên đã được bổ sung vào protein bằng cách tạo ra một codon độc nhất (tái mã hóa) và một RNA vận chuyển tương ứng: aminoacyl – cặp tRNA-synthetase để mã hóa nó với những đặc tính hóa lý và sinh học đa dạng, với mục tiêu sử dụng như một công cụ để khám phá cấu trúc và chức năng của protein, hoặc để tạo mới hoặc tăng cường protein.[26][27] H. Murakami và M. Sisido đã mở rộng một số codon thành 4 và 5 base. Steven A. Benner thì xây dựng mã chức năng thứ 65 (in vivo).[28]
Năm 2015, N. Budisa, D. Söll và các đồng nghiệp đã thông báo sự thay thế hoàn toàn của tất cả 20.899 dư lượng tryptophan (codon UGG) bằng thienopyrrole-alanin phi tự nhiên trong mã di truyền của vi khuẩn Escherichia coli.[29] Năm 2016, sinh vật bán tổng hợp ổn định đầu tiên được tạo ra. Đó là một loại vi khuẩn (đơn bào) có hai base tổng hợp (được gọi là X và Y). Những base sống sót phân chia tế bào.[30][31] Năm 2017, các nhà nghiên cứu ở Hàn Quốc thông báo rằng họ đã tạo ra một con chuột mang mã di truyền mở rộng có thể tạo ra protein chứa amino acid phi tự nhiên.[32]
Tháng 5 năm 2019, các nhà nghiên cứu thông báo họ đã tạo ra một chủng "Syn61" mới của vi khuẩn Escherichia coli. Chủng này có bộ gen tổng hợp đầy đủ được tái cấu trúc (mọi phần chồng chéo mở rộng), tái mã hóa (loại bỏ hoàn toàn việc sử dụng ba trong số 64 codon) và được điều chỉnh thêm để loại bỏ những tRNA và các nhân tố giải phóng hiện không cần thiết. Chủng vi khuẩn này hoàn toàn sống được và phát triển chậm hơn 1,6 lần so với chủng bản sao type hoang dã "MDS42".[33][34]
Đặc tính
[sửa | sửa mã nguồn]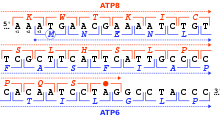
Khung đọc
[sửa | sửa mã nguồn]Một khung đọc được xác định bởi bộ ba nucleotide đầu tiên mà dịch mã dùng để khởi động. Nó thiết lập khung cho một chuỗi các codon liên tiếp và không chồng chéo, được gọi là "khung đọc mở" (ORF). Ví dụ: chuỗi 5'-AAATGAACG-3' (như hình bên), nếu được đọc từ vị trí đầu tiên thì chứa các codon AAA, TGA và ACG; nếu đọc từ vị trí thứ hai thì nó chứa codon AAT và GAA; còn nếu được đọc từ vị trí thứ ba, nó chứa các codon ATG và AAC. Vì thế mọi trình tự có thể được đọc theo hướng 5' → 3' ở ba khung đọc, mỗi khung cho ra một trình tự amino acid riêng biệt: như trong ví dụ đã cho, lần lượt là Lys (K)-Trp (W)-Thr (T), Asn (N)-Glu (E), hoặc Met (M)-Asn (N) (khi dịch mã với mã ty thể của động vật có xương sống). Khi DNA ở dạng sợi kép, 6 khung đọc có thể được xác định, ba khung theo định hướng trên một sợi và ba khung theo hướng ngược lại trên sợi đối diện.[36]:330 Các khung mã hóa protein được xác định bởi một mã mở đầu, thường là codon AUG (ATG) đầu tiên trong trình tự RNA (DNA).[37]
Ở sinh vật nhân chuẩn, ORF ở exon thường bị intron làm gián đoạn.[38]
Mã mở đầu và kết thúc
[sửa | sửa mã nguồn]Dịch mã khởi đầu bằng mã mở đầu. Chỉ riêng mã mở đầu là không đủ để bắt đầu quá trình. Các trình tự gần đó như trình tự Shine-Dalgarno ở E. coli và các yếu tố khởi đầu cũng được yêu cầu bắt đầu dịch mã. Mã mở đầu phổ biến nhất là AUG, được đọc là methionin hoặc formylmethionin (ở vi khuẩn, ty thể và lạp thể). Các mã mở đầu chuyển đổi phụ thuộc vào sinh vật kể cả "GUG" hoặc "UUG"; những codon này thường lần lượt đại diện cho valin và leucin, nhưng là dạng mã mở đầu, chúng được dịch mã thành methionin hoặc formylmethionin.[39]
Ba mã kết thúc có các tên gọi: UAG là amber, UGA là opal (đôi khi còn được gọi là umber) và UAA là ochre. Những mã kết thúc còn được gọi là mã "chấm dứt" hoặc "vô nghĩa". Chúng phát tín hiệu giải phóng polypeptide mới sinh ra từ ribosome vì không có tRNA cùng nguồn gốc nào mang các anticodon bổ sung cho các tín hiệu kết thúc này, thay vào đó cho phép một yếu tố giải phóng liên kết với ribosome.[40]
Tác động của đột biến
[sửa | sửa mã nguồn]
Trong quá trình nhân đôi DNA, đôi khi xảy ra các lỗi trong phản ứng trùng hợp ở sợi thứ hai. Những lỗi này (hay đột biến) có thể tác động đến kiểu hình của sinh vật, đặc biệt nếu chúng xảy ra trong trình tự mã hóa protein của gen. Tỷ lệ lỗi thường là 1 lỗi ở từng 10–100 triệu base—do khả năng "hiệu đính" của DNA polymerase.[41][42]
Đột biến sai nghĩa và đột biến vô nghĩa là những ví dụ về đột biến điểm có thể gây ra các bệnh di truyền như bệnh hồng cầu hình liềm và tan máu bẩm sinh.[43][44][45] Những đột biến sai nghĩa quan trọng về mặt lâm sáng thường thay đổi các đặc tính của gốc amino acid đã mã hóa ở giữa các trạng thái cơ bản, acid, phân cực hoặc không phân cực, trong khi đột biến vô nghĩa dẫn đến một mã kết thúc.[36]
Những đột biến làm gián đoạn trình tự khung đọc bằng cách thêm bớt (thêm hoặc xóa) của một base không phải bội số của 3 nucleotide được gọi là đột biến dịch khung. Những đột biến này thường dẫn đến dịch mã hoàn toàn khác với bản gốc và có thể làm mã kết thúc được đọc, rồi nó cắt ngắn protein.[46] Những đột biến này có thể làm giảm chức năng của protein và do đó rất hiếm xảy ra trong các trình tự mã hóa protein in vivo. Một lý do làm sự di truyền của các đột biến dịch khung hiếm gặp là nếu việc protein được dịch mã là cần thiết để phát triển dưới áp lực chọn lọc mà sinh vật phải đối mặt, thì việc không có protein chức năng có thể gây ra cái chết trước khi sinh vật sống được.[47] Đột biến dịch khung có thể dẫn đến các bệnh di truyền nghiêm trọng như bệnh Tay–Sachs.[48]
Mặc dù đa số đột biến làm thay đổi trình tự protein là có hại hoặc cả lợi lẫn hại, nhưng một vài đột biến lại có lợi.[49] Những đột biến này có thể cho phép sinh vật đột biến chịu được các áp lực môi trường cụ thể tốt hơn các sinh vật hoang dã hoặc sinh sản nhanh hơn. Ở những trường hợp này, đột biến sẽ có xu hướng trở nên phổ biến hơn trong quần thể thông qua chọn lọc tự nhiên.[50] Những virus sử dụng RNA làm vật liệu di truyền của chúng có tỷ lệ đột biến nhanh;[51] đây có thể là một lợi thế, vì những virus này nhờ thế mà tiến hóa nhanh chóng và tránh được các phản ứng phòng thủ của hệ miễn dịch.[52] Ở những quần thể lớn của sinh vật sinh sản vô tính, chẳng hạn như E. coli, nhiều đột biến có lợi có thể cùng xảy ra. Hiện tượng này được gọi là giao thoa vô tính và gây ra cạnh tranh giữa các đột biến.[53]
Thoái hóa
[sửa | sửa mã nguồn]
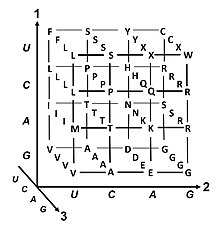
Thoái hóa là tình trạng dư thừa của mã di truyền. Thuật ngữ này được đưa ra bởi Bernfield và Nirenberg. Mã di truyền có tính dư thừa nhưng không mơ hồ (xem bảng mã bên dưới để biết mối tương quan đầy đủ). Ví dụ, mặc dù codon GAA và GAG đều xác định acid glutamic (dư thừa), cả hai không xác định amino acid nào khác (không mơ hồ). Các codon mã hóa một amino acid có thể khác nhau ở bất kỳ chỗ nào nào trong ba vị trí của chúng. Ví dụ, amino acid leucin được xác định bởi các codon YUR hoặc CUN (UUA, UUG, CUU, CUC, CUA hoặc CUG) (khác biệt ở vị trí thứ nhất hoặc thứ ba được thể hiện nhờ sử dụng ký hiệu IUPAC), trong khi amino acid serin được xác định bởi các codon UCN hoặc AGY (UCA, UCG, UCC, UCU, AGU hoặc AGC) (khác biệt ở vị trí thứ nhất, thứ hai hoặc thứ ba).[54] Một hậu quả thực tế của tính dư thừa là các lỗi ở vị trí thứ ba của codon bộ ba chỉ gây ra đột biến thầm lặng hoặc một lỗi không ảnh hưởng đến protein vì tính ưa nước hoặc kỵ nước được duy trì bằng cách thay thế các amino acid tương đương; ví dụ, một codon của NUN (trong đó N = bất kỳ nucleotide nào) có xu hướng mã hóa cho các amino acid kỵ nước. NCN tạo ra gốc amino acid có kích thước nhỏ và tính kỵ nước vừa phải; NAN mã hóa gốc ưa nước có kích thước trung bình. Mã di truyền có cấu trúc phù hợp với tính kỵ/ưa nước đến mức một phép phân tích toán học (phép phân tích suy biến) gồm 12 biến số (4 nucleotide x 3 vị trí) mang lại một mối tương quan đáng chú ý (C = 0,95) nhằm dự đoán tính kỵ/ưa nước của amino acid được mã hóa trực tiếp từ trình tự bộ ba nucleotide, mà không có dịch mã.[55][56] Lưu ý trong bảng dưới đây, 8 amino acid hoàn toàn không bị ảnh hưởng bởi đột biến ở vị trí thứ ba của codon, trong khi ở hình phía trên, đột biến ở vị trí thứ hai có thể gây ra thay đổi cơ bản về tính chất lý hóa của amino acid đã mã hóa. Tuy nhiên, những thay đổi ở vị trí đầu tiên của codon quan trọng hơn những thay đổi ở vị trí thứ hai trên phạm vi toàn cầu.[57] Lý do có thể là đảo ngược điện tích (từ điện tích dương sang điện tích âm hoặc ngược lại) chỉ có thể xảy ra khi đột biến ở vị trí đầu tiên của một số codon nhất định, chứ không thể xảy ra khi thay đổi ở vị trí thứ hai của bất kỳ codon nào. Sự đảo ngược điện tích như vậy có thể gây ra những hậu quả nghiêm trọng đối với cấu trúc hoặc chức năng của protein. Khía cạnh này có thể từng bị các nghiên cứu trước đây đánh giá thấp.[57]
Xu hướng sử dụng codon
[sửa | sửa mã nguồn]Tần suất của codon, còn được gọi là xu hướng sử dụng codon, có thể khác nhau giữa các loài với ý nghĩa chức năng về kiểm soát dịch mã. Codon thay đổi tùy theo sinh vật; ví dụ, codon prolin phổ biến nhất ở E. coli là CCG trong khi ở người, đây là cụm prolin ít được sử dụng nhất.[58]
Bảng tần suất codon bộ gen người[59]
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Mã di truyền thay thế
[sửa | sửa mã nguồn]Amino acid phi tiêu chuẩn
[sửa | sửa mã nguồn]Ở một vài protein, amino acid phi tiêu chuẩn được thay thế cho mã kết thúc tiêu chuẩn, phụ thuộc vào trình tự tín hiệu liên quan trong RNA thông tin. Ví dụ, UGA có thể mã hóa cho selenocysteine còn UAG có thể mã hóa cho pyrrolysine. Selenocysteine được xem là amino acid thứ 21 còn pyrrolysine là amino acid thứ 22.[60] Không như selenocysteine, UAG do pyrrolysine mã hóa được dịch mã với sự tham gia của một synthetase aminoacyl-tRNA riêng biệt.[61] Cả selenocysteine lẫn pyrrolysine đều có thể ở cùng một sinh vật.[60] Mặc dù mã di truyền thường được cố định trong một sinh vật, sinh vật nhân sơ cổ khuẩn Acetohalobium arabaticum có thể mở rộng mã di truyền của nó từ 20 lên 21 amino acid (bằng cách chứa cả pyrrolysine) trong các điều kiện phát triển khác nhau.[62]
Biến thể
[sửa | sửa mã nguồn]
Lúc đầu có một nhận định đơn giản và được nhiều người chấp nhận rằng mã di truyền phải có tính phổ biến: cụ thể là bất kỳ biến thể nào trong mã di truyền sẽ làm chết sinh vật (mặc dù Crick tuyên bố rằng virus là một ngoại lệ). Đây được gọi là lập luận "sự cố bất dịch" (frozen accident) về tính phổ biến của mã di truyền. Tuy nhiên, trong bài báo chuyên đề về nguồn gốc của mã di truyền năm 1968, Francis Crick vẫn tuyên bố rằng tính phổ biến của mã di truyền ở mọi sinh vật là một giả định chưa được chứng minh và có lẽ không đúng trong một vài trường hợp. Ông dự đoán rằng "Mã có tính phổ biến (giống nhau ở mọi sinh vật) hoặc gần như vậy".[64] Biến thể đầu tiên được phát hiện vào năm 1979 bởi các nhà nghiên cứu gen ty thể ở người.[65] Sau đó nhiều biến thể nhẹ đã được phát hiện,[66] bao gồm nhiều mã ty thể chuyển đổi khác nhau.[67] Ví dụ, các biến thể nhỏ này liên quan đến dịch codon UGA thành tryptophan ở các loài Mycoplasma, và dịch CUG thành serine thay vì leucine trong nấm men của "nhánh CTG" (chẳng hạn như Candida albicans).[68][69][70] Vì virus phải sử dụng chung mã di truyền làm vật chủ của chúng nên việc biến đổi mã di truyền tiêu chuẩn có thể cản trở quá trình tổng hợp hoặc hoạt động của protein virus. Tuy nhiên, các loại virus như totivirus lại thích nghi với biến đổi mã di truyền của vật chủ.[71] Ở vi khuẩn và cổ khuẩn, GUG và UUG là các codon mở đầu phổ biến. Trong một số ít trường hợp, một vài protein có thể sử dụng mã mở đầu chuyển đổi.[66] Đáng ngạc nhiên là các biến thể trong giải thích mã di truyền cũng tồn tại trong các gen mã hóa nhân ở người: Năm 2016, các nhà khoa học trong lúc nghiên cứu dịch mã của malate dehydrogenase đã phát hiện ra rằng trong khoảng 4% mRNA mã hóa enzyme này, codon kết thúc được sử dụng tự nhiên để mã hóa amino acid tryptophan và arginine.[72] Kiểu mã hóa lại này ra đời do bởi mã kết thúc đọc xuyên cao[73] và nó được gọi là đọc xuyên dịch mã chức năng (functional translational readthrough).[74]
Bất chấp những khác biệt này, mọi mã xuất hiện tự nhiên mà con người đã biết rất giống nhau. Cơ chế mã hóa giống nhau ở mọi sinh vật: codon ba base, tRNA, ribosome, đọc một hướng và dịch codon đơn lẻ thành các amino acid đơn lẻ.[75] Những biến thể cực đoan nhất xảy ra ở một số tiêm mao nhất định, nơi ý nghĩa của codon kết thúc phụ thuộc vào vị trí của chúng trong mRNA. Khi ở gần đầu 3', chúng đóng vai trò là đầu cuối trong khi ở các vị trí bên trong, chúng mã hóa amino acid như trong Condylostoma magnum[76] hay kích hoạt dịch khung ribosome (ribosomal frameshifting) như ở Euplotes.[77]
Nguồn gốc và biến thể của mã di truyền (kể cả những cơ chế đằng sau khả năng tiến hóa của mã di truyền) đã được nghiên cứu sâu rộng,[78][79] một vài nghiên cứu được tiến hành bằng thực nghiệm tiến hóa mã di truyền của một số sinh vật.[80][81][82][83]
Suy luận
[sửa | sửa mã nguồn]Mã di truyền biến thể mà một sinh vật sử dụng có thể được suy luận bằng xác định các gen bảo tồn cao được mã hóa trong bộ gen đó, rồi so sánh việc sử dụng codon của nó với amino acid trong protein tương đồng của các sinh vật khác. Ví dụ, chương trình FACIL suy luận ra mã di truyền bằng tìm xem các amino acid nào trong vùng protein tương đồng thường được đặt thẳng hàng theo từng codon nhất. Xác xuất thu được amino acid (hay mã kết thúc) ở mỗi codon được thể hiện trong một logo mã di truyền.[63]
Từ tháng 1 năm 2022, Shulgina và Eddy đã tiến hành cuộc khảo sát đầy đủ nhất về mã di truyền, họ sàng lọc 250.000 bộ gen của sinh vật nhân sơ bằng công cụ Codetta của họ. Công cụ này sử dụng cách tiếp cận tương tự như FACIL với cơ sở dữ liệu Pfam lớn hơn. Mặc dù NCBI cung cấp 33 bảng dịch mã, nhưng hai tác giả của khảo sát có thể tìm thấy 5 biến thể mã di truyền mới (được chứng thực bởi các đột biến tRNA) và sửa một số quy kết lỗi.[84]
Nguồn gốc
[sửa | sửa mã nguồn]Mã di truyền là một phần quan trọng trong lịch sử sự sống, theo một phiên bản mà các phân tử RNA tự nhân bản có trước sự sống như chúng ta biết. Đây gọi là giả thuyết thế giới RNA. Theo giả thuyết này, bất kỳ mô hình nào về sự xuất hiện của mã di truyền đều liên quan mật thiết đến mô hình vận chuyển từ ribozyme (enzym RNA) sang protein dưới dạng enzym chính trong tế bào. Nhằm tuân theo giả thuyết thế giới RNA, các phân tử RNA vận chuyển dường như tiến hóa trước các synthetase aminoacyl-tRNA hiện đại, do đó synthetase không thể là phần giải thích cho các mô hình của nó.[85]
Một mã di truyền tiến hóa ngẫu nhiên theo giả thuyết tiếp tục thúc đẩy một mô hình sinh hóa hoặc tiến hóa về nguồn gốc của nó. Nếu amino acid được xếp ngẫu nhiên vào các codon bộ ba thì có thể có 1,5×1084 mã di truyền.[86] Con số này được tìm ra bằng phép tính số cách mà 21 món đồ (20 amino acid cộng với một mã kết thúc) có thể đựng trong 64 thùng, trong đó mỗi món đồ được sử dụng ít nhất một lần.[87] Tuy nhiên, sự phân chia các codon trong mã di truyền không ngẫu nhiên.[88] Cụ thể, mã di truyền tập hợp một số phần chia amino acid nhất định.
Những amino acid chung con đường sinh tổng hợp có xu hướng mang cùng base đầu tiên trong các codon của chúng. Đây có thể là một sự học lại tiến hóa của một mã di truyền sơ khai và đơn giản hơn với ít amino acid hơn, mà sau này đã tiến hóa để mã hóa một bộ amino acid lớn hơn.[89] Điều này cũng có thể phản ánh tính chất hóa học và không gian tác động khác đến codon trong quá trình tiến hóa của amino acid ấy. Những amino acid có tính chất vật lý tương tự cũng có xu hướng chứa các codon tương tự,[90][91] làm giảm các vấn đề mà đột biến điểm và dịch mã sai gây ra.[88]
Bằng giản đồ mã hóa bộ ba di truyền không ngẫu nhiên, một giả thuyết chấp nhận được về nguồn gốc của mã di truyền có thể giải quyết nhiều khía cạnh của bảng codon, chẳng hạn như vắng các codon cho acid D-amino, mẫu codon thứ cấp cho một số amino acid, sự hạn chế các vị trí tương đồng tới vị trí thứ ba, bộ mã nhỏ chỉ có 20 amino acid (thay vì một số bộ có gần 64 amino acid) và mối quan hệ của các mẫu mã kết thúc với các mẫu mã hóa amino acid.[92]
Có ba giả thuyết chính nhắc đến nguồn gốc của mã di truyền. Nhiều mô hình thuộc về một trong số chúng hoặc kết hợp nhiều giả thuyết:[93]
- Đóng băng ngẫu nhiên (Random freeze): mã di truyền được tạo ra ngẫu nhiên. Ví dụ, những ribozym kiểu tRNA ban đầu có thể mang các ái lực khác nhau đối với amino acid; các codon xuất hiện từ một phần khác của ribozyme thể hiện tính biến đổi ngẫu nhiên. Khi đã mã hóa đủ peptide, bất kỳ biến đổi ngẫu nhiên lớn nào trong mã di truyền đều có thể gây chết người; do đó nó trở thành "đóng băng" (frozen).[94]
- Ái lực hóa lập thể (Stereochemical affinity): mã di truyền là kết quả của ái lực cao giữa mỗi amion acid và codon hoặc cụm đối mã của nó; lựa chọn của cụm đối mã ngụ ý rằng những phân tử tiền tRNA khớp với các amino acid tương ứng của chúng bằng ái lực này. Sau đó trong quá trình tiến hóa, sự tương thích này dần bị thay thế bằng sự tương hợp của synthetase aminoacyl-tRNA.[92][95][96]
- Tính tối ưu (Optimality): mã di truyền tiếp tục tiến hóa sau lần tạo đầu tiên, vì thế mã hiện tại tối đa hóa một vài chức năng thích ứng, thường là một số loại giảm thiểu lỗi.[92][93][97]
Các giả thuyết đã xử lý nhiều tình huống khác nhau:[98]
- Những nguyên tắc hóa học chi phối tương tác cụ thể của RNA với amino acid. Những thí nghiệm với aptamer cho thấy rằng một vài amino acid sở hữu ái lực hóa chọn lọc đối với codon của chúng.[99] Các thí nghiệm cho thấy rằng trong số 8 amino acid được thử nghiệm, 6 amino acid thể hiện sự liên kết giữa bộ ba amino acid của RNA.[86][96]
- Mở rộng sinh tổng hợp. Mã di truyền phát triển từ một mã đơn giản hơn trước đây thông qua quá trình "mở rộng sinh tổng hợp". Sự sống nguyên thủy "khám phá" ra những amino acid mới (ví dụ như các sản phẩm phụ của trao đổi chất) và rồi kết hợp một vài trong số này vào bộ máy mã hóa di truyền.[100] Mặc dù nhiều bằng chứng gián tiếp đã được tìm thấy cho thấy rằng trước đây ít loại amino acid được sử dụng hơn,[101] những giả thuyết chính xác và chi tiết về amino acid nào được đưa nhập mã theo thứ tự ra sao đang gây tranh cãi.[102][103] Tuy nhiên, một số nghiên cứu cho rằng Gly, Ala, Asp, Val, Ser, Pro, Glu, Leu, Thr có thể thuộc nhóm amino acid bổ sung đầu, trong khi Cys, Met, Tyr, Trp, His, Phe có thể thuộc nhóm thành nhóm amino acid bổ sung sau.[104][105][106][107]
- Chọn lọc tự nhiên đã dẫn đến phân chia codon của mã di truyền nhằm giảm thiểu tác động của đột biến.[108] Một giả thuyết gần đây[109] đề xuất rằng mã bộ ba bắt nguồn từ những mã sử dụng lâu hơn codon bộ ba (chẳng hạn như codon bộ bốn). Thời gian giải mã bộ ba dài hơn sẽ làm tăng dư thừa codon và có khả năng kháng lỗi cao hơn. Tính năng này có thể cho phép giải mã chính xác khi không có bộ máy dịch mã phức tạp như ribosome, chẳng hạn như trước khi tế bào bắt đầu tạo ra ribosome.
- Kênh thông tin: Lý thuyết thông tin tiếp cận mô hình hóa quá trình dịch mã di truyền thành các amino acid tương ứng như một kênh thông tin dễ gây lỗi.[110] Tiếng ồn vốn có (tức là lỗi) trong kênh đặt ra cho sinh vật một câu hỏi cơ bản: làm thế nào mà một mã di truyền có thể được xây dựng để chống tiếng ồn,[111] mà vẫn dịch thông tin chính xác và hiệu quả? Những mô hình "biến dạng tỷ lệ" này[112] cho rằng mã di truyền bắt nguồn từ tương tác của ba lực tiến hóa xung đột: nhu cầu amino acid đa dạng, [113] khả năng chịu lỗi[108] và phí nguồn sống tối thiểu. Mã xuất hiện tại một đồng hoán khi xây dựng bản đồ codon thành amino acid không hề ngẫu nhiên. Sự xuất hiện của mã mà tô pô chi phối được xác định bởi những lỗi có thể xảy ra và liên quan đến vấn đề tô màu bản đồ.[114]
- Lý thuyết trò chơi: Những mô hình dựa trên trò chơi ra tín hiệu kết hợp các yếu tố của lý thuyết trò chơi, chọn lọc tự nhiên và kênh thông tin. Những mô hình như vậy đã được sử dụng để đề xuất rằng các polypeptide đầu tiên dường như ngắn và không có chức năng enzym. Những mô hình thuyết trò chơi cho rằng việc tổ chức các chuỗi RNA vào tế bào có thể là cần thiết để ngăn chặn sử dụng mã di truyền "lừa bịp", tức là ngăn chặn các loại virus tương đương cổ đại lấn át thế giới RNA.[115]
- Mã kết thúc: Những cụm dịch mã kết thúc cũng là một khía cạnh thú vị đối với vấn đề nguồn gốc của mã di truyền. Dưới dạng ví dụ để giải quyết tiến hóa của mã kết thúc, các nhà khoa học Cho rằng codon kết thúc như vậy để cho chúng có nhiều khả năng kết thúc dịch mã sớm trong trường hợp xảy ra lỗi dịch khung.[116] Ngược lại, một số mô hình phân tử hóa lập thể giải thích nguồn gốc của các mã kết thúc là "không gắn được".[92]
Xem thêm
[sửa | sửa mã nguồn]Chú thích
[sửa | sửa mã nguồn]- ^ Turanov AA, Lobanov AV, Fomenko DE, Morrison HG, Sogin ML, Klobutcher LA, Hatfield DL, Gladyshev VN (tháng 1 năm 2009). “Genetic code supports targeted insertion of two amino acids by one codon”. Science. 323 (5911): 259–61. doi:10.1126/science.1164748. PMC 3088105. PMID 19131629.
- ^ Watson, J. D.; Crick, F. H. (30 tháng 5 năm 1953). “Genetical implications of the structure of deoxyribonucleic acid”. Nature. 171 (4361): 964–967. Bibcode:1953Natur.171..964W. doi:10.1038/171964b0. ISSN 0028-0836. PMID 13063483.
- ^ Stegmann, Ulrich E. (1 tháng 9 năm 2016). “'Genetic Coding' Reconsidered: An Analysis of Actual Usage”. The British Journal for the Philosophy of Science (bằng tiếng Anh). 67 (3): 707–730. doi:10.1093/bjps/axv007. ISSN 0007-0882. PMC 4990703. PMID 27924115.
- ^ Crick, Francis (10 tháng 7 năm 1990). “Chapter 8: The Genetic Code”. What Mad Pursuit: A Personal View of Scientific Discovery. Basic Books. tr. 89–101. ISBN 9780465091386. OCLC 1020240407.[liên kết hỏng]
- ^ Hayes, Brian (1998). “Computing Science: The Invention of the Genetic Code”. American Scientist. 86 (1): 8–14. doi:10.1511/1998.17.3338. ISSN 0003-0996. JSTOR 27856930.
- ^ Strauss, Bernard S (1 tháng 3 năm 2019). “Martynas Yčas: The "Archivist" of the RNA Tie Club”. Genetics. 211 (3): 789–795. doi:10.1534/genetics.118.301754. ISSN 1943-2631. PMC 6404253. PMID 30846543.
- ^ “Francis Crick - Profiles in Science Search Results”. profiles.nlm.nih.gov. Truy cập ngày 21 tháng 7 năm 2022.
- ^ a b Fry, Michael (2022). “Crick's Adaptor Hypothesis and the Discovery of Transfer RNA: Experiment Surpassing Theoretical Prediction”. Philosophy, Theory, and Practice in Biology. 14. doi:10.3998/ptpbio.2628. ISSN 2475-3025.
- ^ Crick, Francis (1955). “On Degenerate Templates and the Adaptor Hypothesis: A Note for the RNA Tie Club”. National Library of Medicine. Truy cập ngày 21 tháng 7 năm 2022.
- ^ Watson, James D. (2007). Avoid Boring People: Lessons from a Life in Science (bằng tiếng Anh). Oxford University Press. tr. 112. ISBN 978-0-19-280273-6. OCLC 47716375.
- ^ Barciszewska, Mirosława Z.; Perrigue, Patrick M.; Barciszewski, Jan (2016). “tRNA--the golden standard in molecular biology”. Molecular BioSystems. 12 (1): 12–17. doi:10.1039/c5mb00557d. PMID 26549858.
- ^ Yanofsky, Charles (9 tháng 3 năm 2007). “Establishing the Triplet Nature of the Genetic Code”. Cell. 128 (5): 815–818. doi:10.1016/j.cell.2007.02.029. PMID 17350564.
- ^ Nirenberg MW, Matthaei JH (tháng 10 năm 1961). “The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides”. Proceedings of the National Academy of Sciences of the United States of America. 47 (10): 1588–602. Bibcode:1961PNAS...47.1588N. doi:10.1073/pnas.47.10.1588. PMC 223178. PMID 14479932.
- ^ Navarro, Sidney (2019). Molecular Biology Gene to Proteins. EDTECH. tr. 264. ISBN 9781839471643.
- ^ Gardner RS, Wahba AJ, Basilio C, Miller RS, Lengyel P, Speyer JF (tháng 12 năm 1962). “Synthetic polynucleotides and the amino acid code. VII”. Proceedings of the National Academy of Sciences of the United States of America. 48 (12): 2087–94. Bibcode:1962PNAS...48.2087G. doi:10.1073/pnas.48.12.2087. PMC 221128. PMID 13946552.
- ^ Wahba AJ, Gardner RS, Basilio C, Miller RS, Speyer JF, Lengyel P (tháng 1 năm 1963). “Synthetic polynucleotides and the amino acid code. VIII”. Proceedings of the National Academy of Sciences of the United States of America. 49 (1): 116–22. Bibcode:1963PNAS...49..116W. doi:10.1073/pnas.49.1.116. PMC 300638. PMID 13998282.
- ^ Navarro, Sidney (2019). Molecular Biology Gene to Proteins. EDTECH. tr. 264. ISBN 9781839471643.
- ^ Taylor, Herbert (2012). Molecular Genetics Pt 2. Elsevier Science. tr. 167. ISBN 9780323146845.
- ^ “The Nobel Prize in Physiology or Medicine 1959” (Thông cáo báo chí). The Royal Swedish Academy of Science. 1959. Truy cập ngày 27 tháng 2 năm 2010.
The Nobel Prize in Physiology or Medicine 1959 was awarded jointly to Severo Ochoa and Arthur Kornberg 'for their discovery of the mechanisms in the biological synthesis of ribonucleic acid and deoxyribonucleic acid'.
- ^ Nirenberg M, Leder P, Bernfield M, Brimacombe R, Trupin J, Rottman F, O'Neal C (tháng 5 năm 1965). “RNA codewords and protein synthesis, VII. On the general nature of the RNA code”. Proceedings of the National Academy of Sciences of the United States of America. 53 (5): 1161–8. Bibcode:1965PNAS...53.1161N. doi:10.1073/pnas.53.5.1161. PMC 301388. PMID 5330357.
- ^ “The Nobel Prize in Physiology or Medicine 1968” (Thông cáo báo chí). The Royal Swedish Academy of Science. 1968. Truy cập ngày 27 tháng 2 năm 2010.
The Nobel Prize in Physiology or Medicine 1968 was awarded jointly to Robert W. Holley, Har Gobind Khorana and Marshall W. Nirenberg 'for their interpretation of the genetic code and its function in protein synthesis'.
- ^ Edgar B (tháng 10 năm 2004). “The genome of bacteriophage T4: an archeological dig”. Genetics. 168 (2): 575–82. doi:10.1093/genetics/168.2.575. PMC 1448817. PMID 15514035.
- ^ Xia, Xuhua (2018). Bioinformatics and the Cell: Modern Computational Approaches in Genomics, Proteomics and Transcriptomics. Springer International Publishing. tr. 30. ISBN 9783319906843.
- ^ Budisa, Nediljko (23 tháng 12 năm 2005). The book at the Wiley Online Library. doi:10.1002/3527607188. ISBN 9783527312436.
- ^ Kubyshkin, V.; Budisa, N. (2017). “Synthetic alienation of microbial organisms by using genetic code engineering: Why and how?”. Biotechnology Journal. 12 (8): 1600097. doi:10.1002/biot.201600097. PMID 28671771.
- ^ Xie J, Schultz PG (tháng 12 năm 2005). “Adding amino acids to the genetic repertoire”. Current Opinion in Chemical Biology. 9 (6): 548–54. doi:10.1016/j.cbpa.2005.10.011. PMID 16260173.
- ^ Wang Q, Parrish AR, Wang L (tháng 3 năm 2009). “Expanding the genetic code for biological studies”. Chemistry & Biology. 16 (3): 323–36. doi:10.1016/j.chembiol.2009.03.001. PMC 2696486. PMID 19318213.
- ^ Simon M (7 tháng 1 năm 2005). Emergent Computation: Emphasizing Bioinformatics. Springer Science & Business Media. tr. 105–106. ISBN 978-0-387-22046-8.
- ^ Hoesl, M. G.; Oehm, S.; Durkin, P.; Darmon, E.; Peil, L.; Aerni, H.-R.; Rappsilber, J.; Rinehart, J.; Leach, D.; Söll, D.; Budisa, N. (2015). “Chemical evolution of a bacterial proteome”. Angewandte Chemie International Edition. 54 (34): 10030–10034. doi:10.1002/anie.201502868. PMC 4782924. PMID 26136259. NIHMSID: NIHMS711205
- ^ “First stable semisynthetic organism created | KurzweilAI”. www.kurzweilai.net. 3 tháng 2 năm 2017. Truy cập ngày 9 tháng 2 năm 2017.
- ^ Zhang Y, Lamb BM, Feldman AW, Zhou AX, Lavergne T, Li L, Romesberg FE (tháng 2 năm 2017). “A semisynthetic organism engineered for the stable expansion of the genetic alphabet”. Proceedings of the National Academy of Sciences of the United States of America. 114 (6): 1317–1322. Bibcode:2017PNAS..114.1317Z. doi:10.1073/pnas.1616443114. PMC 5307467. PMID 28115716.
- ^ Han S, Yang A, Lee S, Lee HW, Park CB, Park HS (tháng 2 năm 2017). “Expanding the genetic code of Mus musculus”. Nature Communications. 8: 14568. Bibcode:2017NatCo...814568H. doi:10.1038/ncomms14568. PMC 5321798. PMID 28220771.
- ^ Zimmer, Carl (15 tháng 5 năm 2019). “Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life? - In a milestone for synthetic biology, colonies of E. coli thrive with DNA constructed from scratch by humans, not nature”. The New York Times. Lưu trữ bản gốc ngày 2 tháng 1 năm 2022. Truy cập ngày 16 tháng 5 năm 2019.
- ^ Fredens, Julius; và đồng nghiệp (15 tháng 5 năm 2019). “Total synthesis of Escherichia coli with a recoded genome”. Nature. 569 (7757): 514–518. Bibcode:2019Natur.569..514F. doi:10.1038/s41586-019-1192-5. PMC 7039709. PMID 31092918.
- ^ Homo sapiens mitochondrion, complete genome. "Revised Cambridge Reference Sequence (rCRS): accession NC_012920", National Center for Biotechnology Information. Retrieved on 27 December 2017.
- ^ a b King RC, Mulligan P, Stansfield W (10 tháng 1 năm 2013). A Dictionary of Genetics. OUP USA. tr. 608. ISBN 978-0-19-976644-4.
- ^ Mark Meneely, Philip; Dawes Hoang, Rachel; Iruka, N. Okeke; Heston, Katherine (2017). Genetics: Genes, Genomes, and Evolution. Đại học báo chí Oxford. tr. 53. ISBN 9780198795360.
- ^ Pierce, Glen; Scott, Lynn (2019). Microbial Physiology Genetics and Ecology. Liên hiệp Anh: EDTECH. tr. 117. ISBN 9781839472732.
- ^ Touriol C, Bornes S, Bonnal S, Audigier S, Prats H, Prats AC, Vagner S (2003). “Generation of protein isoform diversity by alternative initiation of translation at non-AUG codons”. Biology of the Cell. 95 (3–4): 169–78. doi:10.1016/S0248-4900(03)00033-9. PMID 12867081.
- ^ Maloy S (29 tháng 11 năm 2003). “How nonsense mutations got their names”. Microbial Genetics Course. San Diego State University. Truy cập ngày 10 tháng 3 năm 2010.
- ^ Griffiths AJ, Miller JH, Suzuki DT, Lewontin RC, và đồng nghiệp biên tập (2000). “Spontaneous mutations”. An Introduction to Genetic Analysis (ấn bản thứ 7). New York: W. H. Freeman. ISBN 978-0-7167-3520-5.
- ^ Freisinger E, Grollman AP, Miller H, Kisker C (tháng 4 năm 2004). “Lesion (in)tolerance reveals insights into DNA replication fidelity”. The EMBO Journal. 23 (7): 1494–505. doi:10.1038/sj.emboj.7600158. PMC 391067. PMID 15057282.
- ^ Boillée, S; Vande Velde, C; Cleveland, D. W. (2006). “ALS: A disease of motor neurons and their nonneuronal neighbors”. Neuron. 52 (1): 39–59. doi:10.1016/j.neuron.2006.09.018. PMID 17015226.
- ^ Chang JC, Kan YW (tháng 6 năm 1979). “beta 0 thalassemia, a nonsense mutation in man”. Proceedings of the National Academy of Sciences of the United States of America. 76 (6): 2886–9. Bibcode:1979PNAS...76.2886C. doi:10.1073/pnas.76.6.2886. PMC 383714. PMID 88735.
- ^ Boillée S, Vande Velde C, Cleveland DW (tháng 10 năm 2006). “ALS: a disease of motor neurons and their nonneuronal neighbors”. Neuron. 52 (1): 39–59. doi:10.1016/j.neuron.2006.09.018. PMID 17015226.
- ^ Isbrandt D, Hopwood JJ, von Figura K, Peters C (1996). “Two novel frameshift mutations causing premature stop codons in a patient with the severe form of Maroteaux-Lamy syndrome”. Human Mutation. 7 (4): 361–3. doi:10.1002/(SICI)1098-1004(1996)7:4<361::AID-HUMU12>3.0.CO;2-0. PMID 8723688.
- ^ Crow JF (1993). “How much do we know about spontaneous human mutation rates?”. Environmental and Molecular Mutagenesis. 21 (2): 122–9. doi:10.1002/em.2850210205. PMID 8444142.
- ^ Lewis R (2005). Human Genetics: Concepts and Applications (ấn bản thứ 6). Boston, Mass: McGraw Hill. tr. 227–228. ISBN 978-0-07-111156-0.
- ^ Sawyer SA, Parsch J, Zhang Z, Hartl DL (tháng 4 năm 2007). “Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila”. Proceedings of the National Academy of Sciences of the United States of America. 104 (16): 6504–10. Bibcode:2007PNAS..104.6504S. doi:10.1073/pnas.0701572104. PMC 1871816. PMID 17409186.
- ^ Bridges KR (2002). “Malaria and the Red Cell”. Harvard. Bản gốc lưu trữ ngày 27 tháng 11 năm 2011.
- ^ Drake JW, Holland JJ (tháng 11 năm 1999). “Mutation rates among RNA viruses”. Proceedings of the National Academy of Sciences of the United States of America. 96 (24): 13910–3. Bibcode:1999PNAS...9613910D. doi:10.1073/pnas.96.24.13910. PMC 24164. PMID 10570172.
- ^ Holland J, Spindler K, Horodyski F, Grabau E, Nichol S, VandePol S (tháng 3 năm 1982). “Rapid evolution of RNA genomes”. Science. 215 (4540): 1577–85. Bibcode:1982Sci...215.1577H. doi:10.1126/science.7041255. PMID 7041255.
- ^ de Visser JA, Rozen DE (tháng 4 năm 2006). “Clonal interference and the periodic selection of new beneficial mutations in Escherichia coli”. Genetics. 172 (4): 2093–100. doi:10.1534/genetics.105.052373. PMC 1456385. PMID 16489229.
- ^ Watson, James D. (2008). Molecular Biology of the Gene. Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1. :102–117 :521–522
- ^ Michel-Beyerle, Maria Elisabeth (1990). Reaction centers of photosynthetic bacteria: Feldafing-II-Meeting. Springer-Verlag. ISBN 978-3-540-53420-4.
- ^ Füllen G, Youvan DC (1994). "Genetic Algorithms and Recursive Ensemble Mutagenesis in Protein Engineering". Complexity International 1.
- ^ a b Fricke, Markus (2019). “Global importance of RNA secondary structures in protein coding sequences”. Bioinformatics. 35 (4): 579–583. doi:10.1093/bioinformatics/bty678. PMC 7109657. PMID 30101307.
- ^ “Codon Usage Frequency Table(chart)-Genscript”. www.genscript.com. Truy cập ngày 4 tháng 2 năm 2022.
- ^ “Codon usage table”. www.kazusa.or.jp.
- ^ a b Zhang Y, Baranov PV, Atkins JF, Gladyshev VN (tháng 5 năm 2005). “Pyrrolysine and selenocysteine use dissimilar decoding strategies”. The Journal of Biological Chemistry. 280 (21): 20740–51. doi:10.1074/jbc.M501458200. PMID 15788401.
- ^ Krzycki JA (tháng 12 năm 2005). “The direct genetic encoding of pyrrolysine”. Current Opinion in Microbiology. 8 (6): 706–12. doi:10.1016/j.mib.2005.10.009. PMID 16256420.
- ^ Prat L, Heinemann IU, Aerni HR, Rinehart J, O'Donoghue P, Söll D (tháng 12 năm 2012). “Carbon source-dependent expansion of the genetic code in bacteria”. Proceedings of the National Academy of Sciences of the United States of America. 109 (51): 21070–5. Bibcode:2012PNAS..10921070P. doi:10.1073/pnas.1218613110. PMC 3529041. PMID 23185002.
- ^ a b Dutilh BE, Jurgelenaite R, Szklarczyk R, van Hijum SA, Harhangi HR, Schmid M, de Wild B, Françoijs KJ, Stunnenberg HG, Strous M, Jetten MS, Op den Camp HJ, Huynen MA (tháng 7 năm 2011). “FACIL: Fast and Accurate Genetic Code Inference and Logo”. Bioinformatics. 27 (14): 1929–33. doi:10.1093/bioinformatics/btr316. PMC 3129529. PMID 21653513.
- ^ Francis Crick, 1968. "The Origin of the Genetic Code". J. Mol. Biol.
- ^ Barrell BG, Bankier AT, Drouin J (1979). “A different genetic code in human mitochondria”. Nature. 282 (5735): 189–194. Bibcode:1979Natur.282..189B. doi:10.1038/282189a0. PMID 226894. ()
- ^ a b “The Genetic Codes”. National Center for Biotechnology Information (NCBI). 7 tháng 4 năm 2008. Truy cập ngày 10 tháng 3 năm 2010.
- ^ Jukes TH, Osawa S (tháng 12 năm 1990). “The genetic code in mitochondria and chloroplasts”. Experientia. 46 (11–12): 1117–26. doi:10.1007/BF01936921. PMID 2253709.
- ^ Fitzpatrick DA, Logue ME, Stajich JE, Butler G (1 tháng 1 năm 2006). “A fungal phylogeny based on 42 complete genomes derived from supertree and combined gene analysis”. BMC Evolutionary Biology. 6: 99. doi:10.1186/1471-2148-6-99. PMC 1679813. PMID 17121679.
- ^ Santos MA, Tuite MF (tháng 5 năm 1995). “The CUG codon is decoded in vivo as serine and not leucine in Candida albicans”. Nucleic Acids Research. 23 (9): 1481–6. doi:10.1093/nar/23.9.1481. PMC 306886. PMID 7784200.
- ^ Butler G, Rasmussen MD, Lin MF, và đồng nghiệp (tháng 6 năm 2009). “Evolution of pathogenicity and sexual reproduction in eight Candida genomes”. Nature. 459 (7247): 657–62. Bibcode:2009Natur.459..657B. doi:10.1038/nature08064. PMC 2834264. PMID 19465905.
- ^ Taylor DJ, Ballinger MJ, Bowman SM, Bruenn JA (2013). “Virus-host co-evolution under a modified nuclear genetic code”. PeerJ. 1: e50. doi:10.7717/peerj.50. PMC 3628385. PMID 23638388.
- ^ Hofhuis J, Schueren F, Nötzel C, Lingner T, Gärtner J, Jahn O, Thoms S (2016). “The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code”. Open Biol. 6 (11): 160246. doi:10.1098/rsob.160246. PMC 5133446. PMID 27881739.
- ^ Schueren F, Lingner T, George R, Hofhuis J, Gartner J, Thoms S (2014). “Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals”. eLife. 3: e03640. doi:10.7554/eLife.03640. PMC 4359377. PMID 25247702.
- ^ F. Schueren und S. Thoms (2016). “Functional Translational Readthrough: A Systems Biology Perspective”. PLOS Genetics. 12 (8): e1006196. doi:10.1371/journal.pgen.1006196. PMC 4973966. PMID 27490485.
- ^ Kubyshkin V, Acevedo-Rocha CG, Budisa N (tháng 2 năm 2018). “On universal coding events in protein biogenesis”. Bio Systems. 164: 16–25. doi:10.1016/j.biosystems.2017.10.004. PMID 29030023.
- ^ Heaphy SM, Mariotti M, Gladyshev VN, Atkins JF, Baranov PV (tháng 11 năm 2016). “Novel Ciliate Genetic Code Variants Including the Reassignment of All Three Stop Codons to Sense Codons in Condylostoma magnum”. Molecular Biology and Evolution. 33 (11): 2885–2889. doi:10.1093/molbev/msw166. PMC 5062323. PMID 27501944.
- ^ Lobanov AV, Heaphy SM, Turanov AA, Gerashchenko MV, Pucciarelli S, Devaraj RR, và đồng nghiệp (tháng 1 năm 2017). “Position-dependent termination and widespread obligatory frameshifting in Euplotes translation”. Nature Structural & Molecular Biology. 24 (1): 61–68. doi:10.1038/nsmb.3330. PMC 5295771. PMID 27870834.
- ^ Koonin EV, Novozhilov AS (tháng 2 năm 2009). “Origin and Evolution of the Genetic Code: The Universal Enigma”. IUBMB Life. 61 (2): 91–111. doi:10.1002/iub.146. PMC 3293468. PMID 19117371.
- ^ Sengupta S, Higgs PG (tháng 6 năm 2015). “Pathways of Genetic Code Evolution in Ancient and Modern Organisms”. Journal of Molecular Evolution. 80 (5–6): 229–243. Bibcode:2015JMolE..80..229S. doi:10.1007/s00239-015-9686-8. PMID 26054480.
- ^ Xie J, Schultz PG (tháng 8 năm 2006). “A chemical toolkit for proteins--an expanded genetic code”. Nature Reviews Molecular Cell Biology. 7 (10): 775–782. doi:10.1038/nrm2005. PMID 16926858.
- ^ Neumann H, Wang K, Davis L, Garcia-Alai M, Chin JW (tháng 3 năm 2010). “Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome”. Nature. 18 (464): 441–444. doi:10.1038/nrm2005. PMID 16926858.
- ^ Liu CC, Schultz PG (2010). “Adding new chemistries to the genetic code”. Annual Review of Biochemistry. 79: 413–444. doi:10.1146/annurev.biochem.052308.105824. PMID 20307192.
- ^ Chin JW (tháng 2 năm 2014). “Expanding and reprogramming the genetic code of cells and animals”. Annual Review of Biochemistry. 83: 379–408. doi:10.1146/annurev-biochem-060713-035737. PMID 24555827.
- ^ Shulgina, Y; Eddy, SR (9 tháng 11 năm 2021). “A computational screen for alternative genetic codes in over 250,000 genomes”. eLife. 10. doi:10.7554/eLife.71402. PMC 8629427. PMID 34751130.
- ^ Ribas de Pouplana L, Turner RJ, Steer BA, Schimmel P (tháng 9 năm 1998). “Genetic code origins: tRNAs older than their synthetases?”. Proceedings of the National Academy of Sciences of the United States of America. 95 (19): 11295–300. Bibcode:1998PNAS...9511295D. doi:10.1073/pnas.95.19.11295. PMC 21636. PMID 9736730.
- ^ a b Yarus, Michael (2010). Life from an RNA World: The Ancestor Within. Harvard University Press. ISBN 978-0-674-05075-4.
- ^ “Mathematica function for # possible arrangements of items in bins? – Online Technical Discussion Groups—Wolfram Community”. community.wolfram.com (bằng tiếng Anh). Truy cập ngày 3 tháng 2 năm 2017.
- ^ a b Freeland SJ, Hurst LD (tháng 9 năm 1998). “The genetic code is one in a million”. Journal of Molecular Evolution. 47 (3): 238–48. Bibcode:1998JMolE..47..238F. doi:10.1007/PL00006381. PMID 9732450.
- ^ Taylor FJ, Coates D (1989). “The code within the codons”. Bio Systems. 22 (3): 177–87. doi:10.1016/0303-2647(89)90059-2. PMID 2650752.
- ^ Di Giulio M (tháng 10 năm 1989). “The extension reached by the minimization of the polarity distances during the evolution of the genetic code”. Journal of Molecular Evolution. 29 (4): 288–93. Bibcode:1989JMolE..29..288D. doi:10.1007/BF02103616. PMID 2514270.
- ^ Wong JT (tháng 2 năm 1980). “Role of minimization of chemical distances between amino acids in the evolution of the genetic code”. Proceedings of the National Academy of Sciences of the United States of America. 77 (2): 1083–6. Bibcode:1980PNAS...77.1083W. doi:10.1073/pnas.77.2.1083. PMC 348428. PMID 6928661.
- ^ a b c d Erives A (tháng 8 năm 2011). “A model of proto-anti-codon RNA enzymes requiring L-amino acid homochirality”. Journal of Molecular Evolution. 73 (1–2): 10–22. Bibcode:2011JMolE..73...10E. doi:10.1007/s00239-011-9453-4. PMC 3223571. PMID 21779963.
- ^ a b Freeland SJ, Knight RD, Landweber LF, Hurst LD (tháng 4 năm 2000). “Early fixation of an optimal genetic code”. Molecular Biology and Evolution. 17 (4): 511–18. doi:10.1093/oxfordjournals.molbev.a026331. PMID 10742043.
- ^ Crick FH (tháng 12 năm 1968). “The origin of the genetic code”. Journal of Molecular Evolution. 38 (3): 367–79. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ Hopfield JJ (1978). “Origin of the genetic code: a testable hypothesis based on tRNA structure, sequence, and kinetic proofreading”. PNAS. 75 (9): 4334–4338. Bibcode:1978PNAS...75.4334H. doi:10.1073/pnas.75.9.4334. PMC 336109. PMID 279919.
- ^ a b Yarus M, Widmann JJ, Knight R (tháng 11 năm 2009). “RNA-amino acid binding: a stereochemical era for the genetic code”. Journal of Molecular Evolution. 69 (5): 406–29. Bibcode:2009JMolE..69..406Y. doi:10.1007/s00239-009-9270-1. PMID 19795157.
- ^ Brown, Sean M.; Voráček, Václav; Freeland, Stephen (5 tháng 4 năm 2023). “What Would an Alien Amino Acid Alphabet Look Like and Why?”. Astrobiology. doi:10.1089/ast.2022.0107.
- ^ Knight RD, Freeland SJ, Landweber LF (tháng 6 năm 1999). “Selection, history and chemistry: the three faces of the genetic code”. Trends in Biochemical Sciences. 24 (6): 241–7. doi:10.1016/S0968-0004(99)01392-4. PMID 10366854.
- ^ Knight RD, Landweber LF (tháng 9 năm 1998). “Rhyme or reason: RNA-arginine interactions and the genetic code”. Chemistry & Biology. 5 (9): R215–20. doi:10.1016/S1074-5521(98)90001-1. PMID 9751648.
- ^ Sengupta S, Higgs PG (2015). “Pathways of genetic code evolution in ancient and modern organisms”. Journal of Molecular Evolution. 80 (5–6): 229–243. Bibcode:2015JMolE..80..229S. doi:10.1007/s00239-015-9686-8. PMID 26054480.
- ^ Brooks DJ, Fresco JR, Lesk AM, Singh M (tháng 10 năm 2002). “Evolution of amino acid frequencies in proteins over deep time: inferred order of introduction of amino acids into the genetic code”. Molecular Biology and Evolution. 19 (10): 1645–55. doi:10.1093/oxfordjournals.molbev.a003988. PMID 12270892.
- ^ Amirnovin R (tháng 5 năm 1997). “An analysis of the metabolic theory of the origin of the genetic code”. Journal of Molecular Evolution. 44 (5): 473–6. Bibcode:1997JMolE..44..473A. doi:10.1007/PL00006170. PMID 9115171.
- ^ Ronneberg TA, Landweber LF, Freeland SJ (tháng 12 năm 2000). “Testing a biosynthetic theory of the genetic code: fact or artifact?”. Proceedings of the National Academy of Sciences of the United States of America. 97 (25): 13690–5. Bibcode:2000PNAS...9713690R. doi:10.1073/pnas.250403097. PMC 17637. PMID 11087835.
- ^ Trifonov, Edward N. (tháng 9 năm 2009). “The origin of the genetic code and of the earliest oligopeptides”. Research in Microbiology (bằng tiếng Anh). 160 (7): 481–486. doi:10.1016/j.resmic.2009.05.004. PMID 19524038.
- ^ Higgs, Paul G.; Pudritz, Ralph E. (tháng 6 năm 2009). “A Thermodynamic Basis for Prebiotic Amino Acid Synthesis and the Nature of the First Genetic Code”. Astrobiology (bằng tiếng Anh). 9 (5): 483–490. arXiv:0904.0402. Bibcode:2009AsBio...9..483H. doi:10.1089/ast.2008.0280. ISSN 1531-1074. PMID 19566427.
- ^ Chaliotis, Anargyros; Vlastaridis, Panayotis; Mossialos, Dimitris; Ibba, Michael; Becker, Hubert D.; Stathopoulos, Constantinos; Amoutzias, Grigorios D. (17 tháng 2 năm 2017). “The complex evolutionary history of aminoacyl-tRNA synthetases”. Nucleic Acids Research (bằng tiếng Anh). 45 (3): 1059–1068. doi:10.1093/nar/gkw1182. ISSN 0305-1048. PMC 5388404. PMID 28180287.
- ^ Ntountoumi, Chrysa; Vlastaridis, Panayotis; Mossialos, Dimitris; Stathopoulos, Constantinos; Iliopoulos, Ioannis; Promponas, Vasilios; Oliver, Stephen G; Amoutzias, Grigoris D (4 tháng 11 năm 2019). “Low complexity regions in the proteins of prokaryotes perform important functional roles and are highly conserved”. Nucleic Acids Research (bằng tiếng Anh). 47 (19): 9998–10009. doi:10.1093/nar/gkz730. ISSN 0305-1048. PMC 6821194. PMID 31504783.
- ^ a b Freeland SJ, Wu T, Keulmann N (tháng 10 năm 2003). “The case for an error minimizing standard genetic code”. Origins of Life and Evolution of the Biosphere. 33 (4–5): 457–77. Bibcode:2003OLEB...33..457F. doi:10.1023/A:1025771327614. PMID 14604186.
- ^ Baranov PV, Venin M, Provan G (2009). Gemmell NJ (biên tập). “Codon size reduction as the origin of the triplet genetic code”. PLOS ONE. 4 (5): e5708. Bibcode:2009PLoSO...4.5708B. doi:10.1371/journal.pone.0005708. PMC 2682656. PMID 19479032.
- ^ Tlusty T (tháng 11 năm 2007). “A model for the emergence of the genetic code as a transition in a noisy information channel”. Journal of Theoretical Biology. 249 (2): 331–42. arXiv:1007.4122. Bibcode:2007JThBi.249..331T. doi:10.1016/j.jtbi.2007.07.029. PMID 17826800.
- ^ Sonneborn TM (1965). Bryson V, Vogel H (biên tập). Evolving genes and proteins. New York: Academic Press. tr. 377–397.
- ^ Tlusty T (tháng 2 năm 2008). “Rate-distortion scenario for the emergence and evolution of noisy molecular codes”. Physical Review Letters. 100 (4): 048101. arXiv:1007.4149. Bibcode:2008PhRvL.100d8101T. doi:10.1103/PhysRevLett.100.048101. PMID 18352335.
- ^ Sella G, Ardell DH (tháng 9 năm 2006). “The coevolution of genes and genetic codes: Crick's frozen accident revisited”. Journal of Molecular Evolution. 63 (3): 297–313. Bibcode:2006JMolE..63..297S. doi:10.1007/s00239-004-0176-7. PMID 16838217.
- ^ Tlusty T (tháng 9 năm 2010). “A colorful origin for the genetic code: information theory, statistical mechanics and the emergence of molecular codes”. Physics of Life Reviews. 7 (3): 362–76. arXiv:1007.3906. Bibcode:2010PhLRv...7..362T. doi:10.1016/j.plrev.2010.06.002. PMID 20558115.
- ^ Jee J, Sundstrom A, Massey SE, Mishra B (tháng 11 năm 2013). “What can information-asymmetric games tell us about the context of Crick's 'frozen accident'?”. Journal of the Royal Society, Interface. 10 (88): 20130614. doi:10.1098/rsif.2013.0614. PMC 3785830. PMID 23985735.
- ^ Itzkovitz S, Alon U (2007). “The genetic code is nearly optimal for allowing additional information within protein-coding sequences”. Genome Research. 17 (4): 405–412. doi:10.1101/gr.5987307. PMC 1832087. PMID 17293451.
Đọc thêm
[sửa | sửa mã nguồn]- Griffiths AJ, Miller JH, Suzuki DT, Lewontin RC, Gilbert WM (1999). An Introduction to genetic analysis (ấn bản thứ 7). San Francisco: W.H. Freeman. ISBN 978-0-7167-3771-1.
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular biology of the cell (ấn bản thứ 4). New York: Garland Science. ISBN 978-0-8153-3218-3.
- Lodish HF, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell JE (2000). Molecular cell biology (ấn bản thứ 4). San Francisco: W.H. Freeman. ISBN 9780716737063.
- Caskey CT, Leder P (tháng 4 năm 2014). “The RNA code: nature's Rosetta Stone”. Proceedings of the National Academy of Sciences of the United States of America. 111 (16): 5758–9. Bibcode:2014PNAS..111.5758C. doi:10.1073/pnas.1404819111. PMC 4000803. PMID 24756939.
Liên kết ngoài
[sửa | sửa mã nguồn]- Genetic code tại Encyclopædia Britannica (tiếng Anh)
- Mã di truyền: Bảng mã di truyền
- Cơ sở dữ liệu sử dụng codon — Bảng tần suất codon ở nhiều sinh vật
- Lịch sử giải mã di truyền
 GIẢM
26%
GIẢM
26%
 GIẢM
17%
GIẢM
17%
 GIẢM
3%
GIẢM
3%
 GIẢM
16%
GIẢM
16%
 GIẢM
25%
GIẢM
25%


![[Review sách] Ba người thầy vĩ đại - Ba câu hỏi giúp bạn tìm ra giá trị đích thực của cuộc sống](https://down-tx-vn.img.susercontent.com/sg-11134201-22110-v6fza6ytugkvef.webp)



